Resumos
Este estudo teve como objetivo determinar o cariótipo de quatro acessos de Capsicum chinense procedentes do Rio Janeiro, Bahia, Pará e Maranhão. Em todos os acessos, foram observados 2n=2x=24 cromossomos. Polimorfismo cromossômico foi observado no acesso procedente do Rio de Janeiro, o qual apresentou 11 pares de cromossomos metacêntricos e um par submetacêntrico, diferindo dos outros acessos, os quais apresentaram 11 pares de cromossomos metacêntricos e um par de cromossomos acrocêntrico. Todos os acessos apresentaram, em média, valores semelhantes em relação ao comprimento cromossômico. Os dados cariológicos foram submetidos a análises univariada e multivariada, com índices de dissimilaridade obtidos pela distância euclidiana média padronizada e o agrupamento pelo método de Ward, com o objetivo de estimar a divergência genética entre os acessos. A estatística multivariada usando dados citogenéticos foi efetiva para distinguir os acessos de C. chinense, considerando que foi possível alocar o acesso do Rio de Janeiro isolado dos outros acessos. Assim, o presente estudo mostrou a importância de análise multivariada em estudos de caracterização citogenética envolvendo acessos que apresentam variabilidade cromossômica.
caracterização citogenética; variabilidade cromossômica; análise multivariada; cariótipo
This study had the objective to determine the karyotype of four accessions of Capsicum chinense from Rio Janeiro, Bahia, Pará e Maranhão. In all accessions were observed 2n=2x=24 chromosomes. Chromosome polymorphism was observed in the accession from Rio de Janeiro, which presented 11 pairs of metacentric and 1 submetacentric pair of chromosomes, differing from the other accessions, which presented 11 pairs of metacentric and 1 acrocentric pair chromosomes. All the accessions presented the same value, on average, of chromosome length. The karyologic data was submitted to univariate and multivariate analysis with dissimilarity indexes obtained by standardized average of Euclidean distance and grouping clustering by the Ward's method, with the objective of estimating the genetic divergence among accessions. The multivariate statistic using cytogenetic data was effective to distinguish C. chinense accessions, considering that it was possible to allocate the Rio de Janeiro accession isolated from the others accessions. So, the present study showed the importance of the multivariate statistic in cytogenetic characterization study involving accessions that present chromosome variability.
cytogenetic characterization; chromossomic variability; multivariate analysis; karyotype
ARTIGOS CIENTÍFICOS
BIOLOGIA
Polimorfismo cromossômico em Capsicum chinense Jacq.
Chromosome polymorphism in Capsicum chinense Jacq.
Sérgio Alessandro Machado SouzaI; Kellen Coutinho MartinsI; Telma Nair Santana PereiraI,1 1 Autor para correspondência.
ILaboratório de Melhoramento Genético Vegetal (LMGV), Universidade Estadual do Norte Fluminense Darcy Ribeiro (UENF), 28013-602, Campos dos Goytacazes, RJ, Brasil. E-mail: telmasp@uenf.br
RESUMO
Este estudo teve como objetivo determinar o cariótipo de quatro acessos de Capsicum chinense procedentes do Rio Janeiro, Bahia, Pará e Maranhão. Em todos os acessos, foram observados 2n=2x=24 cromossomos. Polimorfismo cromossômico foi observado no acesso procedente do Rio de Janeiro, o qual apresentou 11 pares de cromossomos metacêntricos e um par submetacêntrico, diferindo dos outros acessos, os quais apresentaram 11 pares de cromossomos metacêntricos e um par de cromossomos acrocêntrico. Todos os acessos apresentaram, em média, valores semelhantes em relação ao comprimento cromossômico. Os dados cariológicos foram submetidos a análises univariada e multivariada, com índices de dissimilaridade obtidos pela distância euclidiana média padronizada e o agrupamento pelo método de Ward, com o objetivo de estimar a divergência genética entre os acessos. A estatística multivariada usando dados citogenéticos foi efetiva para distinguir os acessos de C. chinense, considerando que foi possível alocar o acesso do Rio de Janeiro isolado dos outros acessos. Assim, o presente estudo mostrou a importância de análise multivariada em estudos de caracterização citogenética envolvendo acessos que apresentam variabilidade cromossômica.
Palavras-chave: caracterização citogenética, variabilidade cromossômica, análise multivariada, cariótipo.
ABSTRACT
This study had the objective to determine the karyotype of four accessions of Capsicum chinense from Rio Janeiro, Bahia, Pará e Maranhão. In all accessions were observed 2n=2x=24 chromosomes. Chromosome polymorphism was observed in the accession from Rio de Janeiro, which presented 11 pairs of metacentric and 1 submetacentric pair of chromosomes, differing from the other accessions, which presented 11 pairs of metacentric and 1 acrocentric pair chromosomes. All the accessions presented the same value, on average, of chromosome length. The karyologic data was submitted to univariate and multivariate analysis with dissimilarity indexes obtained by standardized average of Euclidean distance and grouping clustering by the Ward's method, with the objective of estimating the genetic divergence among accessions. The multivariate statistic using cytogenetic data was effective to distinguish C. chinense accessions, considering that it was possible to allocate the Rio de Janeiro accession isolated from the others accessions. So, the present study showed the importance of the multivariate statistic in cytogenetic characterization study involving accessions that present chromosome variability.
Key words: cytogenetic characterization, chromossomic variability, multivariate analysis, karyotype.
INTRODUÇÃO
O gênero Capsicum é composto por 31 espécies, sendo cinco domesticadas (C. annuum, C. baccatum, C. chinense, C. frutescens e C. pubescens), e as demais classificadas como semidomesticadas e silvestres. C. chinense, como todas as demais espécies cultivadas de pimenta do gênero Capsicum, teve sua origem na América e, dentre as espécies domesticadas, é a mais difundida na América tropical, onde apresenta uma grande diversidade biológica (PICKERSGILL, 2007).
A espécie C. chinense destaca-se pela sua ampla adaptação às condições de clima equatorial e tropical e por possuir grande variabilidade genética, evidenciada principalmente nos frutos que podem ter diferentes formatos, colorações, tamanhos e níveis de pungência, sendo uma das espécies de pimenta mais produzidas e consumidas no Brasil (LANNES et al. 2007). Devido à grande variabilidade encontrada na espécie, estudos de caracterização molecular e citogenética são de fundamental importância, pois podem evidenciar informações significativas tanto para o melhoramento genético quanto para estudos citotaxonômicos (PICKERSGILL, 2007).
A caracterização citogenética de espécies tropicais amplia as perspectivas de conservação da diversidade vegetal de espécies, com potencial em programas de melhoramento vegetal (PEREIRA et al., 2006). Características desejáveis, tais como, firmeza, uniformidade da maturação, sólidos solúveis e vitamina C (LANNES et al., 2007), presentes em C. chinense, despertam interesse de incluir essa espécie em programas de melhoramento da espécie mais cultivada do gênero Capsicum, o pimentão.
Estudos citogenéticos em relação ao número de cromossomos (MOSCONE et al., 2007) e ao comportamento meiótico (MARTINS et al., 2010), envolvendo espécies cultivadas do gênero Capsicum, são de suma importância, pois o número de cromossomos de uma espécie, em geral, é constante, e pode ser uma característica útil na taxonomia. Contudo, o surgimento de polimorfismo a nível cromossômico, em indivíduos de uma população, pode alterar o padrão cariotípico desses espécimes e assim originar raças cromossômicas distintas (TEODORO-PARDO, 2007).
As espécies cultivadas do gênero Capsicum e algumas espécies silvestres apresentam 2n=2x=24 cromossomos (PICKERSGILL, 1997), entretanto, é evidenciado em algumas espécies silvestres, como, por exemplo, C. buforum, C. capylopodium e C. cornutum, 2n=2x=26 cromossomos (POZZOBON & WITTMANN, 2006). Diferenças na morfologia ou no número de cromossomos podem ocorrer em populações da mesma espécie ou em táxons interespecíficos e, segundo MOSCONE et al. (2007), são consideradas comuns dentro do gênero Capsicum.
Devido à grande importância de C. chinense e ampla variabilidade encontrada nessa espécie, este estudo teve como objetivos, caracterizar e analisar o cariótipo de quatro acessos de C. chinense, provenientes de diferentes regiões do Brasil.
MATERIAL E MÉTODOS
Sementes dos acessos UENF1749, UENF1753, UENF1759 e UENF1708 de C. chinense procedentes do RJ, BA, PA e MA, respectivamente, foram colocadas para germinar em recipientes de 500mL contendo substrato Plantmax®, mantidos em casa de vegetação e em condições recomendadas para a cultura. Esses acessos foram selecionados por apresentarem características de interesse, como teor de vitamina C e sólidos solúveis totais, segundo SOUZA (2008). Após 15 dias, meristemas radiculares dos diferentes acessos foram coletados e pré-tratados individualmente com paradiclorobenzeno (PDB) por 4 horas e 30 minutos à 10°C. Após o pré-tratamento, as pontas de raízes foram lavadas em água destilada, fixadas em solução de Carnoy - etanol absoluto e ácido acético glacial (3:1) e conservadas a -4°C, para utilização posterior. No momento do preparo da lâmina, os meristemas fixados foram lavados em água destilada por cinco minutos e, em seguida, foram submetidos à hidrólise em HCL 1N pré-aquecido a 60°C, durante 10 minutos; após a hidrólise, as pontas foram lavadas em água destilada. Lâminas temporárias foram preparadas colocando-se uma ponta de raiz em uma lâmina e macerando sob uma lamínula. Posteriormente, a lamínula foi retirada com o auxílio do nitrogênio líquido. Em seguida, foi depositada, sobre o macerado, uma gota de solução de Carnoy e a lâmina foi colocada para secar em temperatura ambiente. Finalmente, as lâminas foram coradas com solução de Giemsa 2% (água destilada), por 15 minutos, e posteriormente lavadas e secas ao ar. As lâminas foram observadas em microscópio Olympus BX60 e as imagens capturadas utilizando-se o programa Image-Pro Plus versão 5.1.
Cinco placas metafásicas/acesso, as quais corresponderam a repetições, foram utilizadas para a mensuração dos cromossomos, realizada com o auxílio do Programa MicroMeasure 3.3 (REEVES & TEAR, 2000). Foram mensurados: comprimento total do cromossomo, comprimento do braço longo e do braço curto. Com base nesses dados, foi estimada a razão entre braços (r = braço longo/braço curto), o comprimento do lote haplóide (CLH = somatória dos comprimentos absolutos dos cromossomos metafásicos) e o índice centromérico (ic) que é obtido pela razão entre o comprimento do braço curto do lote haplóide em relação ao comprimento absoluto do cromossomo.
Os cromossomos foram classificados em metacêntrico, submetacêntrico, acrocêntrico e telocêntrico, com base na razão entre braços (r) e no índice centromérico (ic), de acordo com GUERRA (1986), que classifica os cromossomos em metacêntrico (r=1,00-1,49 e ic=50,0-40,1); submetacêntrico (r=1,50-2,99 e ic 40,0-25,1); acrocêntrico (r=3,00-∞ e ic=25,0-0,01); e telocêntrico (r=∞ e ic=0).
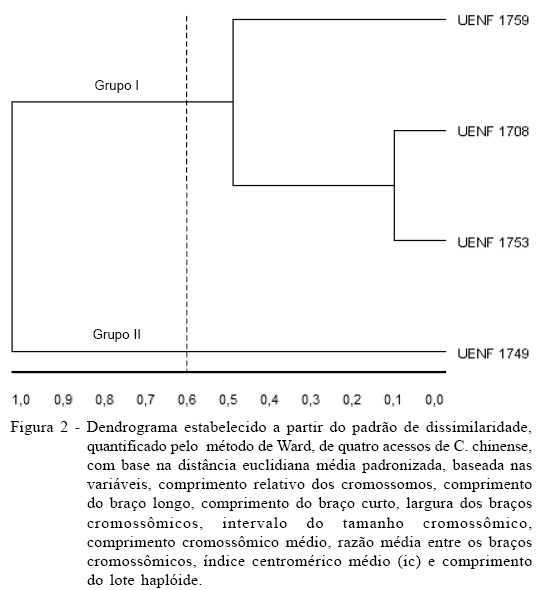
Os dados correspondentes às variáveis intervalo do tamanho cromossômico (ITC), comprimento cromossômico médio (CCM), razão média entre os braços cromossômicos (r), índice centromérico médio (ic) e comprimento do lote haplóide (CLH) foram analisados por estatística descritiva, utilizando-se medidas de tendência central (média) e de variabilidade de dados (desvio-padrão e coeficiente de variação). Foram realizadas análises estatísticas multivariadas, por meio de técnicas de análise de agrupamento. Como medida de dissimilaridade, foi utilizada a distância euclidiana média padronizada e como método hierárquico aglomerativo, o método de WARD (1963). Com base nos cálculos, foi estabelecido o dendrograma, o qual permite verificar o grau de similaridade entre os acessos e grupos similares, utilizando-se o programa GENES (CRUZ, 2006).
RESULTADOS E DISCUSSÃO
Todos os acessos apresentaram o mesmo número cromossômico, 2n=2x=24 cromossomos (Tabela 1; Figura 1), confirmando os dados reportados por MOSCONE et al. (1996), número esse similar aos das demais espécies domesticadas do gênero Capsicum (MOSCONE et al., 2007).
Polimorfismo cromossômico foi evidenciado no acesso UENF 1749, procedente do estado do Rio de Janeiro, o qual apresentou a fórmula cariotípica 11m+1sm diferindo dos demais, que apresentaram 11m+1a e, apesar de apresentarem fórmula cariotípica distinta, o comprimento cromossômico médio dos acessos apresentou valores semelhantes (Tabela 1). Diferenças na morfologia ou no número de cromossomos podem ocorrer em populações da mesma espécie ou em táxons interespecíficos, e são denominados de citótipos ou raças cromossômicas, de acordo com MOSCONE et al. (2007). Na concepção dos mesmos autores, o gênero Capsicum possui diferentes citótipos e eles diferem principalmente na fórmula cariotípica e no comprimento dos cromossomos; diferenças como estas ocorrem devido a variações genéticas entre populações, promovendo, dessa forma, o surgimento de variabilidade genética.
Os dados citogenéticos do presente estudo estão de acordo com GUERRA (2001), que relata o gênero Capsicum como um grupo heterogêneo, do ponto de vista cariológico, apresentando variabilidade no número de cromossomos, localização do centrômero e posição de satélites. Esse polimorfismo, ao nível de morfologia cromossômica nos acessos analisados, evidencia a importância dos estudos cariomorfológicos das espécies do gênero Capsicum. Segundo POZZOBON et al. (2006), a caracterização citogenética é de fundamental importância nos estudos evolutivos do gênero Capsicum. Os mesmos autores ressaltam a existência de duas linhas evolutivas distintas na diversificação do grupo, evidenciando que há uma clara separação entre as espécies silvestres, com o número básico x=13 e as cultivadas apresentando x=12. Essa variabilidade no número básico de cromossomos corrobora a ampla diversidade do gênero Capsicum.
O tamanho médio dos cromossomos variou de 2,59 a 4,12µm (Tabela 1). Esses resultados estão de acordo com os dados de MOSCONE (1996) e GUERRA (2001), que, ao analisarem o cariótipo de acessos de C. chinense provenientes de diferentes regiões geográficas, concluíram que esses valores correspondem a cromossomos que variam de tamanho médio a grande. A variação do tamanho médio dos cromossomos das espécies de Capsicum pode ser atribuída às diferenças significativas no grau de espiralização ou na condensação da cromatina.
A análise do comprimento do lote haplóide (CLH), o qual corresponde ao tamanho médio do genoma, variou de 41,40 a 42,09µm (Tabela 1). Alguns autores consideram esses valores como sendo um genoma pequeno, mesmo C. chinense possuindo cromossomos que variam de tamanho médio a grande (MOSCONE et al., 2003).
A análise de agrupamento com base nos dados cariológicos permitiu a elaboração de um dendrograma hierárquico (Figura 2), no qual se observou a formação de grupos de acessos com algum grau de similaridade e a dissimilaridade entre outros. Foram incluídos, nessa análise multivariada, os dados referentes ao comprimento relativo dos cromossomos, comprimento do braço longo, comprimento do braço curto e largura dos braços cromossômicos, além das variáveis incluídas na tabela 1.
O método hierárquico de Ward possibilitou a distribuição dos acessos dentro dos grupos ao formar o dendrograma. Em um ponto de corte de 60%, o qual correspondeu ao valor médio da dissimilaridade, evidenciou-se a formação de dois grupos: o grupo I com os acessos UENF 1753, UENF 1759 e UENF 1708 e o grupo II apenas com o acesso UENF 1749. Variações cariotípicas intra-específicas são geradas em consequência de rearranjos estruturais e podem significar respostas desses genomas aos diferentes ambientes nos quais são encontrados, como ocorre na espécie C. annuum (TEODORO-PARDO et al., 2007). Essas variações, por sua vez, contribuíram para alocar o acesso procedente do Estado do Rio de Janeiro em um único grupo distinto dos demais.
A identificação das variáveis mais importantes, na formação dos grupos, foi realizada de acordo com o método SINGH (1981), que permite verificar quais características analisadas contribuíram de forma mais eficiente na divergência genética. Verificou-se que o índice centromérico foi, entre as variáveis analisadas, aquele que apresentou maior importância relativa, representando 81,27% da variação entre os acessos analisados (Tabela 2). Segundo TEODORO-PARDO et al. (2007), a presença de modificações estruturais encontradas, como a posição do centrômero, sugere que a morfologia cromossômica pode ter contribuído para a diversificação do gênero Capsicum.
Poucos estudos têm sido desenvolvidos com relação ao do emprego da estatística multivariada em estudos de caracterização citogenética envolvendo acessos de diferentes populações; na maioria dos casos, essas pesquisas demonstram apenas os dados obtidos, sem submetê-los a análises estatísticas mais acuradas que, por sua vez, apresentariam resultados mais satisfatórios. De acordo com ARAUJO et al. (2008), a divergência entre acessos, avaliada por estatística multivariada, pode proporcionar uma descrição sintética da afinidade genética entre acessos e populações de uma determinada espécie em estudo, promovendo uma distinção entre os grupos analisados.
CONCLUSÃO
As diferenças encontradas em populações divergentes de C. chinense evidenciam a presença de citótipos entre os acessos analisados, sendo que os dados cariológicos apresentados podem auxiliar na identificação de populações, bem como fornecer suporte para futuras pesquisas na área de melhoramento genético de Capsicum. O emprego da estatística multivariada, a partir de dados cariomorfológicos, demonstrou ser uma ferramenta eficiente na distinção de acessos de C. chinense provenientes de diferentes localidades. Concomitantemente, evidenciou-se a importância de empregar esse tipo de análise em pesquisas de caracterização citogenética em que há variabilidade cromossômica.
Recebido para publicação 11.05.11
Aprovado em 26.07.11
Devolvido pelo autor 25.08.11
CR-5333
- ARAÚJO, F.P., et al. Divergência genética entre acessos de Passiflora cincinata Mast. com base em descritores morfoagronômicos. Revista Brasileira de Fruticultura, v.30, p.723-730, 2008. Disponível em: <http://www.scielo.br/pdf/rbf/v30n3/27.pdf>. Acesso em: 25 nov. 2010. doi: 10.1590/S0100-29452008000300027.
- CRUZ, C.D. Programa GENES: análise multivariada e simulação Viçosa: UFV, 2006. 175p.
- GUERRA, M.S. Reviewing the chromosome nomenclature of Levan et al. Revista Brasileira de Genética, v.9, p.741-743, 1986. Disponível em: <http://web2.sbg.org.br/gmb/edicoesanteriores/v09n4/pdf/a16v09n4.pdf>. Acesso em: 10 jan. 2008. doi: 10.1590/S0100-84551997000400001.
- GUERRA, N.A. Estudios cromossómicos de cuatro selecciones de Capsicum chinense Jacq. Revista Científica UDO Agrícola, v.1, p.34-41, 2001. Disponível em: <http://www.bioline.org.br/pdf?cg01006>. Acesso em: 25 nov. 2009.
- LANNES, S.D. et al. Growth and quality of Brazilian accessions of Capsicum chinense fruits. Scientia Horticulturae, v.112, p.266-270, 2007. Disponível em: <http:www.elsevier. com/locate/scihorti>. Acesso em: 02 jan. 2008. doi:10.1016/j.scienta.2006.12.029.
- MARTINS, K.C. et al. Meiose e viabilidade polínica de acessos de Capsicum annuum e Capsicum baccatum Ciência Rural, v.40, p.1746-1751, 2010. Disponível em: <http://www.scielo.br/pdf/cr/v40n8/a686cr2712.pdf>. Acesso em: 30 nov. 2010. doi: 10.1590/S0103-84782010000800012.
- MOSCONE, E.A. et al. Fluorescent chromosome banding in the cultivated species of Capsicum (Solanaceae). Plant System Evolution, v.20, p.37-63, 1996. Disponivel em: https://springerlink3.metapress.com/ Acesso em 30 ago. 2009. doi: 10.1007/BF00985817.
- MOSCONE, E.A. et al. Analysis of Nuclear DNA Content in Capsicum (Solanaceae) by flow cytometry and feulgen densitometry. Annals of Botany, n.92, p.21-29. 2003. Disponível em: <http://aob.oxfordjournals.org/content/92/1/21.full.pdf+html>. Acesso em: 06 ago. 2010. doi: 10.1093/aob/mcg105.
- MOSCONE, E.A. et al. The evolution of chili peppers (Capsicum - Solanaceae): a cytogenetic perspective. Acta Horticulturae, v.745, p.137-169, 2007. Disponível em: <http://www.wildchilli.eu/index.php/wild-chilli-species/the-evolution-of-chilli-peppers-new>. Acesso em: 5 mar. 2007.
- PEREIRA, L.P. et al. Número de cromossomos em populações de Achyrocline satureioides Lam. Ciência Rural, v.36, p.678-681, 2006. Disponível em: <http://www.scielo.br/pdf/ cr/v36n2/a52v36n2.pdf>. Acesso em: 2 abr. 2008. doi: 10.1590/S0103-84782006000200052.
- PICKERSGILL, B. Cytogenetics and evolution of Capsicum L. In: TSUCHIYA, T.; GUPTA, P.K. Chromosome engineering in plants: genetics, breeding, evolution Amsterdam: Elsevier, 1991. p.139-160. Disponível em: <http://www.cababstractsplus.org/abstracts/Abstract.aspx?AcNo=19931636082>. Acesso em: 20 fev. 2008.
- PICKERSGILL, B. Genetic resources and breeding of Capsicum spp. Euphytica, v.96, p.129-133, 1997. Disponível em: <http://www.springerlink.com/content/w1h5437x32531771/fulltext.pdf>. Acesso em: 12 mar. 2009. doi: 10.1023/A:1002913228101.
- PICKERSGILL, B. Domestication of plants in the Americas: insights from Mendelian and molecular genetics. Annals of Botany, v.100, p.925-940, 2007. Disponível em: <http://aob.oxfordjournals.org/content/100/5/925.full.pdf>. Acesso em: 28 fev. 2008. doi:10.1093/aob/mcm193.
- POZZOBON, M.T.; WITTMANN, M.T. A meiotic study of the wild and semi-domesticated Brazilian species of genus Capsicum L. (Solanaceae). Cytologia, v.71, n.3, p.275-287, 2006. Disponível em: <http://www.jstage.jst.go.jp/article/cytologia/71/3/71_275/_article>. Acesso em: 20 jun. 2010. doi: 10.1508/cytologia.71.275.
- REEVES, A.; TEAR, J. MicroMensure for Windows, version 3.3 2000. Disponível em: <http://www.colostate.edu/Depts/Biology/MicroMeasure>. Acesso em: 10 jan. 2010.
- SINGH, D. The relative importance of characters affecting genetic divergence. Indian Journal of Genetic and Plant Breeding, v.41, p.237-245, 1981.
- SOUZA, S.A.M. Caracterização citogenética, química e molecular em acessos de Capsicum chinense Jacq. 2008. 55f. Dissertação (Mestrado em Genética e Melhoramento de Plantas) - Curso de Pós-graduação em Genética e Melhoramento de Plantas, Universidade Estadual do Norte Fluminense Darcy Ribeiro, Rio de Janeiro, RJ.
- TEODORO-PARDO, C.V.D. et al. Polimorfismo cromossômico en Capsicum annuum L.(Solanaceae) em recolectas de Puebla, Morelos y Quartenário, México. Agrociencia, v.41, p. 873-881, 2007. Disponível em: <http://www.colpos.mx/agrocien/agrociencia.htm>. Acesso em: 03 mar. 2009.
- WARD, J.H. Hierarquical grouping to optimize an objective function. Journal of the American Statistical Association, v.58, p.236-244, 1963.
Datas de Publicação
-
Publicação nesta coleção
11 Nov 2011 -
Data do Fascículo
Out 2011
Histórico
-
Recebido
11 Maio 2011 -
Aceito
26 Jul 2011