Resumos
Este trabalho objetivou a caracterização molecular do vírus linfotrópico de células T humanas infectando doadores de sangue atendidos na Fundação Centro de Hemoterapia e Hematologia do Pará. Amostras de DNA de 79 indivíduos soropositivos para o vírus linfotrópico de células T humanas foram analisadas por meio da reação em cadeia da polimerase para as regiões genômicas pX, env e 5'LTR, de polimorfismos de comprimento de fragmentos de restrição e do seqüenciamento da região 5LTR, com posterior análise filogenética, definindo o tipo e o subtipo do HTLV circulante na população estudada. Observou-se uma maior prevalência de HTLV-1 (71%) em relação ao HTLV-2 (29%). As amostras de HTLV-1 sequenciadas foram classificadas como pertencentes ao subtipo Cosmopolita, subgrupo Transcontinental, sendo as de HTLV-2 identificadas como HTLV-2c. A análise de polimorfismos de comprimento de fragmentos de restrição da região env e do sequenciamento da região 5'LTR, identificou, pela primeira vez na Amazônia Brasileira, uma amostra de HTLV-2b, enfatizando a necessidade de estudos moleculares contínuos na região para melhor entendimento da epidemiologia de transmissão do HTLV na população e permitir a vigilância epidemiológica da emergência de novos tipos e subtipos.
Doadores de sangue; Região Amazônica; Vírus linfotrópico de células T humanas; Caracterização molecular
This study aimed to perform molecular characterization on the human T-cell lymphotropic virus (HTLV) infecting blood donors attended at the Hematology and Hemotherapy Center-Foundation of Pará. DNA samples from 79 HTLV-seropositive individuals were analyzed by means of the polymerase chain reaction on the pX, env and 5'LTR genomic regions; restriction fragment length polymorphism analysis; and sequencing of the 5'LTR region with subsequent phylogenetic analysis. From this, the HTLV types and subtypes circulating in the study population were defined. There was higher prevalence of HTLV-1 (71%) than of HTLV-2 (29%). HTLV-1 samples were classified as belonging to the Cosmopolitan subtype, Transcontinental subgroup; and the HTLV-2 samples as HTLV-2c. Analysis on the restriction fragment length polymorphisms of the env region and sequencing of the 5'LTR region identified a sample of HTLV-2b, for the first time in the Brazilian Amazon region. This emphasizes the need for ongoing molecular studies in this region, in order to have better understanding of the epidemiology of HTLV transmission in the population, and to enable epidemiological surveillance of the emergence of new types and subtypes.
Blood donors; Amazon region; Human T-cell lymphotropic virus among donors; Molecular characterization
ARTIGO ARTICLE
Caracterização molecular do HTLV-1/2 em doadores de sangue em Belém, Estado do Pará: primeira descrição do subtipo HTLV-2b na região Amazônica
Molecular characterization of HTLV-1/2 among blood donors in Belém, State of Pará: first description of HTLV-2b subtype in the Amazon region
Ethienne Lobato dos SantosI; Bruna Tamegão-LopesII; Luiz Fernando Almeida MachadoI; Marluísa de Oliveira Guimarães IshakI; Ricardo IshakI; José Alexandre Rodrigues de LemosII; Antonio Carlos Rosário VallinotoI
ILaboratório Virologia, Instituto de Ciências Biológicas, Universidade Federal do Pará, Belém, PA
IICentro de Hematologia e Hemoterapia do Estado do Pará, Belém, PA
Endereço para correspondência Endereço para correspondência: Dr. Antonio Carlos Rosário Vallinoto Rua Oliveira Belo 238/1002, Umarizal, 66050-380 Belém, PA, Brasil Tel: 55 91 3201-7587 e-mail: vallinoto@ufpa.br
RESUMO
Este trabalho objetivou a caracterização molecular do vírus linfotrópico de células T humanas infectando doadores de sangue atendidos na Fundação Centro de Hemoterapia e Hematologia do Pará. Amostras de DNA de 79 indivíduos soropositivos para o vírus linfotrópico de células T humanas foram analisadas por meio da reação em cadeia da polimerase para as regiões genômicas pX, env e 5'LTR, de polimorfismos de comprimento de fragmentos de restrição e do seqüenciamento da região 5LTR, com posterior análise filogenética, definindo o tipo e o subtipo do HTLV circulante na população estudada. Observou-se uma maior prevalência de HTLV-1 (71%) em relação ao HTLV-2 (29%). As amostras de HTLV-1 sequenciadas foram classificadas como pertencentes ao subtipo Cosmopolita, subgrupo Transcontinental, sendo as de HTLV-2 identificadas como HTLV-2c. A análise de polimorfismos de comprimento de fragmentos de restrição da região env e do sequenciamento da região 5'LTR, identificou, pela primeira vez na Amazônia Brasileira, uma amostra de HTLV-2b, enfatizando a necessidade de estudos moleculares contínuos na região para melhor entendimento da epidemiologia de transmissão do HTLV na população e permitir a vigilância epidemiológica da emergência de novos tipos e subtipos.
Palavras-chaves: Doadores de sangue. Região Amazônica. Vírus linfotrópico de células T humanas. Caracterização molecular.
ABSTRACT
This study aimed to perform molecular characterization on the human T-cell lymphotropic virus (HTLV) infecting blood donors attended at the Hematology and Hemotherapy Center-Foundation of Pará. DNA samples from 79 HTLV-seropositive individuals were analyzed by means of the polymerase chain reaction on the pX, env and 5'LTR genomic regions; restriction fragment length polymorphism analysis; and sequencing of the 5'LTR region with subsequent phylogenetic analysis. From this, the HTLV types and subtypes circulating in the study population were defined. There was higher prevalence of HTLV-1 (71%) than of HTLV-2 (29%). HTLV-1 samples were classified as belonging to the Cosmopolitan subtype, Transcontinental subgroup; and the HTLV-2 samples as HTLV-2c. Analysis on the restriction fragment length polymorphisms of the env region and sequencing of the 5'LTR region identified a sample of HTLV-2b, for the first time in the Brazilian Amazon region. This emphasizes the need for ongoing molecular studies in this region, in order to have better understanding of the epidemiology of HTLV transmission in the population, and to enable epidemiological surveillance of the emergence of new types and subtypes.
Key-words: Blood donors. Amazon region. Human T-cell lymphotropic virus among donors. Molecular characterization.
Os vírus linfotrópicos de células T humanas 1 e 2 (HTLV-1, HTLV-2), pertencem à família Retroviridae, gênero Deltaretrovirus1 3 e possuem propriedades biológicas semelhantes, apresentando tropismo por linfócitos T e uma associação com raras doenças linfoproliferativas2 4.
A infecção pelo HTLV-1 é endêmica no Japão, Caribe, na América do Sul, na África Sub-saariana e na Melanésia1 2 12 16 17 28, onde está associada ao linfoma e leucemia de células T do adulto (LLcTA), uma neoplasia de linfócitos T CD4+ maduros e, também, à paraparesia espástica tropical ou mielopatia associada ao HTLV-1 (PET/MAH), caracterizada por distúrbios neurodegenerativos crônicos9 10 17. O HTLV-2, menos patogênico que o HTLV-1, é prevalente entre usuários de drogas intravenosas (UDIV) das áreas urbanas dos Estados Unidos e da Europa, sudeste da Ásia e entre diversas populações nativas das Américas, assim como, em tribos pigméias da África Central e em doadores de sangue nas Américas do Norte e do Sul e na Europa7 15 18 19 34.
No Brasil, a infecção pelo HTLV-1 é considerada endêmica, porém com baixo índice de prevalência na população geral, quando comparado com as taxas observadas no Japão11 32. A infecção pelo HTLV-1 e pelo HTLV-2 encontra-se presente em todas as regiões brasileiras, mas as prevalências variam de um estado para o outro, sendo mais elevadas na Bahia, em Pernambuco e no Pará3.
Doadores de sangue no Brasil têm sido avaliados, sorologicamente, para o HTLV-1 e para o HTLV-2 desde 1993. De acordo com dados da Fundação Centro de Hematologia e Hemoterapia de Minas Gerais (HEMOMINAS), a soropositividade para o HTLV mostrou-se associada à história pregressa de transfusão de sangue e, em nível educacional, como um marcador de condição socioeconômica e ao uso de drogas ilegais não endovenosas, confirmando assim, a importância de um monitoramento e refinamento do processo de seleção dos doadores de sangue4. Estudos realizados em dois hemocentros do país têm identificado prevalências que variam de 0,07% a 0,13% (HTLV-1) e de 0,02% a 0,03% (HTLV-2)6 8.
A primeira detecção, em Belém, da infecção pelo HTLV em doadores de sangue descreveu, através da amplificação parcial das regiões genômicas pX e env, a ocorrência do subtipo molecular HTLV-2c19. Assim, o presente estudo teve como objetivo confirmar por método molecular a infecção pelo HTLV em doadores de sangue soropositivos para esse vírus, assim com definir o tipo e o subtipo de HTLV circulante no grupo e as relações filogenéticas deste com os isolados descritos na literatura.
MATERIAL E MÉTODOS
Coleta e caracterização das amostras. O grupo analisado foi composto por 79 indivíduos, de ambos os sexos, com idade acima de 18 anos, doadores de sangue da Fundação Centro de Hemoterapia e Hematologia do Pará (HEMOPA) - demanda espontânea - que apresentaram sorologia positiva (ELISA, Murex, Dartford, UK) para a infecção pelo HTLV-1/2, no período de 12 meses.
Estes doadores foram convocados e, após assinarem o termo de consentimento (aprovado pelo Comitê de Ética em pesquisa com seres humanos da Fundação Centro de Hemoterapia e Hematologia do Pará), foram submetidos à coleta de 5mL de sangue em EDTA para a obtenção de sangue total e posterior extração de DNA genômico (Realizada na Gerência de Biologia Molecular e Celular da Fundação HEMOPA, com o kit GFX - GE Health Care). O DNA foi armazenado a -20ºC e posteriormente encaminhado ao Laboratório de Virologia do Instituto de Ciências Biológicas da Universidade Federal do Pará, onde foi realizada a confirmação molecular da infecção por nested PCR.
Caracterização molecular da infecção. A reação de amplificação gênica foi efetuada em duas etapas (nested PCR), para a amplificação de 159pb da região pX do DNA proviral do HTLV-1/2, seguida da digestão enzimática com TaqI para a caracterização do tipo viral. O subtipo viral foi determinado a partir das amostras caracterizadas como HTLV-1 e HTLV-2 utilizando-se uma nested PCR e RFLP do gene env e o seqUenciamento parcial do segmento 5'LTR como descrito previamente22.
Sequenciamento. Após a purificação do produto da PCR (região 5'LTR), o DNA amplificado foi submetido ao sequenciamento automático. A metodologia utilizada baseou-se na síntese bioquímica da cadeia de DNA através do método de Sanger e cols26 pelo kit da ABI PRISMTM 310 BigDye Terminator v3.1 Matrix Standards (Applied Biosystems), usando-se o equipamento ABI PRISM 310 Genetic Analyzer (Applied Biosystems).
Análise das sequências nucleotídicas. O alinhamento das seqüências nucleotídicas da região 5'LTR, foi efetuada usando o programa Clustal X36. O grau de divergência genética, a partir das sequências obtidas e o padrão do mapa de restrição foi determinado usando-se o programa BioEdit 5.09v13. O perfil filogenético das cepas foi realizado por meio do método de agrupamento de vizinhos (Neighbor-Joining - NJ), usando 1.000 réplicas do banco de dados (bootstrap) com o auxílio do programa MEGA - Molecular Evolutionary Genetics Analysis versão 4.035.
RESULTADOS
Todas as 79 amostras submetidas à reação em cadeia da polimerase (PCR) para região pX amplificaram. Após a análise do polimorfismo de comprimento de fragmentos de restrição (RFLP), 56 (71%) amostras confirmaram a infecção pelo HTLV-1 e 23 (29%) pelo HTLV-2.
Dentre as 23 amostras de HTLV-2, 20 amplificaram para a região de 630pb do gene env e, após análise de RFLP com a enzima XhoI, 19 amostras, apresentaram padrão de restrição dos subtipos HTLV-2a/2c e uma única amostra (BBD 7003), apresentou perfil de ausência do sítio de restrição, sendo então identificada como HTLV-2b. Esta amostra, segundo dados obtidos a partir do inquérito epidemiológico, era procedente de uma mulher casada, profissional da área de saúde, que mencionou: a) já ter sofrido acidente de trabalho, b) nunca ter feito uso de preservativos em suas relações sexuais e c) já ter recebido duas transfusões sanguíneas, há aproximadamente dez anos.
Foram amplificadas 51 amostras para a região 5'LTR do HTLV-1, confirmando assim, a infecção por esse vírus. Dentre estas, 25 foram sequenciadas e 14 submetidas à análise filogenética. Para a região 5'LTR do HTLV-2, foram amplificadas 18 amostras, confirmando portanto a infecção por esse vírus. Destas, 17 amostras foram submetidas à análise filogenética. Foram usadas para as análises filogenéticas aquelas amostras que apresentaram melhor rendimento quanto ao tamanho do fragmento sequenciado. As demais foram submetidas ao BLAST - Basic Local Alignment Search Tool (http://blast.ncbi.nlm.nih.gov/Blast.cgi), para caracterização do subtipo viral.
Através da reação de sequenciamento das bases nucleotídicas das regiões 5LTR do HTLV-1 e do HTLV-2, foi possível reconstruir um segmento de cerca de 730pb dessa região para o HTLV-1 e outro de 780pb para o HTLV-2. As amostras de HTLV-1 sequenciadas para a região 5LTR foram comparadas entre si, com a cepa protótipo HTLV-1ATK30 e com outras sequências disponíveis no GenBank, sendo o mesmo feito com as amostras de HTLV-2, cujas sequências correspondentes à região 5LTR foram comparadas com as cepas protótipo do HTLV-2a (HTLV-2MOT)31 e HTLV-2b (HTLV-2NRA)23, assim como com outras sequências disponíveis no GenBank.
A análise entre as sequências nucleotídicas da região 5'LTR do HTLV-1 das amostras isoladas no presente estudo, identificou uma similaridade média de 95,1% e, quando comparadas com o protótipo HTLV-1ATK, mostrou uma identidade média de 95,2%. Em relação aos isolados do HTLV-2, a análise comparativa entre as sequências isoladas no estudo demonstrou uma similaridade média de 97,7% e a comparação com as cepas protótipo HTLV-2a e HTLV-2b, mostrou um percentual de 95,3% e 92,9%, respectivamente.
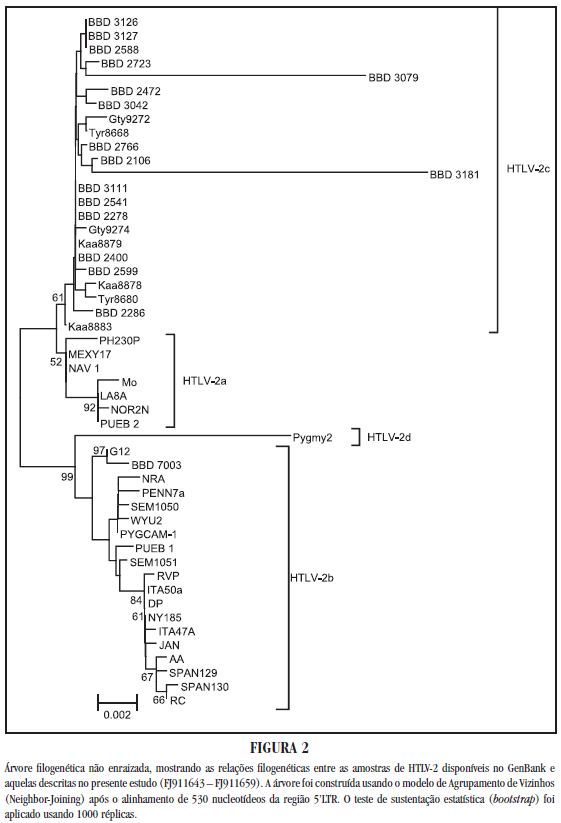
A comparação das sequências nucleotídicas da região 5LTR das amostras de HTLV-1 analisadas no presente estudo, com outros 50 isolados disponíveis no Genbank, possibilitou a construção de uma árvore filogenética que mostra claramente que todos os exemplares aqui isolados agruparam-se com isolados do subtipo Cosmopolita, subgrupo Transcontinental, com valor de boostrap de 63% (Figura 1). Em relação à análise filogenética das sequências nucleotídicas da região 5LTR das amostras de HTLV-2 com outros 56 isolados disponíveis no Genbank, demonstrou que 16 isolados do presente estudo agruparam-se com outras cepas descritas como HTLV-2c, com um valor de boostrap de 61% e uma amostra agrupou com as cepas do subtipo HTLV-2b (Figura 2).
DISCUSSÃO
Por meio da análise molecular das regiões genômicas pX, env e 5LTR do HTLV, foi possível definir o tipo e o subtipo de HTLV circulantes na população de candidatos à doação de sangue da Fundação HEMOPA.
No presente estudo, observou-se uma maior prevalência de HTLV-1 em relação ao HTLV-2, semelhante ao encontrado em doadores de sangue no país de Gales e na Inglaterra40, assim como em outros estudos desenvolvidos no Brasil6 8 27 37. Entretanto, esses resultados contrastam com os obtidos na área urbana de Belém em pacientes co-infectados com o HIV-1, cuja prevalência de HTLV-2 foi maior em relação ao HTLV-137 22, semelhante aos resultados obtidos nos Estados Unidos40.
Algumas amostras, tanto de HTLV-1, quanto de HTLV-2, não chegaram a ser sequenciadas, devido à dificuldade de amplificação da região 5LTR em virtude, talvez, da presença de mutações em sítios específicos dos iniciadores ou à existência de uma baixa carga proviral (< 10 cópias/0,7µg DNA), o que pode ter dificultado, assim, a amplificação dessa região genômica5. Para tanto, foram feitas inúmeras tentativas de amplificação gênica, o que acabou por acarretar a perda de muitas dessas amostras.
Dentre as amostras de HTLV-1 analisadas no presente estudo, todas agruparam-se ao subtipo Cosmopolita, subgrupo Transcontinental, corroborando estudos anteriores que demonstraram uma maior freqüência desse subtipo viral em pacientes com sintomas de PET/MAH e assintomáticos no Brasil21 29 33. Além disso, este subtipo tem sido o único encontrado nas áreas urbana e rural do Estado do Pará22 24, exceto entre os imigrantes japoneses residentes em Tomé-Açú, onde foi identificado o subgrupo Japonês39.
Em relação às amostras de HTLV-2 analisadas, aquelas que tiveram a região 5LTR amplificada agruparam-se com as outras cepas descritas como HTLV-2c, corroborando, também, os resultados anteriores obtidos em doadores de sangue na Cidade de Belém, Pará37. A Amazônia Brasileira é a maior área endêmica para o HTLV-2c20 e a ocorrência desse subtipo na população brasileira, especialmente na Região Amazônica, pode ser atribuída a um efeito de fundador após os eventos migratórios humanos que povoaram a região, atribuindo-se, assim, a hipótese de que se trata de um subtipo molecular único, cuja origem estaria relacionada ao povoamento da região pelos ancestrais ameríndios, disseminando-se nas áreas urbanas do Brasil por meio da miscigenação inter-étnica19 38.
No presente estudo, entre as amostras de HTLV-2 analisadas, foi evidenciado, pela primeira vez na Amazônia Brasileira, um perfil molecular de infecção característico do subtipo HTLV-2b. A presença deste subtipo no Brasil foi recentemente descrita entre doadores de sangue em Porto Alegre (RS), sendo atribuída à migração a partir da fronteira com a Argentina25.
Embora seja difícil precisar a rota de transmissão do HTLV-2b nessa paciente, o presente resultado mostra claramente a importância epidemiológica de estudos contínuos de investigação da emergência de novos agentes infecciosos em novas áreas geográficas, principalmente quando se considera que Belém, provavelmente, representa a principal porta de entrada deste vírus na Amazônia Brasileira.
AGRADECIMENTOS
Aos doadores de sangue que concordaram em participar da pesquisa.
Recebido em: 20/01/09
Aceito em 29/04/2009
Apoio Financeiro: PN-DST & Aids/MS e CNPq.
- 1. Blattner WA, Blayney DW, Robert-Guroff M, Sarngadharan MG, Kalyanaraman VS, Sarin PS, Jaffe ES, Gallo RC. Epidemiology of human T-cell leukemia/lymphoma virus. Journal of Infectious Diseases 147: 406-416, 1983.
- 2. Blattner WA, Kalyanaraman VS, Robert-Guroff M, Lister TA, Galton DA, Sarin PS, Crawford MH, Catovsky D, Greaves M, Gallo RC. The human type-C retrovirus, HTLV, in Blacks from the Caribbean region, and relationship to adult T-cell leukemia/lymphoma. International Journal of Cancer 30: 257-264, 1982.
- 3. Carneiro-Proietti ABF, Ribas JGR, Catalan-Soares BC, Martins ML, Brito-Melo GEA, Martins-Filho OA. Infecção e doença pelos vírus linfotrópicos humanos de células T (HTLV-I/II) no Brasil. Revista da Sociedade Brasileira de Medicina Tropical 35: 499-508, 2002.
- 4. Catalan-Soares BC, Carneiro-Proietti ABF, Proietti FA, GIPH (Interdisciplinary HTLV-I/II Research Group). HTLV-I/II and blood donors: determinants associated with seropositivity in a low risk population. Revista de Saúde Pública 37: 470-476, 2003.
- 5. Cimarelli A, Duclos CA, Gessain A, Casoli C, Bertazzoni U. Clonal expansion of human T-cell leukemia virus type II in patients with high proviral load. Virology 233: 362-364, 1996.
- 6. Colin DD, Alcântara Jr LC, Santos FLN, Uchôa R, Tavares-Neto J. Prevalência da infecção pelo vírus linfotrópico humano de células T e fatores de risco associados à soropositividade em doadores de sangue da cidade de Rio Branco, AC, Brasil (1998-2001). Revista da Sociedade Brasileira de Medicina Tropical 36: 677-683, 2003.
- 7. Egan JF, O'Leary B, Lewis MJ, Mulcahy F, Sheehy N, Hasegawa H, Fitzpatrick F, O'Connor JJ, O'Riordan J, Hall WW. AIDS Research and Human Retroviruses. 15: 699-705, 1999.
- 8. Ferreira JR, Vas RS, Carvalho MB, Guerra C, Fabron AL, Rosemblit J. Human T-lymphoropic vírus type I and type II infections and correlation with risk factors in blood donors from São Paulo, Brazil. Transfusion 35: 258-263, 1995.
- 9. Ferreira Jr OC, Planelles V, Rosenblatt JD. Human T-cell leukemia viruses: epidemiology, biology and pathogenesis. Blood Reviews 11: 91-104, 1997.
- 10. Gessain A, Barin F, Vernant JC, Gout O, Maurs L, Calender A, De The G. Antibodies to Human T-Lymphotropic Virus Type I in patients with tropical spastic paraparesis. Lancet 359: 407-409, 1985.
- 11. Goto K, Sato K, Kurita M, Masuhara N, Iijima Y, Saeki K, Ohno S. The seroprevalence of human T-cell leukaemia/lymphoma virus type I in patients with ocular diaseses, pregnant women and healthy volunteers in Kanto district, central Japan. Scandinavian Journal of Infectious Diseases 29: 219-221, 1997.
- 12. Gotuzzo E, Arango C, Queiroz-Campos A, Istúriz R. Human T-cell lymphotropic virus-I in Latin America. Infectious disease clinics of North América 14: 211-239, 2000.
- 13. Hall TA. BioEdit: a user-friendly biological sequence alignment editor and analysis program for Windows 95/98/NT. Nucleic Acids Symposium Series 41: 95-98, 1999.
- 14. Hall WW, Ishak R, Zhu SW, Novoa P, Eiraku N, Takahashi H, Ferreira MC, Azevedo V, Ishak MO, Ferreira OC, Monken C, Kurata T. Human T-lymphotropic virus type II (HTLV-II): epidemiology, molecular properties, and clinical features of infection. Journal of Acquired Immune Deficiency Syndromes and Human Retrovirology 13 (suppl 1): S204-S214, 1996.
- 15. Hall WW, Ishak R, Zhu SW, Novoa P, Eiraku N, Takahashi H, Nagashima K, Gallo RC. Multiple isolates and characteristics of human T-cell leukemia virus type II. Journal of Virology 66: 2456-2463, 1992.
- 16. Hinuma Y, Komoda H, Chosa T, Kondo T, Kohakura M, Takenaka T, Kikuchi M, Ichimaru M, Yunoki K, Sato I, Matsuo R, Takiuchi Y, Uchino H, Hanaoka M. Antibodies to adult T-cell leukemia-virusassociated antigen (ATLA) in sera from patients with ATL and controls in Japan: a nation-wide sero-epidemiologic study. International journal of cancer. Journal International du Cancer 29: 631-635, 1982.
- 17. International Agency for Research on Cancer. Human immunodeficiency viruses and human Tcell lymphotrophic viruses Lyons: IARC Monographs on the Evaluation of Carcinogenic Risk to Humans,Vol 67, 1996.
- 18. Ishak R, Harrington Jr WJ, Azevedo VN, Eiraku N, Ishak MO, Guerreiro JF, Santos SEB, Kubo T, Monke C, Alexander S, Hall WW. Identification of human T cell lymphotropic virus type IIa infection in the Kayapo, an indigenous population of Brazil. AIDS Research and Human Retroviruses 11:813-821, 1995.
- 19. Ishak R, Ishak MOG, Azevedo VN, Santos DEM, Vallinoto ACR, Saraiva JCP, Crescente JÁ, Hall WW. Detection of HTLV-IIa in blood donors in an urban area of the Amazon Region of Brazil (Belém, PA). Revista da Sociedade Brasileira de Medicina Tropical 31: 193-197, 1998.
- 20. Ishak R, Vallinoto ACR, Azevedo VN, Ishak MOG. Epidemiological aspects of retrovirus (HTLV) infection among Indian populations in the Amazon Region of Brazil Aspectos epidemiológicos da infecção pelo retrovírus HTLV entre populações indígenas da Amazônia brasileira. Cadernos de Saúde Pública 19: 901-914, 2003.
- 21. Kashima S, Alcântara LCJ, Takayanagui OM, Carreto R, Galvão-Castro B, Covas DT. Phylogenetic analysis of Human T-cell lymphotropic viruses type I (HTLV-I). AIDS Research and Human Retroviruses 17 (supl 1): S38, 2001.
- 22. Laurentino RV, Lopes IGL, Azevedo VN, Machado LFA, Lobato L, Ishak MOG, Ishak R, Vallinoto ACR. Molecular characterization of human T-cell lymphotropic virus coinfecting human immunodeficiency virus 1 infected patients in the Amazon region of Brazil. Memórias do Instituto Oswaldo Cruz 100: 371-376, 2005.
- 23. Lee H, Idler KB, Swanson P, Aparicio JJ, Chin KK, Lax JP, Nguyen M, Mann T, Leckie G, Zanetti A, Marinucci G, Chen ISY, Rosenblatt JD. Complete nucleotide sequence of HTLV-II isolate NRA: comparison of envelope sequence variation of HTLV-II isolates from US blood donors US and Italian i. v. drug users. Virology 196: 57-69, 1993.
- 24. Pontes GS, Muto NA, Lopes IGL, Azevedo VN, Machado LFA, Ishak MOG, Ishak R, Vallinoto ACR. Seroepidemiology and molecular epidemiology of HTLV infection in rural and urban populations from the Amazon Region of Brazil. Virus Review and Research 8 (supl 1): 211, 2003.
- 25. Rener JDP, Laurino JP, Menna-Barreto M, Schmitt VM. Molecular evidence of HTLV-II subtype B among an urban population living in South Brazil. AIDS Research and Human Retroviruses 22: 301-306, 2006.
- 26. Sanger F, Nichlen S, Coulson AR. DNA sequences with chain termination inhibitors. Proceedings of the National Academy of Sciences of the United States of America 74: 5463-5468, 1977.
- 27. Santos TJ, Costa CM, Goubau P, Vandamme AM, Desmyter J, Van Doren S, Mota RM, Castro Costa FB, Oliveira AC, Barreto V, Gomes AF, Carneiro-Proietti AB, Bruin VM, Sousa FC, Oriá RB. Western Blot Seroindeterminate Individuals for Human T-lymphotropic Virus 1/2 (HTLV-1/2) in Fortaleza (Brazil): A Serological and Molecular Diagnostic and Epidemiological Approach. Brazilian Journal of Infectious Diseases 7: 202-209, 2003.
- 28. Saxinger W, Blattner WA, Levine PH, Clark J, Biggar R, Hoh M, Moghissi J, Jacobs P, Wilson L, Jacobson R, Crookes R, Strong M, Ansari AA, Dean AG, Nkrumah FK, Mourali N, Gallo RC. Human T-cell leukemia virus (HTLV-I) antibodies in Africa. Science 225:1473-1476, 1984.
- 29. Segurado ACC, Biasutti C, Zeigler R, Rodrigues C, Damas CD, Jorge MLSG, Marchiori PE. Identification of human T-lymphotropic virus type I (HTLVI) subtypes using restricted fragment length polymorphism in a cohort of asymptomatic carriers and patients with HTLV-I associated myelopathy/tropical spastic paraparesis from São Paulo, Brazil. Memórias do Instituto Oswaldo Cruz 97: 329-333, 2002.
- 30. Seike M, Hattori S, Hiraiama Y, Yoshida M. Human adult T-cell leukemia virus: complete nucleotide sequence of the provirus genome integrated in leukemia cell DNA. Proceedings of the National Academy of Sciences of the United States of America 80: 3618-3622, 1983.
- 31. Shimotohno K, Takahashi Y, Shimizu N, Gojobori T, Golde DW, Chen IS. Complete nucleotide sequence of an infectious clone of human T-cell leukemia virus type II: an open reading frame for the protease gene. Proceedings of the National Academy of Sciences of the United States of America 82: 3101-3105, 1985.
- 32. Silva FA, Meis E, Dobbin JA, Oliveira MSP. Leucemia-linfoma de células T do adulto no Brasil: epidemiologia, tratamento e aspectos controversos. Revisiones sobre Biología Celular 48: 585-595, 2002.
- 33. Souza LA, Lopes IGL, Maia EL, Azevedo VN, Machado LFA, Ishak MOG, Ishak R, Vallinoto ACR. Caracterização molecular do HTLV-1 em pacientes com paraparesia espástica tropical/mielopatia associada ao HTLV-1 em Belém, Pará. Revista da Sociedade Brasileira de Medicina Tropical 39: 504-506, 2006.
- 34. Switzer WM, Black FL, Pieniazek D, Biggar RJ, Lal RB, Heneine W. Endemicity and phylogeny of human T cell lymphotropic virus type II subtype A from the Kayapo Indians of Brazil: evidence for limited regional dissemination. AIDS Research and Human Retroviruses 12: 635-640, 1996.
- 35. Tamura K, Dudley J, Nei M, Kuma S. MEGA4: Molecular evolutionary genetics analysis (MEGA) software version 4.0. Molecular Biology and Evolution 24: 1596-1599, 2007.
- 36. Thompson JD, Gibson TJ, Plewniak F, Jeanmougin F, Higgins DG. The CLUSTAL_X windows interface: flexible strategies for multiple sequence alignment aided by quality analysis tools. Nucleic Acids Research 25: 4876-4882, 1997.
- 37. Vallinoto ACR, Azevedo VN, Santos DEM, Caniceiro S, Mesquita FCL, Hall WW, Ishak MOG, Ishak R. Serological Evidence of HTLV-I and HTLV-II Coinfections in HIV-1 Positive Patients in Belém, State of Pará, Brazil. Memórias do Instituto Oswaldo Cruz 93: 407-409, 1998.
- 38. Vallinoto ACR, Ishak MOG, Azevedo VN, Vicente ACP, Otsuki K, Hall WW, Ishak R. Molecular epidemiology of human T-lymphotropic virus type II infection in Amerindian and urban populations of the Amazon region of Brazil. Human Biology 74: 633-644, 2002.
- 39. Vallinoto ACR, Muto NA, Pontes GS, Machado LFA, Azevedo VN, Santos SEB, Ribeiro-dos-Santos AKC, Ishak MOG, Ishak R. Serological and Molecular Evidence of HTLV-I Infection among Japanese Immigrants Living in the Amazon Region of Brazil. Japanese Journal of Infectious Diseases 57: 156-159, 2004.
- 40. Vrielink H, Reesink HW. HTLV-I/II Prevalence in Different Geographic Locations. Transfusion Medicine Reviews 18: 46-57, 2004.
Datas de Publicação
-
Publicação nesta coleção
10 Ago 2009 -
Data do Fascículo
Jun 2009
Histórico
-
Recebido
20 Jan 2009 -
Aceito
29 Abr 2009