Resumos
Foram utilizados registros de pesos do nascimento aos 196 dias de idade de 927 cordeiros da raça Santa Inês, controlados de 1983 a 2000, com os objetivos de estimar parâmetros genéticos e predizer valores genéticos dos animais por meio de modelos de regressão aleatória e compará-los aos obtidos por meio de modelos bicaracterísticas. Os modelos de regressão aleatória foram ajustados por intermédio de polinômios de Legendre. As estimativas de herdabilidade do efeito genético direto aumentaram do nascimento aos 196 dias de idade. As herdabilidades para o efeito materno aumentaram do nascimento aos 56 dias, decrescendo em seguida com a idade. As herdabilidades para o efeito direto obtidas pelas análises bicaracterísticas e regressão aleatória apresentaram tendência oposta. As estimativas obtidas pelas análises bicaracterísticas decresceram do nascimento aos 196 dias de idade, e as obtidas pelos modelos de regressão aleatória aumentaram. As herdabilidades para efeito materno estimadas pelos modelos de regressão aleatória e bicaracterísticas apresentaram o mesmo comportamento, porém em diferentes magnitudes. A correlação de ordem entre os valores genéticos preditos pelos dois modelos foi baixa. As estimativas de herdabilidade e correlações genéticas obtidas pelo modelo de regressão aleatória foram mais coerentes quando comparadas àquelas obtidas pelo modelo bicaracterística.
ovino deslanado; efeito materno; herdabilidade; modelo animal; polinômio de Legendre
Records on weights from birth to 196 days of age of 927 lambs of Santa Ines hairless sheep, from 1983 to 2000, were used to estimate genetic parameters and to predict breeding values by random regression models and to compare these estimates with those obtained by two-traits model. The random regression models were fitted by quadratic and cubic orthogonal Legendre polynomials. The heritability estimates of direct effect increased from birth to 196 days of age. The heritabilities for the maternal effect increased from birth to 56 days, and decreased after that age. The heritabilities for direct effect obtained by two-trait and random regression analyses showed opposite tendency. While the estimates obtained by two-trait analyses decreased from birth to 196 days of age, those obtained by random regression models increased. The heritabilities for maternal effect, obtained by both models, showed the same behavior, however with different magnitude. The Spearman correlation between estimated breeding values by both models was low. The heritability and genetic correlations estimated by random regression model were more consistent than those estimated by two-trait model.
hairless sheep; maternal effect; heritability; animal model; Legendre polynomial
ZOOTECNIA E TECNOLOGIA E INSPEÇÃO DE PRODUTOS DE ORIGEM ANIMAL
Avaliação genética de características de crescimento de ovinos Santa Inês utilizando modelos de regressão aleatória
Genetic evaluation of growth traits of Santa Inês hair sheep using random regression models
J.L.R. SarmentoI, V; R.A. TorresII, V, * * Autor para correspondência ( corresponding author) E-mail: rtorres@ufv.br ; C.S. PereiraII, VI; W.H. SousaIII; P.S. LopesII; C.V. AraújoIV; R.F. EuclydesII, V
IAluno de Pós-Graduação - UFV
IIDepartamento de Zootecnia UFV Av. P.H. Rolfs, s/n 36570-000 Viçosa, MG
IIIEMEPA PB
IVUniversidade Federal Rural do Amazona
VBolsista CNPq
VIBolsista FAPEMIG
RESUMO
Foram utilizados registros de pesos do nascimento aos 196 dias de idade de 927 cordeiros da raça Santa Inês, controlados de 1983 a 2000, com os objetivos de estimar parâmetros genéticos e predizer valores genéticos dos animais por meio de modelos de regressão aleatória e compará-los aos obtidos por meio de modelos bicaracterísticas. Os modelos de regressão aleatória foram ajustados por intermédio de polinômios de Legendre. As estimativas de herdabilidade do efeito genético direto aumentaram do nascimento aos 196 dias de idade. As herdabilidades para o efeito materno aumentaram do nascimento aos 56 dias, decrescendo em seguida com a idade. As herdabilidades para o efeito direto obtidas pelas análises bicaracterísticas e regressão aleatória apresentaram tendência oposta. As estimativas obtidas pelas análises bicaracterísticas decresceram do nascimento aos 196 dias de idade, e as obtidas pelos modelos de regressão aleatória aumentaram. As herdabilidades para efeito materno estimadas pelos modelos de regressão aleatória e bicaracterísticas apresentaram o mesmo comportamento, porém em diferentes magnitudes. A correlação de ordem entre os valores genéticos preditos pelos dois modelos foi baixa. As estimativas de herdabilidade e correlações genéticas obtidas pelo modelo de regressão aleatória foram mais coerentes quando comparadas àquelas obtidas pelo modelo bicaracterística.
Palavras-chave: ovino deslanado, efeito materno, herdabilidade, modelo animal, polinômio de Legendre
ABSTRACT
Records on weights from birth to 196 days of age of 927 lambs of Santa Ines hairless sheep, from 1983 to 2000, were used to estimate genetic parameters and to predict breeding values by random regression models and to compare these estimates with those obtained by two-traits model. The random regression models were fitted by quadratic and cubic orthogonal Legendre polynomials. The heritability estimates of direct effect increased from birth to 196 days of age. The heritabilities for the maternal effect increased from birth to 56 days, and decreased after that age. The heritabilities for direct effect obtained by two-trait and random regression analyses showed opposite tendency. While the estimates obtained by two-trait analyses decreased from birth to 196 days of age, those obtained by random regression models increased. The heritabilities for maternal effect, obtained by both models, showed the same behavior, however with different magnitude. The Spearman correlation between estimated breeding values by both models was low. The heritability and genetic correlations estimated by random regression model were more consistent than those estimated by two-trait model.
Keywords: hairless sheep, maternal effect, heritability, animal model, Legendre polynomial
INTRODUÇÃO
A principal informação para avaliação genética e seleção de ovinos de corte são os pesos corporais obtidos durante o período de crescimento. As pesagens são, normalmente, feitas a intervalos de 28 dias, permitindo que cada animal tenha várias medições de peso, o que caracteriza as chamadas medidas repetidas ou dados longitudinais. Essa terminologia tem sido usada para caracterizar conjuntos de observações provenientes de várias medições tomadas em um mesmo indivíduo ao longo do tempo, como os pesos corporais de um animal medidos em várias idades.
Nos últimos anos, essa estrutura de dados tem recebido tratamento especial por parte de pesquisadores em melhoramento animal. Tendo em vista que os valores genéticos dos animais são tomados como fatores aleatórios e que equações de regressão poderiam ser empregadas na descrição desses fatores, Schaeffer e Dekkers (1994) propuseram a utilização de um modelo animal que tenha coeficientes de regressão de efeito aleatório (Henderson Jr., 1982) para avaliação genética de características que são medidas várias vezes durante a vida do animal.
Os modelos de regressão aleatória, comparados aos modelos multicaracterísticas, apresentam algumas vantagens: abrangem todo o intervalo contínuo em que as medidas forem tomadas, possibilitando predizer o valor genético dos animais em qualquer ponto desse intervalo (Kirkpatrick e Heckman, 1989), permitem a estimação de curvas de crescimento individuais considerando o parentesco existente entre os animais, podem utilizar mais eficientemente as informações disponíveis, sem a necessidade de empregar ajustes arbitrários em idades consideradas como padrão (Meyer e Hill, 1997) e podem analisar os efeitos de ambiente peculiar a cada pesagem (Jamrozik et al., 1997).
Os modelos de regressão aleatória têm sido reconhecidos como ideais para análise de dados longitudinais no melhoramento animal e têm sido utilizados para modelar várias características em algumas espécies de animais domésticos, como bovinos de leite (Jamrozik et al., 1997), bovinos de corte (Albuquerque e Meyer, 2001; Nobre et al., 2003) e ovinos de corte (Lewis e Brotherstone, 2002). No entanto, em ovinos deslanados não se constataram trabalhos que tenham empregado esses modelos na avaliação genética do crescimento.
Os objetivos deste trabalho foram estimar parâmetros genéticos para os efeitos aditivos direto e materno e predizer valores genéticos dos animais por meio de modelos de regressão aleatória para pesos de ovinos Santa Inês, e comparar os resultados aos obtidos com um modelo bicaracterística.
MATERIAL E MÉTODOS
Os dados utilizados neste estudo, relativos ao período de 1983 a 2000, são provenientes de um rebanho experimental de ovinos da raça Santa Inês, pertencentes à Empresa Estadual de Pesquisa Agropecuária da Paraíba (EMEPA-PB), criados em uma fazenda experimental localizada no município de Soledade, semi-árido paraibano.
O arquivo inicial constava de 7.600 registros de pesos do nascimento aos 196 dias de idade. Foram consideradas informações de cordeiros filhos de reprodutores com no mínimo três progênies, de cordeiros filhos de ovelhas com no mínimo duas progênies, de animais com no mínimo três pesagens e de animais nascidos de partos simples e duplo. Também foram mantidos no arquivo grupos de contemporâneos com no mínimo quatro observações. O resumo da estrutura dos dados utilizados está apresentado na Tab. 1.
As pesagens foram realizadas a cada 28 dias. Os pesos corporais foram distribuídos em sete classes de idade da ovelha ao parto, sendo a primeira classe constituída por fêmeas com idade menor ou igual a dois anos e as demais classes acrescidas de um ano, exceto a última que foi constituída por aquelas com mais de sete anos; em duas classes de sexo: machos e fêmeas; em dois tipos de nascimento: nascidos de parto simples e de parto duplo; e em duas estações de pesagens: estação chuvosa (março a julho) e estação seca (agosto a fevereiro).
Os animais permaneceram em pastagem nativa e nativa melhorada durante o período chuvoso. No período seco, os animais receberam, no cocho, suplementação alimentar à base de silagem, palma forrageira (Opuntia ficus sp.), capim-elefante (Peninsetum purpureum) e uma mistura concentrada, além de suplementação mineral durante todo o ano. O manejo sanitário baseou-se no controle da febre aftosa, ecto e endoparasitas e no tratamento clínico da linfadenite caseosa.
A regressão fixa para a trajetória média de crescimento, assim como as regressões aleatórias para os efeitos genéticos aditivos direto e materno e de ambiente permanente de animal foram modeladas por meio de polinômios ortogonais de Legendre, de ordem quadrática (k=3, modelo 1) e cúbica (k=4, modelo 2).
Na modelagem dos efeitos fixos e aleatórios dos pesos do nascimento aos 196 dias de idade, empregou-se o seguinte modelo:
em que
yij é o peso j do animal i, fm é o polinômio de Legendre m da idade padronizada (-1 a +1); Fij é um conjunto de efeitos fixos incluídos no modelo (grupo de contemporâneos, formados pelo ano e estação de pesagem, tipo de nascimento, idade da ovelha ao parto e sexo da cria); bm são os coeficientes de regressão fixos para modelar a trajetória média da população; aim, gim e pim são os coeficientes de regressão aleatória dos efeitos genético aditivo direto e materno e de ambiente permanente de animal, respectivamente, que são específicos para cada cordeiro; kb, ka, km e kc são as ordens de ajustes dos polinômios correspondentes; eij é o efeito aleatório residual.
Em termos matriciais, o modelo de análise pode ser descrito como segue:
 , em que
, em que
 é vetor de N observações referentes a ND animais;
é vetor de N observações referentes a ND animais;  é vetor de efeitos fixos, incluindo os coeficientes de regressão fixos;
é vetor de efeitos fixos, incluindo os coeficientes de regressão fixos;  é vetor ka x Nd de coeficientes de regressão aleatória genéticos aditivos diretos, em que Nd > ND denota o número total de animais na análise, incluindo pais sem registros;
é vetor ka x Nd de coeficientes de regressão aleatória genéticos aditivos diretos, em que Nd > ND denota o número total de animais na análise, incluindo pais sem registros;  é vetor km x ND de coeficientes de regressão aleatória genéticos aditivos maternos;
é vetor km x ND de coeficientes de regressão aleatória genéticos aditivos maternos;  é vetor kc x ND de coeficientes de regressão aleatória de ambiente permanente de animal;
é vetor kc x ND de coeficientes de regressão aleatória de ambiente permanente de animal;  é vetor de erros aleatórios; X, Z1, Z2 e Z3 referem-se às matrizes de incidência dos coeficientes de regressão fixos, coeficientes de regressão aleatória genéticos aditivos direto, materno e de ambiente permanente de animal, respectivamente.
é vetor de erros aleatórios; X, Z1, Z2 e Z3 referem-se às matrizes de incidência dos coeficientes de regressão fixos, coeficientes de regressão aleatória genéticos aditivos direto, materno e de ambiente permanente de animal, respectivamente.
As pressuposições assumidas em relação aos vetores  ,
,  ,
,  e
e  são de que possuem distribuição normal, com E(
são de que possuem distribuição normal, com E( ) = E(
) = E( ) = E(
) = E( ) = E(
) = E( ) =
) =  e Var(
e Var( ) = Ka
) = Ka A, Var(
A, Var( ) = Km
) = Km A, Var(
A, Var( ) = Kc
) = Kc IND e Var(
IND e Var( ) =
) =  , com as covariâncias nulas entre o vetor
, com as covariâncias nulas entre o vetor  e os vetores
e os vetores  e
e  . Ka, Km e Kc são as matrizes de variâncias e covariâncias entre coeficientes de regressão aleatória para os efeitos genéticos aditivos direto, materno e de ambiente permanente de animal, respectivamente; I é uma matriz identidade;
. Ka, Km e Kc são as matrizes de variâncias e covariâncias entre coeficientes de regressão aleatória para os efeitos genéticos aditivos direto, materno e de ambiente permanente de animal, respectivamente; I é uma matriz identidade;  é a variância residual; A é a matriz de numeradores dos coeficientes de parentesco de Wright, entre os animais; e IND é uma matriz identidade.
é a variância residual; A é a matriz de numeradores dos coeficientes de parentesco de Wright, entre os animais; e IND é uma matriz identidade.
Os componentes de (co)variâncias foram estimados pelo método da máxima verossimilhança restrita (REML), utilizando-se a opção DXMRR do pacote estatístico DFREML (Meyer, 1998) e empregando-se um algoritmo livre de derivadas para maximizar o loge da função de verossimilhança.
Os modelos com diferentes ordens de ajustes para os polinômios foram comparados pelo logaritmo da função de verossimilhança (loge L), critério de informação de Akaike (AIC) e critério de informação Bayesiano de Schwarz (BIC). Segundo Albuquerque (2003), os dois últimos impõem penalidades de acordo com o número de parâmetros a serem estimados. Os critérios de informação podem ser definidos como:
AIC = -2logeL + 2p, e
BIC = -2logeL + plog(N - r(X)), em que
p é o número de parâmetros estimados; N o número de observações; r(X) o posto da matriz de incidência dos efeitos fixos do modelo; e loge L o logaritmo da função de máxima verossimilhança restrita.
Para comparar os resultados obtidos pelos modelos de regressão aleatória, foram realizadas, no mesmo conjunto de dados, análises bicaracterísticas por meio de um modelo animal que incluiu os mesmos efeitos fixos e os efeitos aleatórios genéticos direto e materno, levando-se em consideração uma distribuição conjunta entre os pares de características. As estimativas de parâmetros genéticos para determinada característica foram calculadas como a média das análises bicaracterísticas que incluíram a referida característica. Os componentes de (co)variâncias e os parâmetros genéticos foram estimados pelo programa MTDFREML, desenvolvido por Boldman et al. (1995), que utilizaram a metodologia da máxima verossimilhança restrita (REML), por meio de um algoritmo não-derivativo.
Não foi possível incluir, no modelo, o efeito de ambiente permanente materno, em razão do volume de dados disponível. A covariância entre os efeitos genéticos aditivos direto e materno foi assumida como zero.
Para comparar os valores genéticos e calcular o percentual de animais selecionados em comum, as correlações de Pearson e Spearman entre os valores genéticos preditos pelas análises bicaracterísticas e a regressão aleatória foram obtidas para os pesos aos 112 (desmama) e 196 dias de idade, por meio do procedimento CORR do SAS® (User's... 1999).
RESULTADOS E DISCUSSÃO
As estimativas dos valores para o loge L e os critérios de informação utilizados para cada modelo estão sumarizados na Tab. 2. Quando se aumentou a ordem de ajuste do polinômio, k=3 no modelo 1 para k=4 no modelo 2, houve aumento no loge L e diminuição nos valores de AIC, BIC e variância residual. Aplicando o teste de razão de verossimilhança, verificou-se que o valor calculado foi maior que o valor de qui-quadrado tabelado a 1% de probabilidade. De acordo com esses critérios, o modelo 2 proporcionou melhor ajuste, portanto, seria o mais adequado às avaliações genéticas de característica de crescimento dos ovinos Santa Inês em estudo. Lewis e Brotherstone (2002) utilizaram modelos de regressão aleatória na avaliação do crescimento de ovinos Suffolk e verificaram que, ao aumentar a ordem do polinômio de 3 para 5, o ajuste melhorou significativamente.
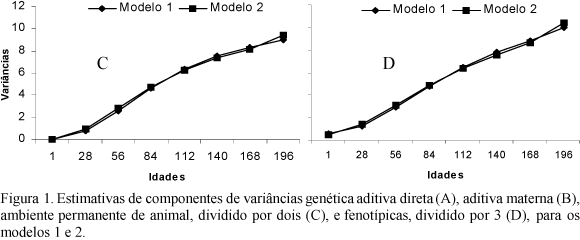
As estimativas das variâncias genéticas aditivas direta e materna, de ambiente permanente de animal e fenotípicas, estão apresentadas na Fig. 1. As variâncias genéticas aditivas diretas estimadas por ambos os modelos seguiram a mesma tendência (Fig. 1A), aumentando do nascimento aos 196 dias de idade; as variâncias estimadas pelo modelo 2 foram ligeiramente superiores às obtidas pelo modelo 1, dos 112 aos 168 dias de idade. Estimativas de variâncias genéticas aditivas diretas crescentes do nascimento aos 150 dias de idade também foram relatadas por Lewis e Brotherstone (2002).
As variâncias estimadas para o efeito materno (Fig. 1B) pelo modelo 1 foram mais constantes ao longo da trajetória de crescimento, em que as estimativas aumentaram até os 140 dias de idade, que é a primeira medida após a desmama, a partir da qual começaram a decrescer. As obtidas pelo modelo 2 apresentaram comportamento semelhante, porém aumentaram até os 84 dias, depois diminuíram até os 168, voltando a aumentar aos 196 dias de idade, entretanto as diferenças foram pequenas. Apesar de os critérios utilizados para avaliar a qualidade do ajuste terem indicado o modelo 2 como o melhor, as variâncias estimadas pelo modelo 1 parecem ser mais coerentes. Lewis e Brotherstone (2002), modelando o efeito materno por meio de funções polinomiais de terceira e quinta ordens, verificaram que não houve aumento significativo no logaritmo da função de verossimilhança quando ajustaram a função de quinta ordem.
As variâncias estimadas para o efeito de ambiente permanente de animal (Fig. 1C) e fenotípicas (Fig. 1D) aumentaram à medida que o animal cresceu e praticamente não houve diferença nas trajetórias obtidas pelos dois modelos. Estes resultados indicaram que a inclusão dos coeficientes de regressão aleatória para esses efeitos permitiram diferenciar as variâncias estimadas ao longo da trajetória de crescimento, evidenciando que funções contínuas devem ser empregadas para modelá-los.
As estimativas de herdabilidade para os efeitos direto e materno e a proporção da variância fenotípica devido à variância de ambiente permanente de animal são apresentadas na Tab. 3. As estimativas obtidas por ambos os modelos de regressão aleatória para a herdabilidade direta foram semelhantes, aumentando do nascimento aos 196 dias de idade. Lewis e Brotherstone (2002) verificaram a mesma tendência, em que as herdabilidades aumentaram ao longo da trajetória de crescimento.
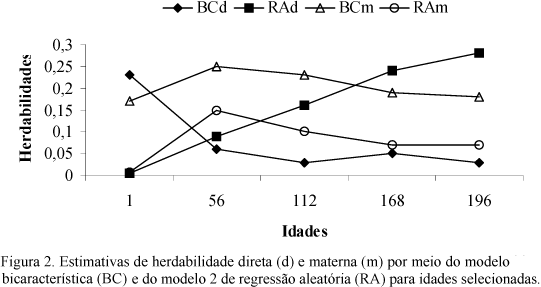
As herdabilidades para efeito materno estimadas pelo modelo 2 aumentaram até os 56 dias e depois decresceram com a idade. As estimadas pelo modelo 1 aumentaram até os 56 dias e permaneceram constantes até a desmama (112 dias), depois decresceram. Apesar de as diferenças serem pequenas, as estimadas pelo modelo 1 são, possivelmente, coerentes, quando comparadas ao comportamento que se conhece em bovinos de corte, em que a proporção da variância fenotípica devido ao efeito materno permanece alta até a desmama. Entretanto, os resultados obtidos pelo modelo 2 podem ter algum sentido para o rebanho estudado. Uma hipótese seria que a contribuição proporcional do efeito materno para a variância fenotípica começou a diminuir antes da desmama. Isso pode ser explicado, provavelmente, pelo fato de que, mesmo estando sob os cuidados maternos, aos 112 dias de idade, os cordeiros já não eram tão dependentes das mães, pois eles as acompanhavam no pastejo, onde tiveram acesso a outros tipos de alimentos, em idades que antecederam à desmama. Com isso a fase de maior dependência em relação à mãe não seria no desmame e sim em idades que antecederam o desmame, o que pode ser bastante positivo, devido ao menor estresse na ocasião do desmame. O resultado obtido pelo modelo 2 de regressão aleatória foi verificado nas análises bicaracterísticas (Fig. 2), porém em diferentes magnitudes. Não foi possível a comparação da modelagem do efeito materno por meio de regressão aleatória, por não terem sido encontrados resultados na literatura consultada. Todavia, Boujenane e Kansari (2002), ao analisarem pesos de ovinos Timahdite, utilizando modelo animal por meio de análises unicaracterística, verificaram maior contribuição do efeito materno para a variância fenotípica aos 70 dias, idade que antecedeu ao desmame.
Apesar de as herdabilidades maternas estimadas por ambos os modelos apresentarem tendência de declínio nas últimas idades estudadas, aos 196 dias de idade houve, ainda, contribuição do efeito materno da ordem de 7% (modelo 2) da variação fenotípica, mostrando que resquícios desse efeito podem ser verificados mesmo após a desmama.
Estimativas da variância de ambiente permanente de animal como proporção das variâncias fenotípicas (c2), para ambos os modelos, apresentaram aumento maior do nascimento aos 28 dias de idade, a partir desses aumentaram levemente até a desmama, tendendo, depois, a decrescer com a idade. Albuquerque (2003) descreveu comportamento similar para esse efeito em dados de bovinos da raça Nelore.
O comportamento das estimativas de herdabilidades para o efeito direto obtidas pelas análises bicaracterísticas e de regressão aleatória pode ser observado na Fig. 2. Enquanto as estimativas obtidas pelas análises bicaracterísticas decresceram do nascimento aos 196 dias de idade, as obtidas pelos modelos de regressão aleatória aumentaram. Com exceção do peso ao nascimento, as estimativas de herdabilidade direta obtidas pelo modelo de regressão aleatória foram todas superiores às obtidas pelas análises bicaracterísticas.
As herdabilidades para efeito materno estimadas pelo modelo de regressão aleatória tenderam a apresentar o mesmo comportamento das obtidas nas análises bicaracterísticas ao longo da trajetória de crescimento, porém foram de menor magnitude. A contribuição do efeito materno aumentou do nascimento aos 56 dias de idade (Fig. 2) e, a partir desse ponto, decresceu com a idade; para todas as idades em que as duas metodologias foram comparadas, as estimativas obtidas pelas análises bicaracterísticas foram maiores.
Ao comparar as trajetórias dos efeitos direto e materno modelados pela regressão aleatória, constatou-se comportamento que parece ser biologicamente mais correto, em que a contribuição do efeito materno foi maior que a do efeito direto no período que antecedeu a desmama. Após os 56 e antes dos 112 dias de idade, a contribuição do efeito materno decresceu e a do efeito aditivo direto aumentou (Fig. 2), o que seria esperado, pois, à medida que o cordeiro cresce, torna-se mais independente da mãe. Ao observar as estimativas de herdabilidade direta e materna obtidas pelas análises bicaracterísticas, não se verificou esse comportamento. Com exceção do peso ao nascer, a herdabilidade do efeito materno foi maior que a do efeito direto para os pesos em todas as idades.
Esses resultados podem indicar que o modelo bicaracterística, provavelmente em razão do baixo número de observações usado, não foi eficiente em separar os efeitos genéticos direto e materno. Nesse tipo de análise, assume-se que os pesos nas diferentes idades são características diferentes. Como o número de observações para cada peso foi pequeno, pode ter havido confundimento entre os efeitos genéticos direto e materno, mesmo usando informações de duas características. Quando os dados foram tratados como longitudinais, em que as pesagens nas diferentes idades foram consideradas como medidas repetidas do peso, o número de observações passou de aproximadamente 900, em cada característica, para 6.945 registros de pesos, confirmando uma das vantagens dessa metodologia para análise de dados que são função da idade: a potencialização do número de informações por animal. Enquanto nas análises bicaracterísticas foram utilizadas duas informações por animal, nas análises por meio dos modelos de regressão aleatória, os animais tinham, no mínimo, três e, no máximo, oito informações de seu peso - aproximadamente 70% dos animais tinham as oito pesagens.
Com base nos resultados apresentados na Fig. 2 e considerando o aumento no número de informações nas análises por meio dos modelos de regressão aleatória, acredita-se que essa metodologia tenha sido mais eficiente para modelar os efeitos aleatórios, assim como na partição dos efeitos genéticos direto e materno, o que mostra que algum confundimento pode acontecer na separação desses efeitos, dependendo do número de informações utilizadas e da metodologia empregada.
As estimativas de correlações genéticas e fenotípicas entre os pesos do nascimento aos 196 dias de idade são apresentadas na Tab. 4. As correlações do peso ao nascimento com os demais pesos mostraram comportamento diferente das correlações entre os pesos dos 28 aos 196 dias. Aquelas foram negativas ou de baixa magnitude, enquanto as últimas variaram de 0,76 a 0,99, sendo mais altas entre idades adjacentes e decrescentes com o aumento de tempo entre elas.
As estimativas de correlações genéticas foram diferentes das obtidas pelas análises bicaracterísticas, em que quase todas as correlações entre as idades foram iguais à unidade (não apresentadas), o que pode ser conseqüência do tamanho da amostra utilizada. Diferentemente dos resultados encontrados neste estudo, Albuquerque (2003) verificou que as estimativas de correlações genéticas entre os pesos obtidas pelos modelos de regressão aleatória foram praticamente iguais às obtidas com análises bicaracterísticas.
Trabalhos encontrados na literatura nacional, que apresentaram estimativas de correlações genéticas entre pesos em determinadas idades em ovinos, utilizaram o método dos quadrados mínimos e não levaram em consideração os efeitos maternos. Em geral, essas estimativas foram de moderada a alta e positivas, variando de 0,33 a 0,92 (Silva et al., 1996; Silva e Araújo, 2000).
As correlações fenotípicas estimadas entre os pesos tenderam a ser altas e positivas entre idades mais próximas, contudo, isso não se verificou entre o peso ao nascer e os pesos obtidos dos 28 aos 196 dias, isto é, as correlações entre essas características foram muito baixas (Tab. 4). Similarmente, as correlações fenotípicas estimadas entre o peso ao nascer e os outros pesos nas análises bicaracterísticas foram menores que as demais correlações, porém foram mais altas que as obtidas pelos modelos de regressão aleatória. Contudo, as correlações entre os pesos dos 28 aos 196 dias, nas análises bicaracterísticas, foram de menor magnitude que as obtidas com os modelos de regressão aleatória, variando de 0,38 a 0,55.
As correlações entre os valores genéticos dos animais aos 112 e 196 dias de idade obtidos com análises bicaracterísticas e regressão aleatória são apresentadas na Tab. 5. Observa-se que tanto as correlações de Spearman quanto as de Pearson, estimadas entre os valores genéticos provenientes das análises bicaracterísticas e regressão aleatória, foram baixas para ambas as características. Esses resultados sugerem que os dois modelos não levaram à seleção dos mesmos animais como os melhores. Correlações entre valores genéticos preditos por meio de análises bicaracterísticas e de regressão aleatória, variando de 0,64 a 0,87, foram obtidas por Albuquerque (2003). De acordo com Nobre et al. (2002), baixas correlações verificadas entre valores genéticos podem ser resultantes das diferenças entre os parâmetros genéticos estimados pelos modelos multicaracterísticas e de regressão aleatória. Meyer (2004), observou, em estudos de simulação, que os modelos de regressão aleatória obtiveram maior acurácia, medida pela correlação dos valores estimados com os simulados, quando comparados a um modelo multicaracterística. Segundo a autora, essa vantagem pode ser atribuída à melhor modelagem das variâncias e dos parâmetros genéticos estimados pelos modelos de regressão aleatória.
Na Tab. 6 são apresentados o número e a percentagem de animais selecionados em comum pelos dois modelos, aplicando-se intensidades de seleção diferentes. Selecionando-se 20% dos melhores animais com base nos valores genéticos preditos por ambos os modelos, seriam coincidentes 34,8 e 39,2% para os pesos aos 112 e 196 dias de idade, respectivamente. Essa coincidência foi menor quando se aumentou a intensidade de seleção, ou seja, quando foram selecionados 10% dos melhores animais, apenas 18,4 e 24,8%, para os mesmos pesos, seriam selecionados por ambos os modelos. Quando se selecionou apenas 1% dos melhores animais, não houve coincidência entre os animais selecionados. Esses resultados mostraram que houve completa mudança na classificação dos animais, o que pode ter importância prática, isto é, os melhores animais selecionados por um modelo não foram os melhores no outro.
Com os modelos de regressão aleatória, os valores genéticos podem ser preditos para qualquer idade dentro do período de crescimento considerado, além de não necessitar de ajustes para idade e considerar os efeitos ambientais específicos do dia da pesagem. Nos modelos multicaracterísticas os valores genéticos preditos são pontuais, ou seja, apenas para as idades em que os pesos forem medidos. Assim, espera-se que os modelos de regressão aleatória estimem parâmetros genéticos e possam predizer os valores genéticos dos animais de forma mais acurada que os modelos bicaracterísticas.
CONCLUSÕES
Os parâmetros genéticos diretos e maternos estimados e os valores genéticos preditos pelo modelo de regressão aleatória mostraram-se mais adequados que os estimados e preditos pelo modelo bicaracterística, indicando que é uma metodologia poderosa para determinar o mérito genético dos animais. Entretanto, as herdabilidades estimadas indicam que a seleção para peso resultará em pequenos ganhos genéticos.
Recebido em 29 de março de 2004
Aceito em 24 de novembro de 2005
Apoio: EMEPA PB e CNPq
- ALBUQUERQUE, L.G. Modelos de dimensão infinita aplicados a características de crescimento de bovinos da raça Nelore. 2003. 83f. Tese (Livre-docente) - Faculdade de Ciências Agrárias e Veterinárias, Universidade Estadual Paulista, Jaboticabal.
- ALBUQUERQUE, L.G.; MEYER, K. Estimates of covariance functions for growth from birth to 630 days of age in Nelore cattle. J. Anim. Sci., v.79, p.2776-2789, 2001.
- BOLDMAN, K.G.; KRIESE, L.A.; Van VLECK, D.L. et al. A manual for use of MTDFREML: a set of programs to obtain estimates of variances and covariances [DRAFT] Lincoln: USDA/ARS, 1995. 120p.
- BOUJENANE, I.; KANSARI, J. Estimates of (co)variances due to direct and maternal effects for body weights in Timahdite sheep. Anim. Sci., v.28, p.409-414, 2002.
- HENDERSON Jr., C.R. Analysis of covariance in the mixed model: higher-level, nonhomogeneous, and random regressions. Biometrics, v.38, p.623-640, 1982.
- JAMROZIK, J.; SCHAEFFER, L.R.; DEKKERS, J.C.M. Genetic evaluation of dairy cattle using test day yields and random regression model. J. Dairy Sci., v.80, p.1217-1226, 1997.
- KIRKPATRICK, M.; HECKMAN, N. A quantitative genetic model for growth, shape, reaction norms, and other infinite-dimensional characters. J. Mathem. Biol., v.27, p.429-450, 1989.
- LEWIS, R.M.; BROTHERSTONE, S. Agenetic evaluation of growth in sheep using random regression techniques. Anim. Sci., v.74, p.63-70, 2002.
- MEYER, K. DXMRR – a program to estimate covariance functions for longitudinal data by REML. In: WORLD CONGRESS OF GENETICS APPLIED TO LIVESTOCK PRODUCTION, 6., 1998, Armidale. Proceedings.. Armidale, 1998. CD-ROM.
- MEYER, K. Scope for a random regression model in genetic evaluation of beef cattle growth. Livest. Prod. Sci., v.86, p.69-83, 2004.
- MEYER, K.; HILL, W.G. Estimation of genetic and phenotypic covariance functions for longitudinal or "repetead" records by restricted maximum likelihood. Livest. Prod. Sci., v.47, p.185-200, 1997.
- NOBRE, P.R.C.; MISZTAL, I.; TSURUTA, S. et al. Analyses of growth curves of Nellore cattle by multiple-trait and random regression models. J. Anim. Sci., v.81, p.918-926, 2003.
- NOBRE, P.R.C.; MISZTAL, I.; TSURUTA, S. et al. Genetic evaluation of growth in beef cattle with random regression model. In: WORLD CONGRESS OF GENETICS APPLIED TO LIVESTOCK PRODUCTION, 7., 2002, Montpellier. Proceedings.. Montpellier, 2002. 1 CD-ROM.
- SCHAEFFER, L.R.; DEKKERS, J.C.M. Random regression in animal models for test day production in dairy cattle. In: WORLD CONGRESS OF GENETICS APPLIED TO LIVESTOCK PRODUCTION, 5., 1994, Guelph. Proceedings.. Guelph, 1994. p.443-446.
- SILVA, F.L.R.; ARAÚJO, A.M. Características de reprodução e de crescimento de ovinos mestiços Santa Inês, no Ceará. Rev. Bras. Zootec., v.29, p.1712-1720, 2000.
- SILVA, F.L.R.; FIGUEIREDO, E.A.P.; SIMPLÍCIO, A.A. et al. Parâmetros genéticos e fenotípicos de características de crescimento de desmame de cordeiros Santa Inês, no Estado do Ceará. Rev. Bras. Zootec., v.25, p.845-852, 1996.
- USER'S guide: statistics. Version 8.0, Cary, NC: SAS Institute, 1999.
Datas de Publicação
-
Publicação nesta coleção
19 Abr 2006 -
Data do Fascículo
Fev 2006
Histórico
-
Recebido
29 Mar 2004 -
Aceito
24 Nov 2005