Resumos
A introdução de nova nomenclatura para os componentes do complexo principal de histocompatibilidade (Major Histocompatibility Complex - MHC) e a descrição de novos métodos moleculares para a tipificação dos alelos do MHC contribuíram, grandemente, para o entendimento e identificação do extenso polimorfismo do sistema. No entanto, para o não especialista, essas novas aquisições têm dificultado o entendimento do papel do MHC em associação com as doenças. Assim, neste artigo, foram revisados os conceitos atuais acerca dos genes e moléculas do MHC, os métodos de tipificação desses marcadores de histocompatibilidade e a nomenclatura vigente para os componentes do sistema. Esses aspectos são extremamente importantes para o entendimento do polimorfismo do MHC, dando embasamento para a compreensão dos mecanismos propostos de associação desses marcadores com as doenças auto-imunes endócrinas como diabetes mellitus do tipo 1, doença de Graves e tireoidite de Hashimoto, doença de Addison, síndrome poliglandular auto-imune e insuficiência ovariana prematura.
HLA; MHC; Diabetes mellitus do tipo 1; Doença de Graves; Tireoidite de Hashimoto; Doença de Addison
The acquisition of a new nomenclature and the development of molecular methods for the assignment of major histocompatibilty complex (MHC) alleles have widely contributed to the understanding of the extensive polymorphism of the system; however, many doubts have arisen to the non-specialist in terms of MHC and disease association. In order to understand the MHC polymorphism and the association with endocrine diseases as type 1 diabetes mellitus, Grave’s disease, Hashimoto’s thyroiditis, Addison’s disease, autoimmune polyglandular syndromes and premature ovarian failure, in this report we revised relevant aspects of MHC molecules, genes, typing methods as well as the new nomenclature.
HLA; MHC; type 1 Diabetes Mellitus; Grave’s disease; Hashimoto’s thyroiditis; Addison’s disease
PERSPECTIVAS
Como entender a associação entre o sistema HLA e as doenças auto-imunes endócrinas
How to understand the association of the HLA system and autoimmune endocrine disorders
Ana Paula M. Fernandes; Léa Maria Z. Maciel; Milton César Foss; Eduardo Antônio Donadi
Departamento de Enfermagem Geral e Especializada (APMF), Escola de Enfermagem de Ribeirão Preto, Universidade de São Paulo; e Departamento de Clínica Médica (LMZM, MCF, EAD), Faculdade de Medicina de Ribeirão Preto, Universidade de São Paulo, Ribeirão Preto, SP
Endereço para correspondência Endereço para correspondência Eduardo Antônio Donadi Professor Associado, Divisão de Imunologia Clínica Departamento de Clínica Médica, Faculdade de Medicina de Ribeirão Preto - USP Av. Bandeirantes 3900 14049-900 Ribeirão Preto, SP Fax: (016) 633-6695 e.mail: eadonadi@fmrp.usp.br
RESUMO
A introdução de nova nomenclatura para os componentes do complexo principal de histocompatibilidade (Major Histocompatibility Complex - MHC) e a descrição de novos métodos moleculares para a tipificação dos alelos do MHC contribuíram, grandemente, para o entendimento e identificação do extenso polimorfismo do sistema. No entanto, para o não especialista, essas novas aquisições têm dificultado o entendimento do papel do MHC em associação com as doenças. Assim, neste artigo, foram revisados os conceitos atuais acerca dos genes e moléculas do MHC, os métodos de tipificação desses marcadores de histocompatibilidade e a nomenclatura vigente para os componentes do sistema. Esses aspectos são extremamente importantes para o entendimento do polimorfismo do MHC, dando embasamento para a compreensão dos mecanismos propostos de associação desses marcadores com as doenças auto-imunes endócrinas como diabetes mellitus do tipo 1, doença de Graves e tireoidite de Hashimoto, doença de Addison, síndrome poliglandular auto-imune e insuficiência ovariana prematura.
Descritores: HLA; MHC; Diabetes mellitus do tipo 1; Doença de Graves; Tireoidite de Hashimoto; Doença de Addison
ABSTRACT
The acquisition of a new nomenclature and the development of molecular methods for the assignment of major histocompatibilty complex (MHC) alleles have widely contributed to the understanding of the extensive polymorphism of the system; however, many doubts have arisen to the non-specialist in terms of MHC and disease association. In order to understand the MHC polymorphism and the association with endocrine diseases as type 1 diabetes mellitus, Graves disease, Hashimotos thyroiditis, Addisons disease, autoimmune polyglandular syndromes and premature ovarian failure, in this report we revised relevant aspects of MHC molecules, genes, typing methods as well as the new nomenclature.
Keywords: HLA; MHC; type 1 Diabetes Mellitus; Graves disease; Hashimotos thyroiditis; Addisons disease
PARA O ENTENDIMENTO DA ASSOCIAÇÃO entre os marcadores de histocompatibilidade e as doenças endocrinológicas, faz-se necessário o conhecimento de alguns aspectos relevantes acerca das moléculas e dos genes de histocompatibilidade.
Os genes de histocompatibilidade são assim chamados porque, quando descobertos, eram os principais elementos (genes principais) relacionados com a rápida rejeição de tecidos transplantados entre animais de experimentação, particularmente entre camundongos, sendo em conjunto denominados Major Histocompatibility Complex (MHC). Assim, o MHC representa o conjunto de genes responsável por codificar as moléculas de histocompatibilidade em uma determinada espécie, sendo chamado no ser humano de sistema HLA (Human Leukocyte Antigen) (1).
O MHC no ser humano está localizado no braço curto do cromossomo 6, e, didaticamente, esses genes podem ser reunidos em 3 grupos, denominados genes de classe I, II e III. Os genes de classe I codificam as moléculas clássicas de histocompatibilidade HLA-A, B e C, os de classe II as moléculas clássicas HLA-DR, DQ e DP. Os genes de classe III, embora estejam incluídos dentro do MHC, não codificam moléculas de histocompatibilidade, e sim outras moléculas, algumas delas fazendo parte do sistema imune outras não. Assim, as proteínas C4 e C2 da via clássica e o fator B da via alternativa do complemento, os fatores de necrose tumoral (TNF-a e TNF-b), a proteína do choque térmico (Hsp 70) e as enzimas 21-hidroxilase são moléculas incluídas na região de classe III. A figura 1 representa de modo esquemático os genes do MHC humano (2).
O MHC é o conjunto de genes mais polimórfico entre os mamíferos. O entendimento do termo polimorfismo é essencial para a compreensão dos mecanismos de associação com as doenças, da evolução das espécies, e, também, para a seleção de doadores em transplantes de órgãos ou células. Alguns genes apresentam uma mesma seqüência de ácidos nucléicos em todos os membros de uma população, sendo chamados não polimórficos (exemplo: b2-microglobulina), e cada variante de um gene polimórfico é denominada de alelo. Exemplificando, as moléculas codificadas por um mesmo gene presente no locus HLA-B apresentam muitas variantes, como HLA-B7, -B8, -B27, -B51, entre outras, encontradas na população em diversas freqüências. Uma vez que os genes de histocompatibilidade são codominantes, tanto os genes de origem materna como os paternos serão expressos. Assim, para os genes HLA de classe I, até 6 moléculas podem ser expressas nas superfícies celulares, ou seja, 2 (duas) HLA-A, 2 (duas) HLA-B e 2 (duas) HLA-C, totalizando 3 moléculas de origem materna e 3 de origem paterna. Pelo fato desses genes estarem muito próximos uns dos outros, são herdados em bloco. O conjunto de genes oriundos do cromossomo materno ou paterno é designado de haplotipo.
AS MOLÉCULAS E OS GENES DO MHC
As moléculas de classe I estão constitutivamente expressas nas superfícies de todas as células nucleadas, sendo compostas por 2 cadeias diferentes (heterodímeros). A cadeia pesada a, polimórfica, é codificada pelos loci HLA-A, -B, ou -C do MHC, situados no cromossomo 6, e a cadeia leve, a b2-microglobulina, não polimórfica, é codificada por gene situado fora do MHC, no cromossomo 15. A cadeia pesada a contém 3 domínios: a1, a2 e a3, sendo que o grande polimorfismo dessas moléculas ocorre nos domínios a1 e a2 (figura 2). Esses domínios são extracelulares e contêm cerca de 90 resíduos de aminoácidos cada. Os peptídeos que vão se ligar às moléculas de classe I são gerados no citosol, pela ação de algumas proteínas chamadas proteassomas (genes LPM 2 e 7, situados no MHC) e transportados ao retículo endoplasmático pelas proteínas TAP 1 e 2 (genes também situados no MHC). Uma vez formado, o peptídeo com cerca de 8 a 12 aminoácidos se encaixa no sulco de ligação ao peptídeo da molécula HLA de classe I ainda no retículo endoplasmático e, então, a molécula HLA ligada ao peptídeo é transportada até a membrana da célula, sendo apresentada ao linfócito T CD8 (4-6).
As moléculas HLA de classe II estão presentes constitutivamente apenas nas superfícies de alguns tipos celulares como macrófagos, linfócitos B e células dendríticas, coletivamente denominadas de células apresentadoras de antígenos. Essas moléculas também são heterodímeros formados por uma cadeia a e uma b, ambas codificadas por genes situados no MHC, ou seja, no cromossomo 6, contendo 2 domínios a1/a2 e b1/b2. Os domínios que apresentam maior polimorfismo são o a1 e o b1 (cada domínio contém cerca de 90 resíduos de aminoácidos) para as moléculas HLA-DQ e DP. Em contraste, para as moléculas HLA-DR, o polimorfismo ocorre no domínio b1 (figura 2).
Os peptídeos apresentados pelas moléculas HLA de classe II têm cerca de 15 a 24 aminoácidos e são processados nos compartimentos endolisossomais, sendo que as moléculas HLA-DM (presentes somente no interior da célula) permitem que o peptídeo se una ao sulco de ligação ao peptídeo da molécula de classe II. As moléculas contendo os peptídeos tornam-se estáveis, sendo então transportadas até as superfícies celulares, facilitando, assim, a apresentação do peptídeo ao receptor do linfócito T CD4 (8).
O sulco de ligação ao peptídeo da molécula de classe I é formado pela interação dos segmentos a1 e a2, ao passo que o da molécula de classe II, pela interação dos segmentos a1 e b1. Os resíduos de aminoácidos polimórficos que caracterizam as diversas moléculas de histocompatibilidade estão localizados nessas fendas de ligação com o peptídeo. Por causa da grande variabilidade de aminoácidos nessa região, moléculas HLA diferentes se ligam e apresentam peptídeos diferentes, sendo reconhecidos por receptores de linfócitos T também diferentes. Essa observação explica, em parte, porque algumas pessoas quando entram em contato com antígenos estranhos, derivados de bactérias, vírus etc., as respostas imunes podem ser diferentes (8,9). A tabela 1 ilustra as propriedades das moléculas MHC de classe I e II (7,4,5).
Uma vez que cada indivíduo possui um número limitado de moléculas HLA nas superfícies de suas células, é correto supor que uma mesma molécula possa acomodar e apresentar vários tipos de peptídeos. Assim, as moléculas HLA apresentam uma ampla capacidade de se ligar a peptídeos; no entanto, a especificidade fina de reconhecimento do antígeno ocorre às custas do receptor do linfócito T. As moléculas de histocompatibilidade, para serem estáveis nas superfícies celulares, devem sempre estar ligadas aos peptídeos e, usualmente, não discriminam os peptídeos estranhos (derivados de agentes patogênicos) daqueles que são do próprio indivíduo, quem faz o reconhecimento do que é próprio ou não, é o linfócito T (6).
MÉTODOS DE DETECÇÃO DO POLIMORFISMO DOS ANTÍGENOS E DOS ALELOS DE HISTOCOMPATIBILIDADE
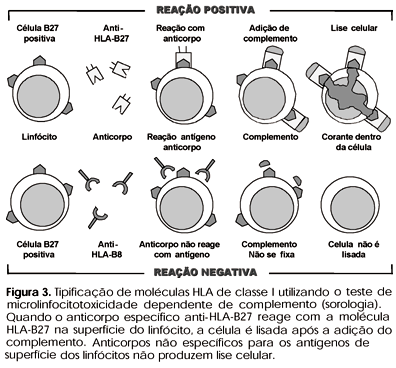
O método clássico de avaliação do polimorfismo das moléculas (antígenos) de histocompatibilidade é o de citotoxicidade celular mediada por anticorpo e dependente do complemento, também chamado método sorológico (figura 3). Esse método é baseado na reação de um anticorpo anti-HLA (geralmente obtido de indivíduos politransfundidos ou multíparas, ou ainda, anticorpo monoclonal obtido de hibridoma) com a molécula HLA presente na superfície celular. Após ocorrer a reação antígeno-anticorpo, a incubação com o complemento promove a lise celular, dando uma reação positiva. Posto que as moléculas HLA de classe I estão expressas constitutivamente nas células nucleadas, e que as células linfomononucleares são de fácil recuperação à partir do sangue periférico, os antígenos HLA-A, B e C são tipificados utilizando-se os linfócitos totais ou os linfócitos T. Uma vez que os linfócitos T possuem poucas moléculas HLA de classe II nas suas superfícies, os linfócitos B são utilizados para tipificar os antígenos HLA-DR e DQ. Os antígenos HLA-DP não são tipificados por esse método devido à falta de anti-soros específicos. A nomenclatura dos antígenos HLA definidos por sorologia utiliza a designação do locus gênico (HLA-A, B, C, DR ou DQ), seguida pela identificação numérica do antígeno (HLA-A1, A2 etc; HLA-B7, B8, B27, B51 etc; HLA-DR1, DR3, DR4 etc e HLA-DQ2, DQ4 etc). A nomenclatura do locus HLA-C incorpora a letra w (HLA-Cw1, Cw2 etc) para diferenciá-la da nomenclatura dos fatores do complemento (C1, C2, C3 etc). Embora o método sorológico ainda seja utilizado, existe uma tendência de substituí-lo pelos métodos moleculares, descritos mais adiante (10-13).
O grande avanço na tipificação do polimorfismo HLA, indubitavelmente, ocorreu no final da década de 80 com o advento das técnicas que utilizam biologia molecular. Nesses casos, não são os antígenos expressos nas superfícies celulares que são tipificados, e sim, os grupos de alelos ou os alelos individualmente do DNA genômico. Para tal, o DNA é extraído das células nucleadas do sangue periférico ou de outra fonte, como células de bochecha, cabelo, ossos etc. O DNA é amplificado pela reação em cadeia da polimerase (polymerase chain reaction-PCR) utilizando oligonucleotídeos específicos para amplificar a região do genoma a ser estudada. Dois métodos têm sido bastante utilizados para tipificação dos alelos HLA de classe I ou II, utilizando iniciadores (primers) ou sondas (probes) de oligonucleotídeos com seqüências conhecidas, sendo denominados sequence specific primers (SSP) ou sequence specific oligonucleotide probes (SSOP). No método SSP são realizadas várias reações de amplificação, cada uma contendo um par de iniciadores capaz de detectar um grupo de alelos ou um alelo. Os produtos de amplificação são submetidos a uma eletroforese em gel de agarose, contendo brometo de etídeo, uma substância fluorescente capaz de se intercalar no DNA, tornando fluorescente os produtos de amplificação quando o gel é submetido à luz ultravioleta. As bandas com cerca de 150-220 pares de bases correspondem aos produtos de amplificação específicos dos genes HLA, enquanto que as bandas com cerca de 800 pares de bases correspondem aos produtos de amplificação interna, isto é, servem para o controle de que a amplificação realmente ocorreu (figura 4). No método SSOP, utiliza-se um par de iniciadores construídos para amplificar uma região genérica de um gene (por exemplo HLA-DR). Em seguida, o DNA amplificado é hibridado com sondas de oligonucleotídeos capazes de reconhecer os diversos grupos de alelos do gene. Nesse método, o DNA amplificado é desnaturado e fixado em membranas de náilon. As sondas marcadas, com material radioativo ou substância fluorescente, são hibridadas aos DNA fixados nas membranas quando incubadas em temperaturas adequadas (figura 5). Um outro método que pode vir a ser muito popular em um futuro próximo é o sequenciamento automático das bases nitrogenadas. A identificação dos alelos é realizada por intermédio da comparação com um banco de dados, referente às seqüências já conhecidas. Assim, com a utilização dos métodos moleculares, foi possível reconhecer que moléculas tipificadas por sorologia, como por exemplo HLA-A2, eram codificadas por gene apresentando cerca de 50 alelos diferentes. Isto é, existem cerca de 50 moléculas HLA-A2 diferentes que a sorologia não consegue discriminar, pois o anticorpo anti-HLA-A2 reconhece um epítopo comum a todas essas moléculas. Para englobar a discriminação do extenso polimorfismo definido pelos métodos moleculares, a nomenclatura do sistema HLA necessitou passar por uma ampla revisão, como sumariado a seguir (10,14).
NOMENCLATURA ATUAL DOS ALELOS DE HISTOCOMPATIBILIDADE
O Comitê Internacional para normatização da denominação dos alelos de histocompatibilidade decidiu racionalizar a nomenclatura do sistema HLA, aumentando o número de dígitos e definindo a cadeia onde o polimorfismo é mais acentuado. As cadeias que formam os heterodímeros das moléculas de histocompatibilidade são designadas pelas letras gregas a ou b, ao passo que os genes que codificam essas cadeias são designados pelas letras A ou B. Uma vez que os loci de classe I apresentam polimorfismo apenas na cadeia a (gene A), o Comitê decidiu manter as designações HLA-A, HLA-B e HLA-Cw como descritas anteriormente. No entanto, aumentou o número de dígitos para 4, com a finalidade de abranger todo o polimorfismo já identificado e, ainda, aquele a ser definido no futuro. Os 2 dígitos iniciais, colocados logo após a designação do locus, referem-se às tipificações definidas previamente por sorologia, e os 2 dígitos seguintes referem-se às denominações dos alelos específicos. Assim, o grupo de alelos que codifica o antígeno HLA-A1 passou a ser denominado genericamente de HLA-A*01, contendo os alelos HLA-A*0101, *0102 e *0103. O grupo de alelos que codifica o antígeno HLA-B27 passou a ser chamado HLA-B*27, englobando os alelos HLA-B*2701 até B*2723, identificando pelo menos 23 variantes da molécula B27, e assim por diante (15,16).
Uma vez que os alelos de classe II apresentam polimorfismo tanto nas cadeias a e b, foram acrescentadas as letras A e B na denominação do alelo. Da mesma forma que, para os alelos de classe I, os 2 dígitos iniciais indicam as especificidades prévias definidas pela sorologia, ao passo que as 2 últimas identificam o alelo. Assim, o gene que codifica a molécula HLA-DR1 passou a ser denominado genericamente de HLA-DRB1*01 (equivalente ao DR1 da sorologia), abrangendo os alelos DRB1* 0101 *0102 e *0103, sendo a letra B representativa da cadeia b, altamente polimórfica nos genes HLA-DR. Posto que a região HLA-DR possui diversos genes para as cadeias b (ver figura 1), cada locus recebe o número correspondente ao gene, como HLA-DRB1*1101 (equivalente ao DR11 da sorologia). A tabela 2 sumaria os loci de histocompatibilidade descritos até a última nomenclatura, bem como os números de alelos em cada loci, identificados até o presente (15,16).
COMO IDENTIFICAR A ASSOCIAÇÃO DE MARCADORES DE HISTOCOMPATIBILIDADE COM AS DOENÇAS
Dois tipos de estudos têm sido utilizados para abordar a associação entre os marcadores de histocompatibilidade com as doenças, ou seja, os estudos populacionais e os familiares, sendo que os populacionais são mais fáceis de serem realizados. Nos estudos populacionais, as freqüências dos antígenos ou dos alelos HLA, observadas em um grupo de pacientes não aparentados, são comparadas com aquelas observadas em indivíduos controle sadios. A ocorrência de associação é avaliada pela comparação das freqüências dos marcadores de histocompatibilidade em pacientes e controles, utilizando-se os dados de uma tabela de contingência 2x2 e empregando-se o teste exato de Fisher ou o Chi-quadrado. A força da associação é usualmente calculada pelo risco relativo (RR), que indica quão mais freqüente uma doença ocorre em indivíduos portadores de um marcador de histocompatibilidade em relação aos indivíduos que não apresentam o marcador. Nos casos em que o RR é maior do que 1, a freqüência do marcador nos pacientes é maior do que nos controles, indicando que o marcador está associado com susceptibilidade à doença. Quando o RR é menor do que 1, tem significado inverso, isto é, o marcador está associado com proteção contra o desenvolvimento da doença. Nos casos em que o RR é maior do que 1, pode-se calcular a fração etiológica (FE) que estima a magnitude de susceptibilidade ao marcador, em termos populacionais, isto é, quanto (usualmente definido em termos percentuais) o marcador confere susceptibilidade à doença. Nos casos em que o RR é menor do que 1, calcula-se a fração preventiva (FP), indicando a magnitude de proteção do marcador, também definido como valor percentual (17-20).
HLA E AS DOENÇAS AUTO-IMUNES ENDÓCRINAS
Doenças auto-imunes endócrinas, coletivamente, afetam entre 5-10% da população em geral. A maioria dessas doenças possui mecanismos complexos com a participação de vários fatores, iniciando com a susceptibilidade genética principalmente pela associação dos genes HLA, eventos ambientais e resposta auto-imune com a presença de auto-anticorpos e/ou linfócitos auto-reativos, culminando com anormalidades metabólicas (21).
Muitos estudos acerca das associações entre os alelos HLA e as endocrinopatias auto-imunes se concentram no diabetes mellitus tipo 1 e doença de Graves, mas outras endocrinopatias também têm sido estudadas, incluindo a tireoidite de Hashimoto, a doença de Addison, a síndrome poliglandular auto-imune e a insuficiência ovariana prematura.
DIABETES MELLITUS DO TIPO 1
A DM-1 tem sido considerada uma doença com herança poligênica complexa. Cerca de 20 genes podem estar associados com susceptibilidade à doença, mas apenas 13 apresentam evidências estatisticamente significantes de associação. A maior contribuição vem da região onde estão localizados os genes do MHC, situado no cromossomo 6p21, contribuindo em cerca de 40% na susceptibilidade à doença (genes IDDM1). Contribuições de menor valor são conferidas por genes de fora do MHC (22).
Os primeiros estudos acerca da susceptibilidade imunogenética ao DM-1 datam da década de 70, sendo encontradas associações com as moléculas HLA-B8 e B15. Posteriormente, foi verificado que as associações mais importantes eram com as moléculas de classe II HLA-DR3 e HLA-DR4, em desequilíbrio de ligação com os antígenos HLA-B8 e B15, respectivamente. Subseqüentemente, os genes DQA1*0301 e *0501 e os DQB1*0201 e *0302 se revelaram os mais fortemente associados à doença (23). Atualmente, tem sido sugerido que ambos genes DR e DQ influenciam na susceptibilidade ou proteção à DM-1, promovendo efeitos sinérgicos ou antagonistas de acordo com o haplotipo (24,25).
Em caucasianos, os indivíduos heterozigotos HLA-DR3/DR4 apresentam RR mais elevado para o desenvolvimento da doença. Em contraste, o antígeno HLA-DR2 e mais fracamente o HLA-DR5 são raramente encontrados entre os pacientes com DM-1, estando associados com resistência ao desenvolvimento da doença (26). Em japoneses, os antígenos HLA-DR4 e HLA-DR9 são os mais freqüentes (27,28). No final da década de 80, foi verificado que os haplotipos HLA-DR/DQ que conferiam susceptibilidade à doença tinham características peculiares, sendo que o haplotipo HLA-DRB1*04/DQB1*0302 estava associado ao desenvolvimento da doença e o HLA-DRB1* 04/DQB1*0301 não estava (23). A diferença entre os alelos HLA-DQB1*0302 e o DQB1*0301 está localizada em uma seqüência de nucleotídeos, correspondente ao primeiro domínio polimórfico da molécula DQb, onde existem apenas quatro códons responsáveis pela síntese de aminoácidos presentes no sulco de ligação da molécula HLA. Um desses códons, localizado na posição 57 nos alelos de resistência, apresenta o resíduo ácido aspártico. Nos alelos de susceptibilidade, esse aminoácido não é encontrado na posição 57, sendo sugerido que a presença de um resíduo diferente dele nessa posição estaria correlacionada com susceptibilidade à doença. No entanto, em japoneses, a molécula HLA-DQ que confere susceptibilidade a diabetes apresenta ácido aspártico na posição 57 (29). Estudos utilizando embriões de camundongos NOD mostram que a introdução de genes I-A a/b ou Ab, que codificam o ácido aspártico na posição 57 da cadeia b da molécula de classe II, diminuem ou impedem o desenvolvimento da doença nesses camundongos transgênicos (30). Atualmente, parece claro que a susceptibilidade à doença é muito complexa e envolve a combinação de diferentes alelos de classe II, promovendo efeitos antagônicos ou sinérgicos. A figura 6 ilustra essas combinações e seus efeitos na susceptibilidade ou resistência ao DM-1.
Em caucasianos, a susceptibilidade ao DM-1 está associada com os haplotipos HLA-DRB1*03-DQA1*0501-DQB1*0201 e HLA-DRB1*04-DQA1 *0301-DQB1*0302, enquanto que o haplotipo HLA-DRB1*1501-DQA1*0102-DQB1*0602 está fortemente associado com proteção contra o desenvolvimento da doença (31). Em estudos realizados em populações altamente miscigenadas, como os pacientes brasileiros com DM-1 da região Nordeste do Estado de São Paulo, as especificidades HLA-DR3 DR4 e HLA DQB1*0302 também conferem susceptibilidade ao desenvolvimento da doença (32,33).
DOENÇA DE GRAVES
A Doença de Graves (DG) é uma doença multifatorial causada pela interposição complexa de fatores genéticos, ambientais e constitucionais, levando a perda da tolerância a antígenos tireoidianos e a iniciação de uma reação auto-imune mantida dirigida à tireóide e a antígenos orbitais, ainda não identificados.
A susceptibilidade genética é sugerida pela observação da incidência familiar da doença. Entretanto, a concordância da DG em gêmeos idênticos varia de 30 a 50% (34), sendo, porém, bem maior que em gêmeos dizigóticos, que se mostra em torno de 5%. A conclusão destes estudos sugere que ambos, genes e o ambiente, contribuem para o desenvolvimento da doença.
Estudos populacionais têm demonstrado diversas associações entre DG e o polimorfismo dos genes de MHC. Associações consistentes têm sido demonstradas com genes de classe II, especialmente DR3 em populações caucasianas, procedentes da Europa, Austrália e Estados Unidos.
Haplotipos diversos parecem estar envolvidos em diferentes grupos étnicos: HLA-DQA1*0501 (35), HLA-A2 e HLA-DPB1*0501 em japoneses (36), HLA-B46, HLA-DR9 e HLD-DRB1*0303 em chineses (37), HLA-A10, HLA-B8, HLA-DQ2 em indianos (38) e HLA-A10, HLA-DR1 em sulafricanos (39). Na população brasileira, os alelos HLA-DRB1*0301 e DQA1*0501 mostram maior envolvimento, conferindo susceptibilidade à doença, com RR de 2,8 e 3,74, respectivamente (40).
Estudo recente (41) demonstrou que, quando a transmissão do gene de alto risco, HLA-DQA* 0501DQB*0201, vem de origem paterna, há maior susceptibilidade em pacientes jovens de desenvolverem a doença tireoidiana auto-imune (Graves ou Hashimoto).
Em geral, as associações com HLA têm demonstrado RR baixos, mesmo com alelos que apresentam alta prevalência na população geral; contudo, diversos estudos sugerem que a região HLA contribui com cerca de 20% para susceptibilidade genética à DG (42,43). Estes achados indicam que o locus HLA, apesar de representar um fator de risco estabelecido, explica somente uma pequena fração da predisposição genética total e que possivelmente outros genes estão envolvidos.
A pesquisa de outros determinantes antigênicos na DG envolve também loci diferentes do HLA. Têm sido investigados os genes envolvidos na resposta imune, como receptores a e b da célula T (44), antagonista do receptor da IL-1 (45), receptor-2 do fator de necrose tumoral (37,46), o proteassoma multifuncional do MHC de classe I (47). Mais recentemente, o polimorfismo do antígeno-4 do linfócito T (CTLA-4) tem sido associado com a DG em caucasianos e japoneses (42). Os estudos de outros genes, com potenciais antigênicos, como os auto antígenos tireoidianos (peroxidase, a tireoglobulina e o receptor de TSH) foram excluídos por estudos da associação e de análise de ligação (48-51). Com o projeto do Genoma Humano, outros genes foram identificados como potencialmente envolvidos na susceptibilidade à doença tireoidiana. Eles estão localizados no cromossoma 14q31 (6D-1), no cromossoma 20 (6D-2) e no cromossoma X, porém estes resultados ainda necessitam confirmação (52-55).
TIREOIDITE DE HASHIMOTO
A Tireoidite de Hashimoto (TH) é um distúrbio autoimune decorrente de resposta imune anormal à glândula tireóide, tanto do ponto de vista humoral como celular. Assim como na DG, a demonstração de que parentes de casos índices têm maior probabilidade para desenvolverem a doença indica a existência de predisposição genética. Jenkins e cols. (56) encontraram evidências para a associação entre TH e HLA-DR3 e -DR4 com RR de 2,3 e 1,6, respectivamente, em caucasianos. Outros estudos também indicam uma associação fraca e heterogênea entre HLA-DR3, -DR4 e -DR5 no hipotireoidismo auto-imune, seja ele com bócio ou não, o que reflete uma maior heterogeneidade das tireoidites auto-imunes ou uma diferença real entre os genes de susceptibilidade à doença, quando comparados com a DG (57-59).
Estudos envolvendo outros loci mostram fraca associação com a imunoglobulina de cadeia pesada, tanto em japoneses (60) como em caucasianos (61). De maneira similar, polimorfismo para o receptor b da célula T foi encontrado com freqüência maior em pacientes com TH (62). Entretanto, em outra investigação, somente houve associação com a cadeia a do receptor da célula T e não com a cadeia b (63). Outro importante gene regulatório estudado é o CTLA-4. Esta proteína é expressa nas células T ativadas e compete com o CD28 para os receptores co-estimuladores B7-1 e B7-2. Ligação preferencial do B7 ao CTLA-4 induz regulação negativa e silenciamento da célula T ativada. Funcionamento anômalo deste fator poderia estar envolvido na auto-imunidade, como tem sido demonstrado no camundongo deficiente em CTLA-4, que desenvolve infiltração linfocitária generalizada em muitos órgãos (64). Com os relatos do envolvimento do CTLA-4 com a DG (65), 2 polimorfismos deste gene foram estudados. O mecanismo de sua potencial participação na patogênese do TH é desconhecido.
DOENÇA DE ADDISON
Doença de Addison (DA), caracterizada pela destruição do córtex adrenal e conseqüente insuficiência adrenocortical primária, está associada com os alelos DR3 (21,66). Outros marcadores HLA também tem sido identificados, apresentando uma freqüência aumentada e positivamente correlacionados com os haplotipos HLA-DRB1*03-DQA1*05-DQB1*02 (DR3-DQ2) e DRB1*04-DQA1*03-DQB1*0302 (DR4-DQ8). Por outro lado, foi identificado o haplotipo DRB1*01-DQA1*0101-DQB1*0501 (DR1-DQ5), negativamente associado com a doença (67).
SÍNDROME POLIGLANDULAR AUTO-IMUNE
Síndrome poliglandular auto-imune (Autoimmune Polyglandular Syndrome - APS) foi inicialmente definida como insuficiência múltipla das glândulas endócrinas associada a uma doença auto-imune. Neufeld e cols. (68) sugeriram uma classificação de 4 tipos principais de APS, baseado apenas em critérios clínicos. APS-1 é caracterizado pela presença de candidíase crônica, hipoparatireoidismo crônico e DA, APS-2 freqüentemente apresenta a DA, doenças auto-imunes tireoidianas e/ou DM-1. Doenças auto-imunes tireoidianas, exceto DA e/ou hipoparatireoidismo, são as principais características da APS-3 e, finalmente, diferentes combinações clínicas de doenças auto-imunes, não incluídas nas classificações anteriores, estão presentes na APS-4 (69).
Estudos demonstram que APS do tipo 2 e 3 estão associadas com os alelos HLA DR3/DR4. Por outro lado, o alelo HLA-DQA*0102 parece ser o marcador para a resistência às doenças auto-imunes tireoidianas. Estudo recente mostrou que pacientes com APS do tipo 2 apresentaram freqüências aumentadas dos alelos HLA-DQA1*0301 e -DQA1*0501, enquanto que HLA-DQA1*0301 está associado com APS tipo 3 (70).
Genes do HLA parecem modificar a expressão clínica da síndrome, assim, HLA-A28 é mais freqüente entre pacientes com hipoparatireoidismo e DM-1 e HLA-A3 na insuficiência ovariana (71). Outros estudos apontam para a importância do HLA-DR3 e -DQB1*0201 na APS tipo 2 e, HLA-DR4, -DQB1*0302 quando associada com DM-1 (72).
INSUFICIÊNCIA OVARIANA PREMATURA
Definida pelo início da menopausa antes dos 40 anos, afeta cerca de 1% das mulheres, ocorrendo em um terço das mulheres com doenças auto-imunes, tireoidianas ou não (73). Estudo canadense verificou a associação com HLA-DR3 (74), porém outros não obtiveram dados similares (75,76). Investigações serão necessárias para definir a associação com os genes HLA.
CONCLUSÃO
Como observado, a maioria das doenças endócrinas auto-imunes apresentam marcadores HLA de susceptibilidade semelhantes nas populações caucasianas, usualmente incluindo os grupos de alelos HLA-DRB1*03/*04 e DQB1*02/0302, sendo que, para o DM-1, os RRs conferidos por esses grupos de alelos são mais elevados do que os conferidos por esses alelos nas outras doenças endócrinas auto-imunes. Esses achados indicam que, embora os antígenos e alelos de histocompatibilidade estejam envolvidos na patogenia dessas endocrinopatias, outros fatores genéticos, alguns já identificados, também estão envolvidos. Futuros progressos possivelmente acontecerão com os estudos familiares com múltiplos membros acometidos, em andamento em diversos centros, vinculando o uso de marcadores polimórficos em todo o genoma para identificar loci de susceptibilidade até agora não suspeitados.
Recebido em 11/11/02
Revisado em 21/07/03
Aceito em 01/08/03
- 1. Ellis SA, Ballingall KT. Cattle MHC: evolution in action? Immunol Rev 1999;167:159-68.
- 2. Nepom GT, Erlich H. MHC class II molecules and autoimmunity. Annu Rev Immunol 1991;9:493-525.
- 3. Donadi EA. Aspectos moleculares do complexo principal de histocompatibilidade: como entender a associação entre o sistema HLA e as doenças reumáticas. Rev Bras Reumatol 2001;41:225-36.
- 4. Eren E, Travers P. The structure of the major histocompatibilty complex and its molecular interactions. In: R Lechler & A Warrens editors. HLA in Health and Disease. 2nd ed. San Diego: Academic Press; 2000 p. 23-33.
- 5. Mackay I, Rosen FS. The HLA system: Second of two parts. N Engl J Med 2000;343:782-6.
- 6. Milner CN, Campbell D, Trowsdale J. Molecular genetics of the major histocompatibility complex. In: Lechler R, Warrens A, eds. HLA in health and disease. 2nd ed. San Diego: Academic Press; 2000 p.51-72.
- 7. Mackay I, Rosen FS. The HLA system: First of two parts. N Engl J Med 2000;343:702-9.
- 8. McDevitt HO. Discovering the role of the major histocompatibility complex in the immune response. Annu Rev Immunol 2000;18:1-17.
- 9. Louzada-Júnior P, Smith AG, Hansen JA, Donadi EA. HLA-DRB1 and -DQB1 alleles in the Brazilian population of the northeastern region of the state of São Paulo. Tissue Antigens 2001;57:158-62.
- 10. Bignon JD, Fernández-Viña MA, Cheneau ML, Fauchet R, Schreuder GMTh, Clayton J, et al. HLA DNA class II typing by PCR-SSOP: 12th International Histocompatibility Workshop experience. In: Charron D, editor. Genetic diversity of HLA: functional and medical implication. Paris: EDK Medical and Scientifical Publisher; 1997 p.26-8.
- 11. Colombe BW. Histocompatibility testing. In: Stites DP, Terr AI, Parslow TG, eds. Medical immunology. 9th ed. Stanford: Appleton & Lange; 1997 p.286-308.
- 12. Dyer PA, Martin S, Stanford RE. Serological methods in HLA typing. In: Lechler R, Warrens A, eds. HLA in health and disease. 2nd ed. San Diego: Academic Press; 2000 p.429-40.
- 13. Erlich HA. Polymerase chain reaction-based methods of HLA typing. In: Lechler R, Warrens A, eds. HLA in health and disease. 2nd ed. San Diego: Academic Press; 2000 p.451-61.
- 14. Mason PD, Lechler RI. Cellular methods in testing histocompatibility. In: In: Lechler R, Warrens A, eds. HLA in health and disease. 2nd ed. San Diego: Academic Press; 2000 p.441-50.
- 15. Bodmer JG, Bodmer WF, Marsh SGE. HLA nomenclature. In: Lechler R, Warrens A, eds. HLA in health and disease. 2nd ed. San Diego: Academic Press; 2000 p.147-61.
- 16. Marsh SGE, Bodmer JG, Albert ED, Bodmer WF, Bontrop RE, Dupont B, et al. Nomenclature for factors of the HLA system, 2000. Human Immunol 2001;62:418-68.
- 17. Dyer P, Warrens NA. Design and interpretation of studies of the major histocompatibility complex in disease. In: Lechler R, editor. HLA and disease. 1st ed. London: Academic Press; 1994 p.93-121.
- 18. Svejgaard A. HLA and disease. In: Rose MR, Friedman H, Fahey JC, eds. Manual of clinical and laboratory immunology. Washington: American Society for Microbiology; 1986 p.912-20.
- 19. Vyse TJ, Morley BJ. The analysis of genetic susceptibility. In: Lechler R, Warrens A, eds. HLA in health and disease. 2nd ed. San Diego: Academic Press; 2000 p.107-28.
- 20. Warrens NA. Appendix: Statistical considerations in analyzing HLA and disease associations. In: Lechler R, Warrens A, eds. HLA in health and disease. 2nd ed. San Diego: Academic Press; 2000 p.129-37.
- 21. Tait KF, Gough SC. The genetics of autoimmune endocrine disease. Clin Endocrinol 2003;59:1-11.
- 22. Owerbach D, Naya FJ, Tsai MJ, Allander SV, Powell DR, Gabbay HG. Analysis of candidate for susceptibility to type 1 diabetes. Diabetes 1997;46:1069-74.
- 23. Nepom GT, Kwok WW. Molecular basis for HLA-DQ associations with IDDM. Diabetes 1998;47:1177-84.
- 24. She JX. Susceptibility to type I diabetes: HLA-DQ and DR revised. Immunology Today 1996;17:323-9.
- 25. She JX, Marron MP. Genetic susceptibility factors in type 1 diabetes: linkage, disequilibrium and functional analyses. Curr Opi Immun 1998;10:682-8.
- 26. Nepom GT. A unified hypothesis for the complex genetics of HLA associations with IDDM. Diabetes 1990;39:1153-7.
- 27. Ronningen KS, Spurkland A, Tait BD, Drummond B, Lopez-Larrea C, Baranda FS, et al. HLA class II associations in insulin-dependent diabetes mellitus among blacks, Caucasoid and Japanese. Disease component 1995;W6.4:713-22.
- 28. Thorsby E, Roningen KS. Particular HLA-DQ molecules play a dominant role in determining susceptibility or resistance to type 1 (insulin-dependent) diabetes mellitus. Diabetologia 1993;36:371-7.
- 29. Nepom GT. Class II antigens and disease susceptibility. Annu Rev Med 1995;46:17-25.
- 30. Quartey-Papafio R, Lund T, Chandler P, Picard J, Ozegbe P, Day S, et al. Aspartate at position 57 of nonobese diabetic I-Ag7 beta-chain diminishes the spontaneous incidence of insulin-dependent diabetes mellitus. J Immunol 1995;154:5567-75.
- 31. Boitard C, Caillat-Zucman S, Timsit J. Insulin-dependent diabetes and human leukocyte antigens. Diabetes Metab 1997;23:22-8.
- 32. Eizirik DL, Monteiro CMC, Voltarelli JC, Foss MC. Frequency of HLA antigens in a Brazilian type I diabetic population. Braz J Med Biol Res 1987;20:533-7.
- 33. Fernandes APM, Louzada-Junior P, Donadi, EA. HLA-DRB1, DQB1 and DQA1 allele profile in Brazilian patients presenting with type 1 diabetes mellitus. Ann NY Acad Sci 2002;958:305-8.
- 34. Brix TH, Kyvik KD, Hegedus L. What is the evidence of genetic factors in the etiology of Graves disease? A brief review. Thyroid 1998;8:727-31.
- 35. Yanagawa T, Mangklabruks A, DeGroot LJ. Strong association between HLA-DQA1*0501 and Graves disease in a male Caucasian population. J Clin Endocrinol Metab 1994;79:227-9.
- 36. Dong RP, Kimura A, Okubo R, Shinagawa H, Tamai H, Nishimura Y, et al. HLA-A and DPB1 loci confer susceptibility to Graves disease. Hum Immunol 1992;35:165-72.
- 37. Cavan DA, Penny MA, Jacobs KH, Jelly MA, Jenkins D, Mijovic CH, et al. Analysis of a Chinese population suggests that the TNFB gene is not a susceptibility gene for Graves disease. Hum Immunol 1994;40:135-7.
- 38. Tandon N, Mehra NK, Taneja V, Vaidya MC, Kochupillai N. HLA antigens in Asian Indian patients with Graves disease. Clin Endocrinol 1990;33:21-6.
- 39. Omar MA, Hammond MG, Desai RK, Motala AA, Aboo N, Seedat MA. HLA class I and II antigens in South African blacks with Graves disease. Clin Immunol Immunopathol 1990;54:98-102.
- 40. Maciel LM, Rodrigues SS, Dibbern RS, Navarro PA, Donadi EA. Association of the HLA-DRB1*0301 and HLA-DQA1*0501 alleles with Graves disease in a population representing the gene contribution from several ethnic backgrounds. Thyroid 2001;11:31-5.
- 41. Segni M, Pani MA, Pasquino AM, Badenhoop K. Familial clustering of juvenile thyroid autoimmunity: higher risk is conferred by human leukocyte antigen DR3-DQ2 and thyroid peroxidase antibody status in fathers. J Clin Endocrinol Metab 2002;87:3779-82.
- 42. Vaidya B, Imrie H, Perros P, Dickinson J, McCarthy MI, Kendall-Taylor P, et al. Cytotoxic T lymphocyte antigen-4 (CTLA-4) gene polymorphism confers susceptibility to thyroid associated orbitopathy. Lancet 1999;354:743-4.
- 43. Roman SH, Greenberg D, Rubinstein P, Wallenstein S, Davies TF. Genetics of autoimmune thyroid disease: lack of evidence for linkage to HLA within families. J Clin Endocrinol Metab 1992;74:496-503.
- 44. Davies TF, Kendler DL. Mechanisms of human autoimmune thyroid disease 1992. Monogr Pathol 1993;35:103-17.
- 45. Blakemore AI, Watson PF, Weetman AP, Duff GW. Association of Graves disease with an allele of the interleukin-1 receptor antagonist gene. J Clin Endocrinol Metab 1995;80:111-5.
- 46. Badenhoop K, Schwarz G, Schleusener H, Weetman AP, Recks S, Peters H, et al. Tumor necrosis factor beta gene polymorphisms in Graves disease. J Clin Endocrinol Metab 1992;74:287-91.
- 47. Heward JM, Allahabadia A, Sheppard MC, Barnett AH, Franklyn JA, Gough SC. Association of the large multifunctional proteasome (LMP2) gene with Graves disease is a result of linkage disequilibrium with the HLA haplotype DRB1*0304-DQB1*02-DQA1*0501. Clin Endocrinol 1999;51:115-8.
- 48. Mangklabruks A, Cox N, DeGroot LJ. Genetic factors in autoimmune thyroid disease analyzed by restriction fragment length polymorphisms of candidate genes. J Clin Endocrinol Metab 1991;73:236-44.
- 49. Cuddihy RM, Schaid DS, Bahn RS. Multivariate analysis of HLA loci in conjunction with a thyrotropin receptor codon 52 polymorphism in conferring risk of Graves disease. Thyroid 1996;6:261-5.
- 50. Roux N, Shields DC, Misrahi M, Ratanachaiyavong S, McGregor AM, Milgrom E. Analysis of the thyrotropin receptor as a candidate gene in familial Graves disease. J Clin Endocrinol Metab 1996;81:3483-6.
- 51. Tomer Y, Davies TF. The genetic susceptibility to Graves disease. Baillieres Clin Endocrinol Metab 1997;11:431-50.
- 52. Barbesino G, Tomer Y, Concepcion ES, Davies TF, Greenberg DA. Linkage analysis of candidate genes in autoimmune thyroid disease. II. Selected gender-related genes and the X-chromosome. International Consortium for the Genetics of Autoimmune Thyroid Disease. J Clin Endocrinol Metab 1998;83:3290-5.
- 53. Barbesino G, Tomer Y, Concepcion E, Davies TF, Greenberg DA. Linkage analysis of candidate genes in autoimmune thyroid disease: 1. Selected immunoregulatory genes. International Consortium for the Genetics of Autoimmune Thyroid Disease. J Clin Endocrinol Metab 1998;83:1580-4.
- 54. Tomer Y, Barbesino G, Greenberg DA, Concepcion E, Davies TF. Linkage analysis of candidate genes in autoimmune thyroid disease. III. Detailed analysis of chromosome 14 localizes Graves disease-1 (GD-1) close to multinodular goiter-1 (MNG-1). International Consortium for the Genetics of Autoimmune Thyroid Disease. J Clin Endocrinol Metab 1998;83:4321-7.
- 55. Tomer Y, Barbesino G, Greenberg DA, Concepcion E, Davies TF. A new Graves disease-susceptibility locus maps to chromosome 20q11.2. International Consortium for the Genetics of Autoimmune Thyroid Disease. Am J Hum Genet 1998;63:1749-56.
- 56. Jenkins D, Penny MA, Fletcher JA, Jacobs KH, Mijovic CH, Franklyn JA, et al. HLA class II gene polymorphism contributes little to Hashimotos thyroiditis. Clin Endocrinol 1992;37:141-5.
- 57. Farid NR, Thompson C. HLA and autoimmune endocrine disease 1985. Mol Biol Med 1986;3:85-97.
- 58. Shi Y, Zou M, Robb D, Farid NR. Typing for major histocompatibility complex class II antigens in thyroid tissue blocks: association of Hashimotos thyroiditis with HLA-DQA0301 and DQB0201 alleles. J Clin Endocrinol Metab 1992;75:943-6.
- 59. Thompson C, Farid NR. Post-partum thyroiditis and goitrous (Hashimotos) thyroiditis are associated with HLA-DR4. Immunol Lett 1985;11:301-3.
- 60. Nakai Y, Tanaka I, Fukata J, Nakao K, Oki S, Takai S, et al. Evidence for gamma-MSH-like immunoreactivity in ectopic ACTH-producing tumors. J Clin Endocrinol Metab 1980;50:1147-8.
- 61. Farid NR, Barnard JM, Marshall WH, Woolfrey I, ODriscoll RF. Thyroid autoimmune disease in a large Newfoundland family: the influence of HLA. J Clin Endocrinol Metab 1977;45:1165-72.
- 62. Ito M, Tanimoto M, Kamura H, Yoneda M, Morishima Y, Uamauchi K, et al. Association of HLA antigen and restriction fragment length polymorphism of T cell receptor beta chain gene with Graves disease and Hashimotos thyroiditis. J Clin Endocrinol Metab 1989;69:100-4.
- 63. Weetman AP, So AK, Roe C, Walport MJ, Foroni L. T cell receptor alpha chain V region polymorphism linked to primary autoimmune hypothyroidism but not Graves disease. Hum Immunol 1987;20:167-73.
- 64. Thompson CB, Allison JP. The emerging role of CTLA-4 as an immune attenuator. Immunity 1997;7:445-50.
- 65. Yanagawa T, Ito K, Kaplan EL, Ishikawa N, DeGroot LJ. Absence of association between human spumaretrovirus and Graves disease. Thyroid 1995;5:379-82.
- 66. Gambelunghe G, Falorni A, Ghaderi M, Laureti S, Tortoioli C, Santeusanio F, et al. Microsatellite polymorphism of the MHC class I chain-related (MIC-A and MIC-B) genes marks the risk for autoimmune Addisons disease. J Clin Endocrinol Metab 1999;84:3701-7.
- 67. Myhre AG, Undlien DE, Lovas K, Uhlving S, Nedrebo BG, Fougner KJ, et al. Autoimmune adrenocortical failure in Norway autoantibodies and human leukocyte antigen class II associations related to clinical features. J Clin Endocrinol Metab 2002;87:618-23.
- 68. Neufeld M, Maclaren N, Blizzard R. Autoimmune polyglandular syndromes. Pediatr Ann 1980;9:154-62.
- 69. Betterle C, Zanchetta R. Update on autoimmune polyendocrine syndromes (APS). Acta Biomed Ateneo Parmense 2003;74:9-33.
- 70. Wallaschofski H, Meyer A, Tuschy U, Lohmann T. HLA-DQA1*0301-associated susceptibility for autoimmune polyglandular syndrome type II and III. Horm Metab Res 2003;35:120-4.
- 71. Ahonen P, Koskimies S, Lokki ML, Tiilikainen A, Perheentupa J. The expression of autoimmune polyglandular disease type I appears associated with several HLA-A antigens but not with HLA-DR. J Clin Endocrinol Metab 1988;66:1152-7.
- 72. Huang W, Connor E, Rosa TD, Muir A, Schatz D, Silverstein J, et al. Although DR3-DQB1*0201 may be associated with multiple component diseases of the autoimmune polyglandular syndromes, the human leukocyte antigen DR4-DQB1*0302 haplotype is implicated only in beta-cell autoimmunity. J Clin Endocrinol Metab 1996;81:2559-63.
- 73. Hock A, Schoemaker J, Drexhage HA. Premature ovarian failure and ovarian autoimmunity. Endocr Rev 1997;18:107-34.
- 74. Walfish PG, Gottesman IS, Shewchuk AB, Bain J, Hawe BS, Farid NR. Association of premature ovarian failure with HLA antigens. Tissue Antigens 1983;21:168-9.
- 75. Jaroudi KA, Arora M, Sheth KV, Sieck UV, Willemsen WN. Human leukocyte antigen typing and associated abnormalities in premature ovarian failure. Hum Reprod 1994;9:2006-9.
- 76. Anasti JN, Adams S, Kimzey LM, Defensor RA, Zachary AA, Nelson LM. Karyotypically normal spontaneous premature ovarian failure: evaluation of association with the class II major histocompatibility complex. J Clin Endocrinol Metab 1994;78:722-3.
Datas de Publicação
-
Publicação nesta coleção
26 Jan 2004 -
Data do Fascículo
Out 2003
Histórico
-
Aceito
01 Ago 2003 -
Revisado
21 Jul 2003 -
Recebido
11 Nov 2002