Resumos
O objetivo desta comunicação foi descrever a detecção de coexistência de variantes HIV-1 com inserções de dois aminoácidos entre os códons 69 e 70 da transcriptase reversa. Tais variantes foram isoladas de paciente do sexo masculino, 16 anos de idade, em tratamento no interior do estado de São Paulo. Após confirmação de falha terapêutica, foi realizado teste de resistência a antirretrovirais, a partir do qual foram detectadas duas variantes contendo inserções dos aminoácidos Ser-Gly/Ser-Ala no códon 69 da transcriptase reversa, além da mutação T69S. Tais inserções possuem baixa prevalência, não foram relatadas em caráter de coexistência no Brasil e estão relacionadas com a resistência a múltiplas drogas, tornando o achado relevante do ponto de vista epidemiológico.
HIV-1, genética; Transcriptase Reversa do HIV; Proteínas do Vírus da Imunodeficiência Humana; Farmacorresistência Viral Múltipla
El objetivo de ésta comunicación fue describir la detección de coexistencia de variantes VIH-1 con inserciones de dos aminoácidos entre los codones 69 y 70 de la transcriptasa inversa. Tales variantes fueron aisladas de paciente del sexo masculino, 16 años de edad, en tratamiento en el interior del estado de Sao Paulo. Posterior a la confirmación de la falla terapéutica, se realizó prueba de resistencia para antirretrovirales, a partir del cual se detectaron dos variantes que contenían dos inserciones de los aminoácidos Ser-Gly/Ser-Ala en el codón 69 de la transcriptasa inversa, además de la mutación T69S. Tales inserciones poseen baja prevalencia, no fueron relatadas en carácter de coexistencia en Brasil y están relacionadas con la resistencia a múltiples drogas, tomando el resultado relevante desde el punto de vista epidemiológico.
VIH-1, genética; Transcriptasa Inversa del VIH; Proteínas del Virus de la Inmunodeficiencia Humana; Farmacorresistencia Viral Múltiple
The aim of this communication was to describe the detection of the coexistence of HIV-1 variant with dipeptide insertion between codons 69 and 70 of reverse transcriptase. These variants were isolated from a 16-year-old male patient, undergoing treatment in the city of Marilia, SP, Southeastern Brazil. After confirmation of treatment failure, resistance to antiretroviral drugs testing was performed and two variants with the insertions of the aminoacids Ser-Gly/Ser-Ala at codon 69 of reverse transcriptase were detected, besides the T69S mutation. These insertions have low prevalence, have not been reported in situations of coexistence in Brazil and are related to multidrug resistance, which makes this epidemiological finding relevant.
HIV-1, genetics; HIV Reverse Transcriptase; Human Immunodeficiency Virus Proteins; Drug Resistance, Multiple, Viral
INTRODUÇÃO
A pressão seletiva do esquema terapêutico antirretroviral (ARV) pode resultar na emergência de variantes resistentes, as quais pré-existem minoritariamente no paciente infectado com HIV. Mutações de ponto de uma ou duas bases no gene da transcriptase reversa (TR) estão associadas com resistência aos inibidores dessa enzima. Além disso, há relatos de que a resistência a múltiplos análogos nucleosídeos pode também ser resultante de diversas inserções/mutações existentes nos vírus. 11 . Larder BA, Bloor S, Kemp SD, Kurt H, Desmet RL, Miller V, et al. A family of insertion mutations between codons 67 and 70 of human immunodeficiency virus type 1 reverse transcriptase confer multinucleoside analog resistance. Antimicrob Agents Chemother. 1999;43(8):1961-7. , 55 . Winters MA, Coolley KL, Girard YA, Levee DJ, Hamdan H, Shafer RW, et al. A 6-basepair insert in the reverse transcriptase gene of human immunodeficiency virus type 1 confers resistance to multiple nucleoside inhibitors. J Clin Invest.1998;102(10):1769-75.DOI:10.1172/JCI4948
https://doi.org/10.1172/JCI4948...
O uso prolongado da terapia ARV com zidovudina, frequentemente junto ou seguida pela administração de outros inibidores nucleosídeos, está associado com a seleção de variantes com inserção de aminoácidos entre os resíduos 69 e 70 da TR do HIV-1. 33 . Tamalet C, Yahi N, Tourrès C, Colson P, Quinson AM, Poizot-Martin I, et al. Multidrug resistance genotypes (insertions in the â3-â4 finger subdomain and MDR Mutations) of HIV-1 reverse transcriptase from extensively treated patients: incidence and association with other resistance mutations. Virology . 2000;270(2):310-6. DOI:10.1006/viro.2000.0261
https://doi.org/10.1006/viro.2000.0261...
, 55 . Winters MA, Coolley KL, Girard YA, Levee DJ, Hamdan H, Shafer RW, et al. A 6-basepair insert in the reverse transcriptase gene of human immunodeficiency virus type 1 confers resistance to multiple nucleoside inhibitors. J Clin Invest.1998;102(10):1769-75.DOI:10.1172/JCI4948
https://doi.org/10.1172/JCI4948...
O objetivo deste estudo foi descrever a detecção de coexistência de variantes HIV-1 com inserções de dois aminoácidos entre os códons 69 e 70 da TR.
DESCRIÇÃO DO CASO
Paciente do sexo masculino, 16 anos, pardo e com exposição vertical ao vírus HIV. O caso foi notificado em 1996 e, desde então, manteve carga viral detectável e níveis de CD4+ baixo, sem, no entanto, desenvolver sintomas da síndrome da imunodeficiência adquirida (Aids). A menor contagem de linfócitos CD4+ ocorreu em 2011, 234 cels/mm 33 . Tamalet C, Yahi N, Tourrès C, Colson P, Quinson AM, Poizot-Martin I, et al. Multidrug resistance genotypes (insertions in the â3-â4 finger subdomain and MDR Mutations) of HIV-1 reverse transcriptase from extensively treated patients: incidence and association with other resistance mutations. Virology . 2000;270(2):310-6. DOI:10.1006/viro.2000.0261
https://doi.org/10.1006/viro.2000.0261...
, e a carga viral no mesmo ano foi de 4,624 log. O uso de ARV teve início em 2007 com os inibidores da TR zidovudina e didanosina, seguido da introdução de efavirenz em 2008. Após o compromisso de adesão do paciente, o teste de resistência a antirretrovirais (TRUGENE ® HIV-1 Genotyping Test on an OpenGene ® DNA Sequencing System, Siemens Healthcare Diagnostics , Deerfield, IL, USA) foi solicitado em 2011 e realizado no Laboratório de Biologia Molecular do Hemocentro da Faculdade de Medicina de Botucatu, Universidade Estadual Paulista (UNESP), pertencente à Rede Nacional de Genotipagem do HIV-1 (RENAGENO) do Ministério da Saúde.
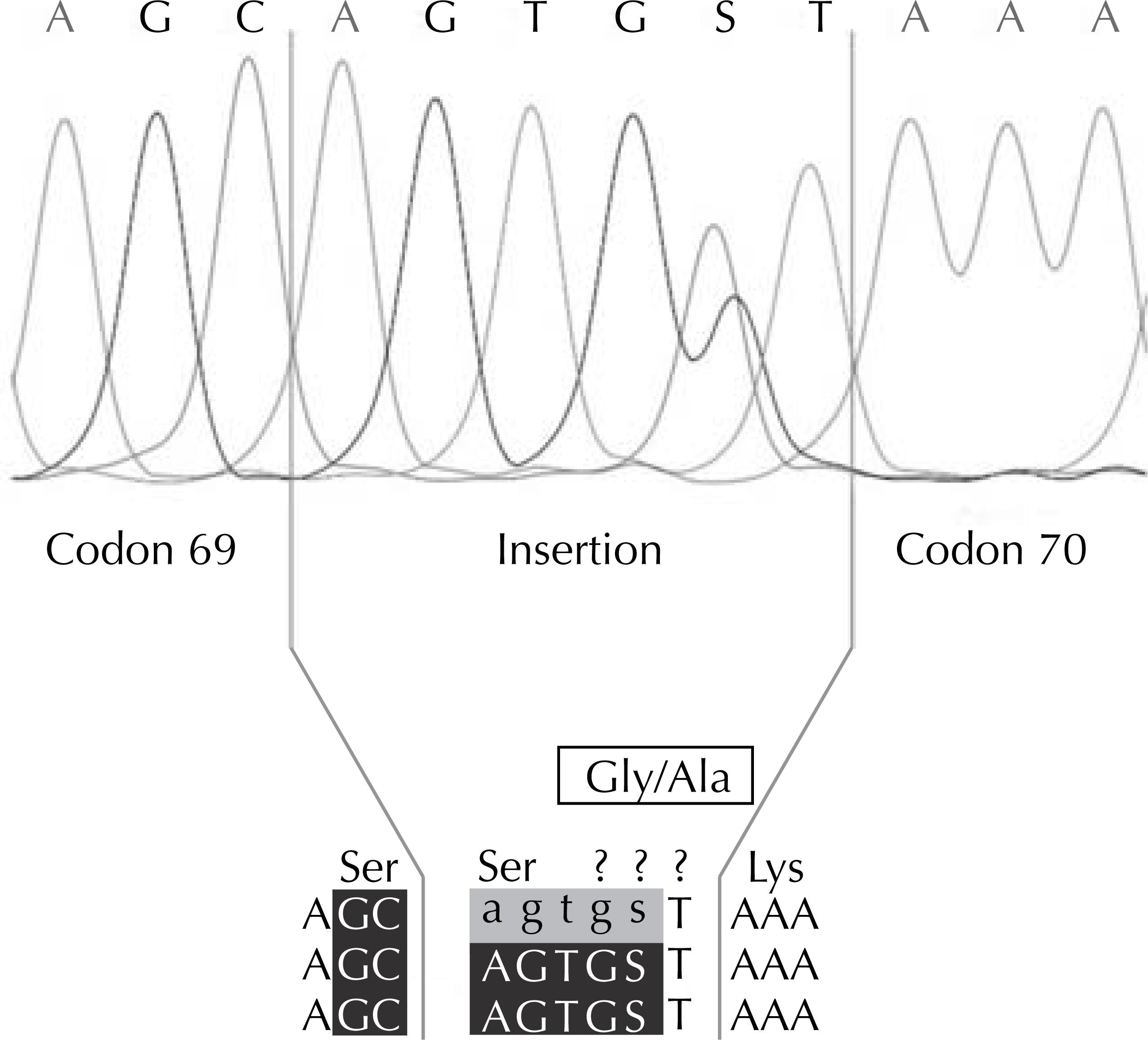
O subtipo viral caracterizado foi F1, utilizando o gene da transcriptase reversa, segundo análise realizada pela ferramenta REGA HIV Subtyping Tool . a a REGA HIV Subtyping Tool [citado 2012 nov 19]. Disponível em: http://www.bioafrica.net/rega-genotype/html/subtypinghiv.html A sequência obtida evidenciou a mutação T69S, seguida das inserções Ser-Gly (SG) / Ser-Ala (SA), o que sugere a coexistência de variantes virais com inserções de aminoácidos distintos no códon 69, confirmada a partir de sequências geradas por primers forward e reverse , segundo especificações da técnica utilizada. A análise do eletroferograma ( Figura ) mostrou as sequências de nucleotídeos AGT e G S T, simultaneamente.
Parte do eletroferograma de uma sequência genômica da região da transcriptase reversa do HIV-1, evidenciando a inserção de dois aminoácidos (Ser e Gly/Ala) entre os códons 69 e 70, a qual foi confirmada a partir de sequências geradas por primers forward e reverse , segundo especificações da técnica utilizada (TRUGENE ® hiv-1 Genotyping Test on na OpenGene ® DNA Sequencing System , Siemens Healthcare Diagnostics , Deerfield , IL, EUA).
O perfil de mutação incluiu as mutações A62V, K101P/Q, K103N, V106I, V179D/E, Y188L, T215Y no gene da transcriptase reversa (TR), além das mutações L10I e M36I na protease. Não foi observada nenhuma resistência aos inibidores de protease. Em contrapartida, houve resistência a múltiplas drogas da classe dos inibidores de TR.
A sequência obtida foi analisada utilizando o algoritmo Blast Basic Local Alignment Search Tool (Blast), b b National Center for Biotechnology Information (NCBI). Basic Local Alignment Search Tool (BLAST); 2012 [citado 2012 nov 20]. Disponível em: http://blast.ncbi.nlm.nih.gov/Blast.cgi disponível no National Center for Biotechnology Information (NCBI), sendo constatada uma similaridade de 93%.
DISCUSSÃO
A inserção de dois aminoácidos entre os resíduos 69 e 70 da TR do HIV-1 por si só não confere resistência significativa. No entanto, quando associada às substituições, como T215Y, M41L, A62V ou L210W, pode ocasionar a emergência de variantes resistentes a múltiplos análogos de nucleosídeos. 11 . Larder BA, Bloor S, Kemp SD, Kurt H, Desmet RL, Miller V, et al. A family of insertion mutations between codons 67 and 70 of human immunodeficiency virus type 1 reverse transcriptase confer multinucleoside analog resistance. Antimicrob Agents Chemother. 1999;43(8):1961-7. , 33 . Tamalet C, Yahi N, Tourrès C, Colson P, Quinson AM, Poizot-Martin I, et al. Multidrug resistance genotypes (insertions in the â3-â4 finger subdomain and MDR Mutations) of HIV-1 reverse transcriptase from extensively treated patients: incidence and association with other resistance mutations. Virology . 2000;270(2):310-6. DOI:10.1006/viro.2000.0261
https://doi.org/10.1006/viro.2000.0261...
, 55 . Winters MA, Coolley KL, Girard YA, Levee DJ, Hamdan H, Shafer RW, et al. A 6-basepair insert in the reverse transcriptase gene of human immunodeficiency virus type 1 confers resistance to multiple nucleoside inhibitors. J Clin Invest.1998;102(10):1769-75.DOI:10.1172/JCI4948
https://doi.org/10.1172/JCI4948...
As mutações de inserção entre os códons 69 e 70 estão presentes, com baixa prevalência (aproximadamente 0,5% a 2,4%), 22 . Masquelier B, Race E, Tamalet C, Descamps D, Izopet J, Buffet-Janvresse C, et al. Genotypic and phenotypic resistance patterns of human immunodeficiency virus type 1 variants with insertions or deletions in the reverse transcriptase (RT): multicenter study of patients treated with RT inhibitors. Antimicrob Agents Chemother.2001;45(6):1836-42. DOI:10.1128/AAC.45.6.1836-1842.2001
https://doi.org/10.1128/AAC.45.6.1836-18...
, 33 . Tamalet C, Yahi N, Tourrès C, Colson P, Quinson AM, Poizot-Martin I, et al. Multidrug resistance genotypes (insertions in the â3-â4 finger subdomain and MDR Mutations) of HIV-1 reverse transcriptase from extensively treated patients: incidence and association with other resistance mutations. Virology . 2000;270(2):310-6. DOI:10.1006/viro.2000.0261
https://doi.org/10.1006/viro.2000.0261...
, 44 . Van Vaerenbergh K, Van Laethem K, Albert J, Boucher CA, Clotet B, Floridia M, et al. Prevalence and characteristics of multinucleoside-resistant human immunodeficiency virus type 1 among European patients receiving combinations of nucleoside analogues. Antimicrob Agents Chemother . 2000;44(8):2109-17. DOI:10.1128/AAC.44.8.2109-2117.2000
https://doi.org/10.1128/AAC.44.8.2109-21...
em indivíduos experimentados com inibidores da TR análogos nucleosídeos, sendo mais comum a inserção dos dipeptídeos serina-serina, serina-arginina e serina-glicina. Tais rearranjos estão localizados na estrutura hairpin loop β3-β4 da TR do HIV-1 e estão suscetíveis à interação com o processo de ligação de nucleotídeos. 22 . Masquelier B, Race E, Tamalet C, Descamps D, Izopet J, Buffet-Janvresse C, et al. Genotypic and phenotypic resistance patterns of human immunodeficiency virus type 1 variants with insertions or deletions in the reverse transcriptase (RT): multicenter study of patients treated with RT inhibitors. Antimicrob Agents Chemother.2001;45(6):1836-42. DOI:10.1128/AAC.45.6.1836-1842.2001
https://doi.org/10.1128/AAC.45.6.1836-18...
Em 1999, Larder et al 11 . Larder BA, Bloor S, Kemp SD, Kurt H, Desmet RL, Miller V, et al. A family of insertion mutations between codons 67 and 70 of human immunodeficiency virus type 1 reverse transcriptase confer multinucleoside analog resistance. Antimicrob Agents Chemother. 1999;43(8):1961-7. relataram que a alteração 69Ser-(Ser-Ser), por si só, demonstrou apenas uma diminuição significante na suscetibilidade ao 3TC. No entanto, Winters et al 55 . Winters MA, Coolley KL, Girard YA, Levee DJ, Hamdan H, Shafer RW, et al. A 6-basepair insert in the reverse transcriptase gene of human immunodeficiency virus type 1 confers resistance to multiple nucleoside inhibitors. J Clin Invest.1998;102(10):1769-75.DOI:10.1172/JCI4948
https://doi.org/10.1172/JCI4948...
(1998) reportaram que a mutação 69Ser-(Ser-Ala) ou 69Ser-(Ser-Gly), mesmo sem demais mutações genotípicas, conferia resistência a múltiplas drogas.
A detecção da substituição única, Thr69Ser, sem inserção, sugere que a mutação possa se desenvolver antes da inserção de aminoácido. No entanto, ainda não é conhecido especificamente o mecanismo preciso para a ocorrência da inserção. 11 . Larder BA, Bloor S, Kemp SD, Kurt H, Desmet RL, Miller V, et al. A family of insertion mutations between codons 67 and 70 of human immunodeficiency virus type 1 reverse transcriptase confer multinucleoside analog resistance. Antimicrob Agents Chemother. 1999;43(8):1961-7.
No Brasil, não há relatos que demonstram a presença de variantes HIV-1 com inserção de dipeptídeos entre os códons 69 e 70 da TR em coexistência, o que torna o achado relevante do ponto de vista epidemiológico, uma vez que tal inserção está relacionada com a resistência a múltiplos análogos de nucleosídeos e pouco se sabe sobre sua prevalência e significância clínica.
REFERÊNCIAS
-
1Larder BA, Bloor S, Kemp SD, Kurt H, Desmet RL, Miller V, et al. A family of insertion mutations between codons 67 and 70 of human immunodeficiency virus type 1 reverse transcriptase confer multinucleoside analog resistance. Antimicrob Agents Chemother. 1999;43(8):1961-7.
-
2Masquelier B, Race E, Tamalet C, Descamps D, Izopet J, Buffet-Janvresse C, et al. Genotypic and phenotypic resistance patterns of human immunodeficiency virus type 1 variants with insertions or deletions in the reverse transcriptase (RT): multicenter study of patients treated with RT inhibitors. Antimicrob Agents Chemother.2001;45(6):1836-42. DOI:10.1128/AAC.45.6.1836-1842.2001
» https://doi.org/10.1128/AAC.45.6.1836-1842.2001 -
3Tamalet C, Yahi N, Tourrès C, Colson P, Quinson AM, Poizot-Martin I, et al. Multidrug resistance genotypes (insertions in the â3-â4 finger subdomain and MDR Mutations) of HIV-1 reverse transcriptase from extensively treated patients: incidence and association with other resistance mutations. Virology . 2000;270(2):310-6. DOI:10.1006/viro.2000.0261
» https://doi.org/10.1006/viro.2000.0261 -
4Van Vaerenbergh K, Van Laethem K, Albert J, Boucher CA, Clotet B, Floridia M, et al. Prevalence and characteristics of multinucleoside-resistant human immunodeficiency virus type 1 among European patients receiving combinations of nucleoside analogues. Antimicrob Agents Chemother . 2000;44(8):2109-17. DOI:10.1128/AAC.44.8.2109-2117.2000
» https://doi.org/10.1128/AAC.44.8.2109-2117.2000 -
5Winters MA, Coolley KL, Girard YA, Levee DJ, Hamdan H, Shafer RW, et al. A 6-basepair insert in the reverse transcriptase gene of human immunodeficiency virus type 1 confers resistance to multiple nucleoside inhibitors. J Clin Invest.1998;102(10):1769-75.DOI:10.1172/JCI4948
» https://doi.org/10.1172/JCI4948
-
a
REGA HIV Subtyping Tool [citado 2012 nov 19]. Disponível em: http://www.bioafrica.net/rega-genotype/html/subtypinghiv.html
-
b
National Center for Biotechnology Information (NCBI). Basic Local Alignment Search Tool (BLAST); 2012 [citado 2012 nov 20]. Disponível em: http://blast.ncbi.nlm.nih.gov/Blast.cgi
Datas de Publicação
-
Publicação nesta coleção
Ago 2013
Histórico
-
Recebido
18 Dez 2012 -
Aceito
25 Abr 2013