Resumos
Este trabalho teve como objetivo caracterizar molecularmente genótipos elite, e recomendados, de bananeira, por meio de marcadores RAPD e microssatélites. Foram utilizados 47 primers de RAPD e 34 primers de microssatélites. Foi também conduzido um ensaio de contaminação, utilizando-se o primer AGMI 24-25, cuja variedade Tropical foi considerada a amostra-padrão, e as cultivares Caipira e Prata Graúda como contaminantes. Os marcadores permitiram separar as cultivares de acordo com a origem e a constituição genômica e definiram padrões moleculares para algumas cultivares avaliadas. As cultivares Garantida, Preciosa e Pacovan Ken apresentaram alta similaridade genética com ambos marcadores. O primer AGMI 24-25 demonstrou alta capacidade discriminatória de genótipos em ensaios de contaminação.
Musa spp.; certificação genética; proteção varietal; fingerprinting; marcadores moleculares
The objective of this work was to characaterize elite banana genotypes, and those recommended, using RAPD and microsatellites. Forty-seven RAPD primers and 34 microsatellites primers were used. A contamination essay using the AGMI 24-25 primer was also carried out. Tropical variety was considered the standard sample and the Caipira and Prata Graúda, contaminants. Markers were able to separate the cultivars according to their origin and genomic constitution as well as defined molecular profiles for some of the cultivars evaluated. Garantida, Preciosa and Pacovan Ken cultivars presented genetic similarity with both markers. The AGMI 24-25 primer demonstrated high capacity to discriminate the genotypes in the contamination essay.
Musa spp.; genetic certification; varietal protection; fingerprinting; molecular markers
GENÉTICA
Diferenciação molecular de cultivares elites de bananeira
Molecular differentiation of elite banana cultivars
Onildo Nunes de JesusI; Terezinha Rangel CâmaraII; Cláudia Fortes FerreiraIII; Sebastião de Oliveira e SilvaIII; Kátia Nogueira PestanaIV; Taliane Leila SoaresIV
IUniversidade Federal Rural de Pernambuco (UFRPE), Dep. de Agronomia, Rua D. Manoel de Medeiros, s/nº, Dois Irmãos, CEP 52171-900 Recife, PE. E-mail: onildonunes@yahoo.com.br
IIUFRPE, Dep. de Química Vegetal. E-mail: tkrcamara@bol.com.br
IIIEmbrapa Mandioca e Fruticultura Tropical, Caixa Postal 007, CEP 44380-000 Cruz das Almas, BA. E-mail: claudiaf@cnpmf.embrapa.br, ssilva@cnpmf.embrapa.br
IVUniversidade Federal da Bahia, Escola de Agronomia, CEP 44380-000 Cruz das Almas, BA. E-mail: katypestana@yahoo.com.br, talialeila@hotmail.com
RESUMO
Este trabalho teve como objetivo caracterizar molecularmente genótipos elite, e recomendados, de bananeira, por meio de marcadores RAPD e microssatélites. Foram utilizados 47 primers de RAPD e 34 primers de microssatélites. Foi também conduzido um ensaio de contaminação, utilizando-se o primer AGMI 24-25, cuja variedade Tropical foi considerada a amostra-padrão, e as cultivares Caipira e Prata Graúda como contaminantes. Os marcadores permitiram separar as cultivares de acordo com a origem e a constituição genômica e definiram padrões moleculares para algumas cultivares avaliadas. As cultivares Garantida, Preciosa e Pacovan Ken apresentaram alta similaridade genética com ambos marcadores. O primer AGMI 24-25 demonstrou alta capacidade discriminatória de genótipos em ensaios de contaminação.
Termos para indexação:Musa spp., certificação genética, proteção varietal, fingerprinting, marcadores moleculares.
ABSTRACT
The objective of this work was to characaterize elite banana genotypes, and those recommended, using RAPD and microsatellites. Forty-seven RAPD primers and 34 microsatellites primers were used. A contamination essay using the AGMI 24-25 primer was also carried out. Tropical variety was considered the standard sample and the Caipira and Prata Graúda, contaminants. Markers were able to separate the cultivars according to their origin and genomic constitution as well as defined molecular profiles for some of the cultivars evaluated. Garantida, Preciosa and Pacovan Ken cultivars presented genetic similarity with both markers. The AGMI 24-25 primer demonstrated high capacity to discriminate the genotypes in the contamination essay.
Index terms:Musa spp., genetic certification, varietal protection, fingerprinting, molecular markers.
Introdução
A bananeira (Musa spp.) é cultivada de Norte a Sul do País e a maioria dos bananicultores são pequenos agricultores que utilizam essa fruta como fonte de alimentação e renda.
As cultivares de bananeira usadas no País apresentam baixa produtividade e suscetibilidade às principais pragas e doenças da cultura. No entanto, novas variedades de bananeira vêm sendo produzidas pela Embrapa Mandioca e Fruticultura Tropical, resultando na recomendação de algumas com resistência às sigatokas e ao mal-do-panamá.
Com o surgimento de novas cultivares, teve início a preocupação em caracterizar geneticamente esses genótipos para registro e proteção, permitindo, assim, o controle sobre sua multiplicação e comercialização (Priolli et al., 2002; Bonamico et al., 2004).
No Brasil, a proteção de cultivares está amparada pela lei nº 9.456, de 1997. Essa lei assegura, ao titular, o direito à reprodução comercial da cultivar no território brasileiro, ficando vedados a terceiros, durante o prazo de proteção, a produção com fins comerciais e o oferecimento à venda ou à comercialização do material de propagação da cultivar sem autorização (Brasil, 1998). Assim, torna-se necessária a caracterização precisa da cultivar, com aplicação de descritores homogêneos quanto a sua expressão entre os estádio de desenvolvimento e ao longo dos ciclos (Carvalho et al., 2000).
Os melhoristas têm utilizado características morfológicas e bioquímicas para registrar e proteger suas variedades. Entretanto, embora o emprego desse método seja valioso, há limitações em casos de cultivares aparentadas (Priolli et al., 2002), especificamente no que tange a cultivares resultantes do programa de melhoramento de bananeira da Embrapa, em que se usa um pequeno número de genitores femininos, que são muito próximos geneticamente. Além disso, esses descritores são influenciados pelo ambiente, são complexos em suas interações alélicas (Lombard et al., 2005) e constituem uma base pobre para medir a identidade, por apresentarem uma medida indireta da composição genética do material (Binneck et al., 2002).
Os descritores de DNA, por sua vez, apresentam a vantagem de representarem o genótipo, mantendo consistência nos resultados, evitando o problema da avaliação dos dados com base na expressão do fenótipo (Wünsch & Hormaza, 2002). A possibilidade de acessar a variabilidade genética diretamente no DNA, vem fazendo com que, cada vez mais, sejam disponibilizadas técnicas precisas que possam vir a auxiliar o processo de proteção intelectual de materiais genéticos (Padilha et al., 2002).
O RAPD (Random Amplified Polimorphic DNA) consiste em utilizar primers de seqüência arbitrária para dirigir reações de amplificação, gerando, assim, grande quantidade de polimorfismo de fragmentos de DNA, oriundos de diferentes partes, espalhados por todo o genoma (incluindo regiões de DNA repetitivo). Essa técnica pode ser usada no fingerprinting, além de permitir a detecção de divergências mínimas entre espécies ou clones (Brammer, 2000). A utilização do RAPD para fins de proteção é limitada, devido principalmente a baixa repetibilidade entre laboratórios. No entanto, a conversão dessa técnica em marcadores do tipo SCAR (Sequence Characterized Amplified Regions) possibilita contornar o problema da repetibilidade (Carvalho et al., 2000), podendo ser usada na identificação varietal.
Alternativamente, os SSR (Simple Sequence Repeats) consistem em mono a tetranucleotídeos repetidas em tandem, que são amplificados por primers específicos. São marcadores altamente polimórficos, possuem expressão co-dominante e são multialélicos e de alta repetibilidade entre laboratórios. Em virtude do grande número de regiões repetitivas, os SSR se tornam marcadores altamente informativos e cada indivíduo pode apresentar um fingerprinting único (Decroocq et al., 2004).
O RAPD já foi utilizado em vários trabalhos de caracterização e identificação de genótipos de bananeira (Crouch et al., 1998; Paz, 2000; Pillay et al., 2000; Creste et al., 2001, 2003). A caracterização por marcadores moleculares pode auxiliar na identificação e certificação da qualidade genética do material em uso (Fachinello et al., 2000) e na detecção de pureza genética dos lotes (Ramos, 2004), garantindo que as características de interesse acrescentadas aos materiais comerciais sejam mantidas e expressas nos cultivos subseqüentes (Padilha et al., 2003). O uso de técnicas moleculares em lotes de mudas de plantas de propagação vegetativa poderá tornar-se fundamental em futuro próximo, podendo até mesmo detectar variantes somaclonais.
Este trabalho teve como objetivo caracterizar molecularmente genótipos elites e recomendados de bananeira por meio de marcadores RAPD e microssatélites.
Material e Métodos
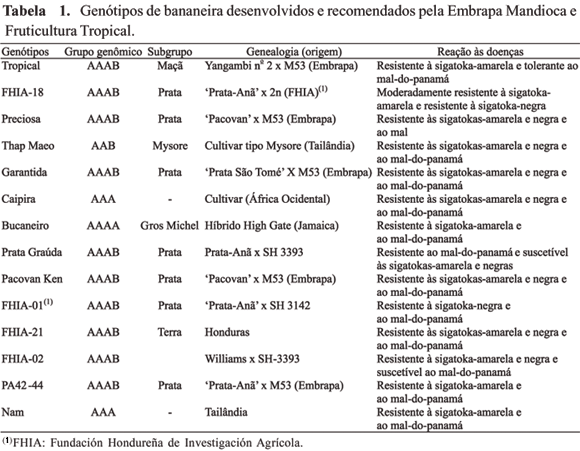
Foram caracterizadas molecularmente 14 cultivares de bananeira adaptadas, desenvolvidas e recomendadas pela Embrapa Mandioca e Fruticultura Tropical, que pertencem a diferentes grupos genômicos e possuem resistência variada a diversas doenças (Tabela 1).
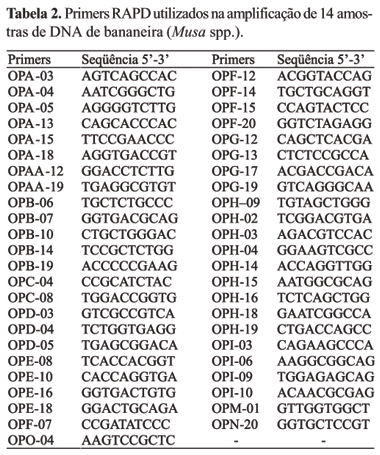
A extração de DNA foliar foi realizada no Laboratório de Virologia e Biologia Molecular da Embrapa Mandioca e Fruticultura Tropical, a partir de folhas jovens, seguindo o método descrito por Doyle & Doyle (1990). A avaliação da quantidade e qualidade do DNA foi efetuada mediante análise comparativa das amostras em gel de agarose 0,8%, corado com brometo de etídio (0,5 µg mL-1). As amostras foram diluídas em água ultrapura, padronizando a concentração de trabalho em 10 ng µL-1. As reações de amplificação por meio do RAPD (Tabela 2) foram conduzidas de acordo com os procedimentos descritos por Williams et al. (1990). As bandas de DNA foram visualizadas em gel de agarose (1,5%), sob luz ultravioleta, e as imagens, captadas pelo sistema Kodak Digital de fotodocumentação.
As reações de amplificação por SSR foram realizadas em volume final de 25 µL, contendo: 50 mM KCl, 10 mM Tris-HCl (pH 8,3), 2,4 mM MgCl2, 100 mM de cada dNTPs, 0,2 mM de primer, 50 ng de DNA e uma unidade de Taq polimerase (Pharmacia Biotech, EUA). Os primers foram cedidos pelo Laboratório de Biologia Molecular do Centro de Energia Nuclear na Agricultura, USP, Piracicaba, SP, e pela Embrapa Recursos Genéticos e Biotecnologia, Brasília, DF. As amplificações foram realizadas no termociclador My-Cycler Thermocycler (BioRAD) e cada primer foi testado conforme a temperatura de anelamento sugerida pelo fabricante, seguido de um programa de touchdown. Os ciclos de amplificação foram os seguintes: uma etapa a 3 min, dez ciclos a 94ºC por 40 s, 40 s em touchdown de 65ºC com diminuição de 1ºC a cada ciclo, 72ºC por 1 min, 24 ciclos a 94ºC por 40 s, 55ºC por 40 s, um ciclo final de 72ºC por 4 min e um ciclo a 4ºC até a retirada do termociclador.
O preparo do gel de poliacrilamida (5%), montagem das placas e revelação com prata encontram-se em Souza (2002). Depois da montagem das placas, o gel de poliacrilamida 5% foi preaquecido até a temperatura de 45ºC, sob eletroforese a 45 W durante 45 min (Souza, 2002). As amostras foram desnaturadas por 5 min a 95ºC em 5 µL de tampão desnaturante (10 mM de NaOH, 0,05% de xylenocyanol, 0,05% de azul de bromofenol e 20 mM de EDTA em formamida) e colocadas imediatamente em gelo, sendo aplicados 8 µL de cada amostra, sob uma eletroforese a 80 W durante 90 min. Depois da secagem do gel por 24 horas, as imagens foram escaneadas e armazenadas em arquivos eletrônicos.
Os produtos das amplificações foram computados como ausência (0) e presença (1) de bandas para as 14 cultivares elites avaliadas. A análise dos bootstrap, realizada com 1.000 reamostragens, e a dissimilaridade entre os genótipos, calculada pelo complemento do coeficiente de Jaccard, foram realizadas com o programa Genes (Cruz, 2001). A partir das matrizes de distância genética, os dendrogramas foram gerados pelo método da média aritmética não ponderada (UPGMA), com o auxílio do programa Statistica (StatSoft, 2005).
Com o intuito de demonstrar a eficiência dos marcadores microssatélites na detecção de contaminantes, em lotes de mudas de bananeira, foi efetuada uma simulação, misturando-se diferentes volumes de DNA exógeno. Nessa simulação, a mistura foi submetida a duas condições de amplificação, utilizando o primer AGMI 24-25. Em ambas as condições, a cultivar Tropical, com padrão de bandas conhecido, foi misturada com diferentes quantidades de DNA da cultivar Prata Graúda (T1) e com quantidades de DNA duas fontes de contaminação, as cultivares Caipira e Prata Graúda (T2). Na mistura de dois contaminantes, volumes iguais das duas amostras foram misturados e retiradas alíquotas (Tabela 3).
Resultados e Discussão
Foram avaliados 47 primers de RAPD, que produziram um total de 328 bandas, sendo 82 monomórficas e 246 polimórficas, que foram utilizadas nas análises. A média de bandas polimórficas por primer foi de 5,35, oscilando entre uma banda para os primers OPE-10, OPF-G-17 e OPE-08, e 16 bandas para o primer OPAA-12.
Nas análises com microssatélites, testaram-se 34 primers, dos quais 20 produziram bandas evidentes e foram utilizados nas análises. Sete primers apresentaram bandas não evidentes, três apresentaram falha na amplificação na maioria dos genótipos, três primers não geraram produtos de amplificação e apenas um primer apresentou bandas monomórficas (Tabela 4).
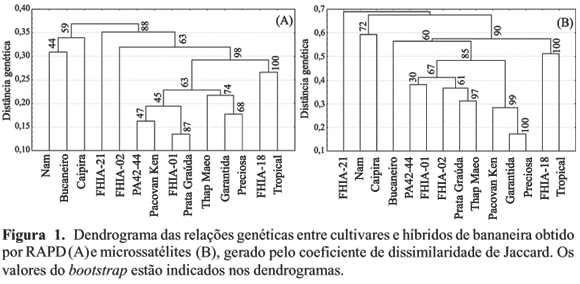
Observou-se que não houve muitas diferenças nos agrupamentos gerados pelos marcadores RAPD e SSR, quando analisados os dados separadamente, o que já era esperado, em virtude de ambos revelarem a variabilidade no DNA (Figura 1). Ambas as técnicas apresentaram valores altos de bootstrap (acima de 50%) na maioria dos grupos formados. Houve maior consistência das bifurcações, quando as cultivares foram analisadas com microssatélites.
Os marcadores RAPD foram capazes de separar as cultivares, segundo a origem e os grupos genômicos, a uma distância de 0,35. Em um grupo, foram inseridos todos os genótipos portadores do genoma A, e no outro foram agrupados todos os genótipos triplóides e tetraplóides portadores dos genomas A e B, incluindo, nesse último, todos os híbridos tetraplóides avaliados. O primeiro grupo incluiu as variedades Nam (AAA), Caipira (AAA) e Bucaneiro (AAAA). O segundo grupo incluiu as variedades Thap Maeo (AAB) e os híbridos tetraplóides AAAB, representados pelas cultivares FHIA-01, FHIA-02, FHIA-18, FHIA-21, Garantida, Preciosa, Pacovan Ken, Prata Graúda, Tropical e o híbrido PA42-44.
Os marcadores microssatélites revelaram comportamento semelhante aos apresentados pelos marcadores RAPD, em relação à separação, de acordo com o grupo genômico e origem dos híbridos, porém FHIA-21 ficou separado dos demais genótipos AAAB. Resultados semelhantes foram obtidos por Bhat et al. (1995) com marcadores RFLP e RAPD, por Howell et al. (1994), utilizando nove primers RAPD, e por Souza (2002) que, por meio de marcadores microssatélites, conseguiu separar cultivares e híbridos de bananeira segundo o nível de ploidia, grupo genômico e subgrupos.
Em ambas as técnicas, nota-se que a cultivar FHIA-21, um plátano, foi separada das demais cultivares. Essa classificação, assim obtida, se assemelha à caracterização morfológica, que separa a FHIA 21 dos demais genótipos por ser do subgrupo Terra (Plátano). Cultivares desse subgrupo apresentam frutos compridos com quinas evidentes, polpas rosadas e consistentes, ideais para o consumo cozido ou frito (Moreira, 1999). Os demais genótipos AAAB são do tipo Prata ou Maçã e mais adequados para consumo in natura.
Os híbridos tetraplóides Garantida, Preciosa e Pacovan Ken, respectivamente, resultantes dos cruzamentos das cultivares (AAB) Prata, São Tomé e Pacovan e com o genitor masculino diplóide (AA) M53, ficaram agrupados, quando se utilizou marcadores microssatélites. As cultivares Preciosa e Pacovan Ken são irmãos completos e a 'Pacovan' é uma mutação da 'Prata', intensificando, assim, o grau de parentesco entre os genótipos. Morfologicamente existem poucas diferenças entre essas três cultivares.
O resultado da amplificação com 22 primers via RAPD reforça, ainda mais, a alta similaridade genética dessas cultivares (Tabela 5). Apenas a cultivar Preciosa apresentou bandas características, enquanto a Garantida e a Pacovan Ken não diferiram no que tange a alguns primers avaliados. Creste et al. (2003), ao trabalhar com marcadores SSR, encontraram dificuldade em discriminar tais genótipos. O arranjo dos dendrogramas, alocando cultivares com origem comum, como dos mesmos subgrupos, é justificado pelos altos níveis de similaridade genética (Creste et al., 2003).
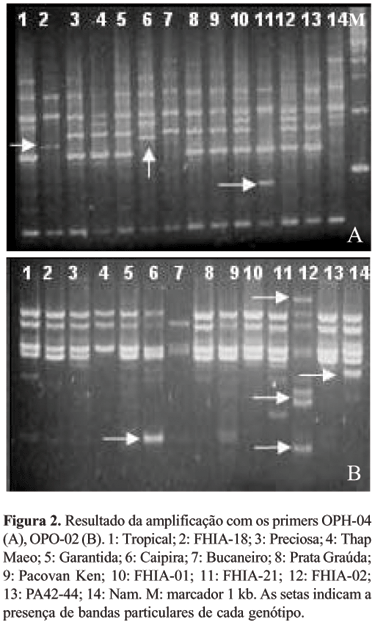
A identificação de cultivares, utilizando marcadores do tipo RAPD, é bastante divulgada em trabalhos de caracterização molecular (This et al., 1997; Sawazaki et al., 2002; Bianchi et al., 2003; Crochemore et al., 2004). Em alguns casos, ela é baseada na seleção de bandas e primers (Binneck et al., 2002) que, em experimentos posteriores, podem ser utilizados para confecção de marcadores do tipo SCAR. Neste trabalho, as cultivares FHIA-18, Caipira e FHIA-21 produziram um fragmento de banda específico com o primer OPH-04. Enquanto os genótipos Caipira, FHIA-02, FHIA-21 e Nam tiveram um padrão de banda característico com o primer OPO-02 (Figura 2).
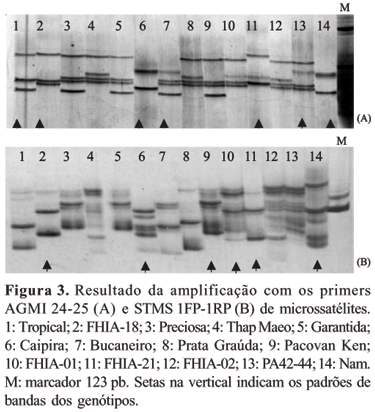
Os primers de microssatélites AGMI 24-25, Ma 3/103, STMS 1FP-1RP e Ma 1/24 permitiram a identificação das cultivares Bucaneiro, Caipira, FHIA-01, FHIA-18, FHIA-2, Tropical, PA42-44 e Nam (Figura 3). O primer AGMI 24-25 foi também utilizado por Kaemmer et al. (1997) na identificação de genótipos com o genoma B. Tal fato foi confirmado nos trabalhos de Creste et al. (2003) e neste estudo. No entanto, as cultivares Terra (AAB) (Creste et al., 2003) e FHIA 21 (AAAB) no presente estudo, que são plátanos, não apresentaram o alelo de identificação produzido pelo primer AGMI 24-25. De forma similar, o primer Ma 1/103 também identificou alelos característicos do genoma B, não demonstrando a presença desses alelos no plátano Terra (Creste et al., 2003) e plátano FHIA-21 neste trabalho.
O número de alelos dos SSRs variou de 4 a 10, porém o marcador utilizado não permitiu estabelecer os tamanhos dos alelos amplificados. As variações no tamanho (pb) dos alelos foram diferentes da observada por Creste et al. (2003) que, por sua vez, diferiram dos obtidos por Kaemmer et al. (1997), embora tenham sido utilizados os mesmos marcadores. Essa discrepância pode ser devida às condições de desnaturação durante a eletroforese, às diferenças na composição de bases e ao marcador de peso molecular usado (Testolin et al., 2000).
Com os primers AGMI 24-25 e STMS 1FP-1RP, nove genótipos foram discriminados. Desses, sete foram pelos diferentes alelos amplificados pelo primer AGMI 24-25. Isso indica sua alta capacidade discriminatória e reforça a sua característica multialélica.
A superioridade dos SSR em relação ao RAPD pode ser demonstrada por sua capacidade de identificar sete dos 14 genótipos avaliados com o padrão de bandas gerado por um único primer, o AGMI 24-25. Souza (2002), trabalhando com o mesmo primer, conseguiu uma discriminação de 83% dos genótipos avaliados. No entanto, quando se analisa a capacidade de separar genótipos aparentados, nota-se que o RAPD separou as cultivares de mesma origem em subgrupos diferentes no dendrograma. Tal fato é justificado pela alta cobertura do genoma proporcionada pela técnica, permitindo identificação de diferenças mínimas entre os genótipos avaliados (Brammer, 2000).
Não foram produzidas bandas características para os genótipos Preciosa, Garantida e FHIA-02. Todavia, a caracterização dessas cultivares pôde ser complementada com as informações dos primers RAPD. Os marcadores RAPD e SSR foram capazes de gerar um fingerprinting para discriminar as cultivares elites de bananeira estudadas (Tabela 6). As informações acumuladas dos perfis moleculares dos genótipos poderão ser registradas em bancos de dados, permitindo a acumulação de informações para o fingerprinting varietal.
A simulação da contaminação em lotes de muda de bananeira é mostrada na Figura 4. Observa-se que o primer SSR AGMI24-25 permitiu verificar variações mínimas presentes no DNA. Tanto no tratamento T1 como no T2, esse primer foi capaz de identificar todos os alelos das amostras utilizadas como contaminantes. Observou-se, no entanto, que a intensidade das bandas aumentam com o aumento da quantidade de DNA contaminante e que a melhor visualização da contaminação é registrada quando a concentração do contaminante é de 30%.
Embora com baixa visibilidade, foi possível detectar o DNA da 'Tropical' em uma proporção de 10% de DNA contaminante. Tal fato pode estar associado à utilização de iniciadores para amplificar uma região específica do genoma, mesmo em baixa concentração (Ramos, 2004). Assim, com uma reação contendo uma massa final de 50 ng de DNA, o primer é capaz de amplificar um DNA específico que esteja presente em uma quantidade de 5 ng. Esse resultado indica a alta especificidade da técnica de SSR na detecção de qualquer vestígio mínimo de DNA exógeno ou contaminante.
Verifica-se que a técnica é confiável, quando se busca identificar a pureza varietal nas amostras analisadas. Por isso, vários trabalhos relatam a utilização dos marcadores moleculares para identificação de pureza genética em diversas espécies (Priolli et al., 2002; Padilha et al., 2003; Ramos, 2004).
Conclusões
1. As cultivares Garantida, Preciosa e Pacovan Ken apresentam alta similaridade genética com ambos os marcadores.
2. O primer AGMI 24-25 apresenta alta capacidade de discriminar os genótipos avaliados e as contaminações envolvendo diferentes genótipos.
3. Os descritores baseados em microssatélites e RAPD permitem definir padrões moleculares somente para algumas cultivares avaliadas.
Agradecimentos
À Universidade Federal Rural de Pernambuco; aos técnicos do Laboratório de Virologia e Biologia Molecular da Embrapa Mandioca e Fruticultura Tropical; ao Centro de Energia Nuclear na Agricultura, USP; à Embrapa Recursos Genéticos e Biotecnologia; à Capes e à Fapesb, pelo apoio recebido.
Recebido em 8 de fevereiro de 2006 e aprovado em 28 de julho de 2006
- BHAT, K.V.; JARRET, R.L.; RANA, R.S. DNA profiling of banana and plantain cultivars using random amplified polymorphic DNA (RAPD) and restriction fragment length polymorphism (RFLP) markers. Electrophoresis, v.16, p.1736-1745, 1995.
- BIANCHI, V.J.; FACHINELLO, J.C.; SCHUCH, M.W. RAPDs na caracterização genético-molecular e no estudo da variabilidade genética de cultivares de ameixeira. Revista Brasileira de Fruticultura, v.25, p.272-274, 2003.
- BINNECK, E.; NEDEL, J.L.N.; DELLAGOSTIN, O.A. Análise de RAPD na identificação de cultivares: uma metodologia útil? Revista Brasileira de Sementes, v.24, p.183-196, 2002.
- BONAMICO, N.; AIASSA, J.; IBAÑEZ, M.; DI RENZO, M.; DÍAZ, D.; SALERNO, J. Caracterización y clasificación de híbridos simples de maíz con marcadores SSR. Revista de Investigaciones Agropecuarias, v.33, p.129-144, 2004.
- BRAMMER, S.P. Marcadores moleculares: princípios básicos e uso em programas de melhoramento genético vegetal. Passo Fundo: Embrapa Trigo, 2000. 7p. (Embrapa Trigo. Documentos online, 3). Disponível em: <http://www.cnpt.embrapa.br/biblio/p_do03.htm>. Acesso em: 5 jun. 2005.
- BRASIL. Legislação brasileira sobre proteção de cultivares Brasília: MA/SDR/SNPC, 1998. 115p.
- CARVALHO, M.L.M.; VIEIRA, M.G.G.C.; VON PINHO, E.R. Técnicas moleculares em sementes: aplicação de técnicas moleculares no controle de qualidade das sementes. Biotecnologia Ciência e Desenvolvimento, v.3, p.44-47, 2000.
- CRESTE, S.; TULMANN NETO, A.; FIGUEIRA, A. Detection of single sequence repeat polymorphisms in denaturing polyacrylamide sequencing gels by silver staining. Plant Molecular Biology Reporter, v.19, p.299-306, 2001.
- CRESTE, S.; TULMANN NETO, A.; SILVA, S.O.; FIGUEIRA, A. Genetic characterization of banana cultivars (Musa spp.) from Brazil using microsatellite markers. Euphytica, v.132, p.259-268, 2003.
- CROCHEMORE, M.L.; NUNES, L.M.; ANDRADE, A.; MOLINARI, H.B.C.M.; VASCONCELLOS, M.E. Varietal identification of coffe seed by RAPD technique. Brazilian Archives of Biology and Technology, v.47, p.7-11, 2004.
- CROUCH, J.H.; VUYLSTEKE, D.; ORTIZ, R. Perspectives on the application of biotechnology to assist the genetic enhancement of plantain and banana (Musa spp.). Electronic Journal of Biotechnology, v.1, p.11-22, 1998.
- CRUZ, C.D. Programa Genes: versão Windows. Viçosa: UFV, 2001. 642p.
- DECROOCQ, V.; HAGEN, L.S.; FAVÉ, M.G.; EYQUARD, J.P.; PIERRONNET, A. Microsatellite markers in the hexaploid Prunus domestica species and parentage lineage of three European plum cultivars using nuclear and chloroplast simple-sequence repeats. Molecular Breeding, v.13, p.135-142, 2004.
- DOYLE, J.J.; DOYLE, J.L. Isolation of plant DNA from fresh tissue. Focus, v.12, p.13-15, 1990.
- FACHINELLO, J.C.; MUSACCHI, S.; ZUCCHERELLI, S. Polimorfismo enzimático em tecido de pereira. Pesquisa Agropecuária Brasileira, v.32, p.1427-1432, 2000.
- HOWELL, E.C.; NEWBURY, H.J.; SWENNEN, R.L.; WITHERS, L.A.; FORD-LLOYD, B.V. The use of RAPD for identifying and classifying Musa germplasm. Genome, v.37, p.328-332, 1994.
- KAEMMER, D.; FISCHER, D.; JARRET, R.L.; BAURENS, F.C.; GRAPIN, A.; DAMBIER, D.; NOYER, J.L.; LANAUD, C.; KAHL, G.; LAGODA, P.J.L. Molecular breeding in genus Musa: a strong case for STMS marker technology. Euphytica, v.96, p.49-63, 1997.
- LOMBARD, V.; BARI, C.P.; DUBREUIL, P.; BLOUET, F.; ZHANG, D. Potential use of AFLP markers for the distinction of rapeseed cultivars. In: INTERNATIONAL RAPESEED CONGRESS, 20., Canberra, 1999. [Electronic proceedings]. Gosford: Regional Institute, 1999. Disponível em: <http://www.regional.org.au/au/gcirc/4/587.htm>. Acesso em: 28 dez. 2005.
- MOREIRA, R.S. Banana - teoria e prática de cultivo 2.ed. São Paulo: Fundação Cargill, 1999. CD-ROM.
- PADILHA, L.; GUIMARÃES, C.T.; PAIVA, E. Avaliação da pureza genética em sementes de milho utilizando marcadores microssatélites Sete Lagoas: Embrapa Milho e Sorgo, 2003. 3p. (Embrapa Milho e Sorgo. Circular técnica, 30).
- PADILHA, L.; GUIMARÃES, C.T.; VIEIRA, M.G.G.C.; CRESTE, I.R.P.; PARENTONI, S.N.; PACHECO, C.A.P.; SANTOS, M.X.; GAMA, E.E.G.; PAIVA, E. Microssatélites fluorescentes na diferenciação de linhagens de milho. In: CONGRESSO NACIONAL DE MILHO E SORGO, 24., 2002, Florianópolis. Anais Florianópolis: ABMS, 2002. p-1-5. Disponível em: <http://www.abms.org.br/resumo37.doc>. Acesso em: 9 ago. 2004.
- PAZ, O.P. Caracterização de germoplasma de bananeira com RAPD 2000. 69p. Dissertação (Mestrado) - Universidade Federal da Bahia, Cruz das Almas.
- PILLAY, M.; NWAKANMA, D.C.; TENKOUANO, A. Identification de RAPD markers linked to A and B genome sequences in Musa L. Genome, v.43, p.763-767, 2000.
- PRIOLLI, R.H.G.; MENDES-JUNIOR, C.T.; ARANTES, N.E.; CONTEL, E.P.B. Characterization of Brazilian soybean cultivars using microsatellite markers. Genetics and Molecular Biology, v.25, p.185-193, 2002.
- RAMOS, N.P. Determinação da pureza varietal em lotes de sementes de milho através de marcadores morfológico e microssatélites 2004. 104p. Tese (Doutorado) - Escola Superior de Agricultura Luiz de Queiroz, Piracicaba.
- SAWAZAKI, H.E.; BARBOSA, W.; COLOMBO, C.A. Caracterização e identificação de cultivares e seleções de pereiras através de marcadores RAPD. Revista Brasileira de Fruticultura, v.24, p.447-452, 2002.
- SOUZA, S.A.C.D. de. Avaliação da variabilidade genética em Musa spp. utilizando marcadores microssatélites 2002. 86p. Tese (Doutorado) - Escola Superior de Agricultura Luiz de Queiroz, Piracicaba.
- STATSOFT INC. (Tulsa, Estados Unidos). Statistica for Windows v.6.0: computer program manual. Tulsa, 2005. Disponível em: <http://www.statsoft.com/textbook/stathome.html>. Acesso em: 12 nov. 2005.
- TESTOLIN, R.; MARRAZZO, T.; CIPRIANI, G.; QUARTA, R.; VERDE, I.; DETTORI, M.T.; PANCALDI, M.; SANSAVINI, S. Microsatellite DNA in peach (Prunus persica L. Batsch) and its use in fingerprinting and testing the genetic origin of cultivars. Genome, v.43, p.512-520, 2000.
- THIS, P.; CUISSET, C.; BOURSIQUOT, J.M. Development of stable RAPD makers for identification of grapevine rootstocks and the analysis of genetic relationships. American Journal of Enology and Viticulture, v.48, p.492-501, 1997.
- WILLIAMS, J.G.K.; KUBELIK, A.R.; LIVAK, K.J. DNA polymorphisms amplified by arbitrary primers are useful as genetic markers. Nucleic Acids Research, v.18, p.6531-6535, 1990.
- WÜNSCH, A.; HORMAZA, J.I. Cultivar identification and genetic fingerprinting of temperate fruit tree species using DNA markers. Euphytica, v.125, p.59-67, 2002.
Datas de Publicação
-
Publicação nesta coleção
07 Fev 2007 -
Data do Fascículo
Dez 2006
Histórico
-
Recebido
08 Fev 2006 -
Aceito
28 Jul 2006