Resumo
Although in recent years there has been an increasing amount of literature on nanotechnology and their clinical applications, it is still scarce a deep understanding of the interactions at the molecular levels between nanoparticles and cells. Studies demonstrating the underlying mechanisms of nanoparticles endocytosis, intracellular trafficking, and cellular processing are imperative to understand better how cells interact with those materials and their possible undesired effects, e.g. nanotoxicity. The rising awareness concerning nanoparticles applications and its interactions with the cellular environment is part of the new research field called Nanotoxicology. The cumulative knowledge in nanotoxicology will allow us to foresee toxic effects, establish regulations and limits for nanoparticles applications. In this work, we discuss the theoretical concepts about studying endocytosis and intracellular trafficking of nanoparticles. The nanoparticles-cell interactions are a multi-step process, which can be divided into nanoparticles' internalization, intracellular processing and triggering effects of nanomaterials on eukaryotic cells. Finally, we discuss the main techniques used to study this process: flow cytometry, use of endocytosis inhibitors and confocal microscopy.
Keywords:
endocytosis; intracellular trafficking; nanoparticles, nanotoxicology; nano-cell interactions
Keywords:
endocytosis; intracellular trafficking; nanoparticles, nanotoxicology; nano-cell interactions
INTRODUÇÃO
Nanotecnologia é um dos ramos mais promissores de tecnologia, sobre o qual se deposita grandes expectativas.11 Martins, M. A.; Trindade, T.; Quim. Nova 2012, 35, 1434. Somente em 2015, foram publicados cerca de 9400 artigos relacionados ao termo "Nanotechnology" no banco de dados Science Direct; além disso, diversos produtos têm sido testados e comercializados, por exemplo, protetores solares, cosméticos, produtos biomédicos, biosensores e sistemas para transporte e entrega de fármacos.22 Perugini, P.; Simoni, S.; Scalia, S.; Genta, I.; Modena, T.; Conti, B.; Pavanetto, F.; Int. J. Pharm. (Amsterdam, Neth.) 2002, 246, 37. Entretanto, há uma crescente preocupação com a segurança e possíveis efeitos nocivos de nanomateriais que vêm sendo desenvolvidos,33 Brown, D. M.; Wilson, M. R.; MacNee, W.; Stone, V.; Donaldson, K.; Toxicol. Appl. Pharmacol. 2001, 175, 191. pois dados e regulamentações internacionais referentes à segurança e uso desses materiais ainda são escassos, ainda estão em debate ou em fase de implementação.44 Asharani, P. V.; Lian Wu, Y.; Gong, Z.; Valiyaveettil, S.; Nanotechnology 2008, 19, 255102. Uma nova área da ciência destinada ao estudo de efeitos adversos de nanomateriais foi recentemente criada, a Nanotoxicologia.55 Arora, S.; Rajwade, J. M.; Paknikar, K. M.; Toxicol. Appl. Pharmacol. 2012, 258, 151. O conhecimento acumulado nessa área tem como objetivo prever efeitos tóxicos e auxiliar nas regulamentações e diretrizes para aplicação da nanotecnologia66 Oberdörster, G.; Oberdörster, E.; Oberdörster, J.; Environ. Health Perspect. 2005, 113, 823; Franchi, L. P.; Santos, R. A.; Matsubara, E. Y.; de Lima, J. C.; Rosolen, J. M.; Takahashi, C. S.; Quim. Nova 2012, 35, 571. e uso de produtos a ela relacionados. A Figura 1 mostra o crescimento do número de artigos científicos que contêm o termo "nanotoxicology" e estão no banco de dados do National Center for Biotechnology Information (NCBI), subdivisão do National Institute of Health (NIH/USA).
Crescimento do número de artigos científicos publicados, rela cionados ao termo "nanotoxicology" normalizado a cada 10 mil artigos publicados no website "PubMed" (http://www.ncbi.nlm.nih.gov/pubmed), parte do National Center for Biotechnology Information (NCBI), subdivisão do National Institute of Health (NIH/USA), entre os anos 2000 e 2015. Pesquisa realizada em 02/05/2016
Apesar do número de artigos em nanotoxicologia ser crescente, esta é uma área ainda muito recente (com menos de uma década), e ainda não é possível determinar exatamente quais impactos os sistemas naturais e artificiais com dimensões nanométricas podem causar ao ser humano e ao meio ambiente.33 Brown, D. M.; Wilson, M. R.; MacNee, W.; Stone, V.; Donaldson, K.; Toxicol. Appl. Pharmacol. 2001, 175, 191.,77 Paschoalino, M. P.; Marcone, G. P. S.; Jardim, W. F.; Quim. Nova 2010, 33, 421; Dimer, F. A.; Friedrich, R. B.; Beck, R. C. R.; Guterres, S. S.; Quim. Nova 2013, 36, 1520. Apesar de todo conhecimento acumulado nesse período, ainda é difícil fazer generalizações sobre a biodisponibilidade, biodistribuição, degradação, eliminação e atividade biológica de nanoestruturas, bem como sobre os processos moleculares envolvidos em sua internalização, tráfego e processamento celular.44 Asharani, P. V.; Lian Wu, Y.; Gong, Z.; Valiyaveettil, S.; Nanotechnology 2008, 19, 255102.,88 Cancino, J.; Marangoni, V. S.; Zucolotto, V.; Quim. Nova 2014, 37, 521.
Portanto, o conhecimento sobre como as células lidam e interagem com estes materiais é parte essencial para avanços dessa nova área.99 Stern, S. T.; Adiseshaiah, P. P.; Crist, R. M.; Part. Fibre Toxicol. 2012, 9, 1. Estudos que demonstram como nanopartículas são internalizadas, processadas (ex. degradadas) pelas células e as respostas celulares desencadeadas são imperativos para estabelecer procedimentos de segurança na fabricação e nas aplicações comerciais e médicas de nanomateriais.1010 Sahu, S. C.; Casciano, D. A.; Nanotoxicity: from in vivo and in vitro models to health risks, 1st ed., Wiley: Wiltshire, 2009. Para atingir esse objetivo, os primeiros passos devem envolver estudos moleculares sobre interações nanopartículas-células, endocitose, tráfego intracelular e resposta celular a estes materiais.
Por que estudar a endocitose, o tráfego e o processamento intracelular de nanopartículas?
Formas de interação nanopartículas-células
A membrana plasmática é a principal interface celular, através da qual o interior da célula se comunica com o meio externo.1111 Ivanov, A. I.; Exocytosis and Endocytosis, 1st ed., Springer: New York, 2008. O primeiro contato da célula com qualquer material extracelular é através de seus componentes lipídicos (ex. fosfolipídios) e proteicos (ex. receptores membranares).1212 Alberts, B.; Johnson, A.; Lewis, J.; Raff, M.; Roberts, K.; Walter, P.; Molecular Biology of the Cell, 5th ed., Garland Science: New York, 2007. Sendo assim, um dos primeiros testes para avaliar a interação dos nanomaterias com células eucarióticas é avaliar a integridade da membrana. Para isso é possível dosar a presença ou atividade de enzimas intracelulares (ex. lactato desidrogenase)1313 Marquis, B. J.; Love, S. A.; Braun, K. L.; Haynes, C. L.; RSC Adv. 2009, 134, 425. ou testar a integridade da membrana com moléculas que não a permeiam (ex. azul de tripano ou iodeto de propídio).1414 Nogueira, D. R.; Mitjans, M.; Rolim, C. M. B.; Pilar Vinardell, M.; Nanomaterials 2014, 4, 454. Por exemplo, Carlson e colaboradores1515 Carlson, C.; Hussain, S. M.; Schrand, A. M.; K. Braydich-Stolle, L.; Hess, K. L.; Jones, R. L.; Schlager, J. J.; J. Phys. Chem. B 2008, 112, 13608. demonstraram que nanopartículas de prata (diâmetro de 15 a 55 nm) causaram danos à membrana, os autores avaliaram o extravasamento da enzima lactato desidrogenase para o meio extracelular, levando macrófagos à apoptose em menos de 24 h. Os autores sugeriram que a perda da integridade de membrana foi responsável pela morte celular.
Nanopartículas podem acessar o citoplasma de células eucarióticas, graças a danos causados à membrana, ou sendo internalizadas pelas células. Para discernir entre essas possibilidades, é importante avaliar os efeitos observados em condições normais e compará-los a resultados obtidos à 4 ºC,1616 Dijkstra, J.; van Galen, W. J. M.; Hulstaert, C. E.; Kalicharan, D.; Roerdink, F. H.; Scherphof, G. L.; Exp. Cell Res. 1984, 150, 161. ou em presença de inibidores do metabolismo energético (ex. cloreto de amônia combinado com 2-desoxiglicose).1717 Bhattacharjee, S.; Ershov, D.; Fytianos, K.; van der Gucht, J.; Alink, G. M.; Rietjens, I. M. C. M.; Marcelis, A. T. M.; Zuilhof, H.; Part. Fibre Toxicol. 2012, 9, 1. Experimentos in vitro à 4 ºC, bem como inibidores do metabolismo energético, diminuem a produção de ATP, portanto, inibem o processo de endocitose, responsável pela internalização das nanopartículas.1818 Saraste, J.; Palade, G. E.; Farquhar, M. G.; Cell 1986, 83, 6425. O comprometimento da produção de energia, pela diminuição da temperatura ou por agentes que reduzem a produção de ATP; podem trazer informações importantes sobre interações nanosistemas-célula. Assim, efeitos em decorrência de danos causados à membrana seriam observados em ambas as situações (controle e experimentos à 4 ºC ou em presença de inibidores do metabolismo energético), enquanto que efeitos decorrentes da internalização dos nanomateriais seria drasticamente diminuído em baixas temperaturas ou na presença do inibidor.
Além do inibidores do metabolismo energético, é possível utilizar um inibidor de endocitose generalizado, atualmente o mais utilizado é o Dynosore, que pressupõe inibir a dinamina, uma GTPase da qual depende a maioria das vias de endocitose.1111 Ivanov, A. I.; Exocytosis and Endocytosis, 1st ed., Springer: New York, 2008. Comparando os resultados destes experimentos com os obtidos sem o uso desse inibidor, obtém- se os primeiros indícios de que as partículas são ou não endocitadas pelas células. Os detalhes dessa estratégia serão apresentados por metodologias a seguir.
Endocitose
Um dos mais importantes mecanismos de comunicação celular com o meio externo envolve o fluxo transmembranar e bidirecional de vesículas, este fenômeno é o movimento de internalização de conteúdos extracelulares, denominado endocitose.1111 Ivanov, A. I.; Exocytosis and Endocytosis, 1st ed., Springer: New York, 2008. Esta pode ser dividida em fagocitose (somente para algumas células especializadas, como macrófagos) e pinocitose (presente virtualmente em todas as células eucaróticas) a qual, por sua vez, pode ser subdividida em endocitose mediada por clatrina, endocitose mediada por caveolina e macropinocitose. Atualmente outras vias têm sido propostas, como endocitose independente de clatrina e caveolina.1212 Alberts, B.; Johnson, A.; Lewis, J.; Raff, M.; Roberts, K.; Walter, P.; Molecular Biology of the Cell, 5th ed., Garland Science: New York, 2007. Para mais detalhes destes mecanismos, consultar a referência.1919 Ferreira, L. A. B.; Radaic, A.; Pugliese, G. O.; Valentini, M. B.; Oliveira, M. R.; Jesus, M. B.; Acta Farmacêutica Portuguesa 2014, 3, 149.
Estudos têm mostrado que o formato, tamanho, características (como porosidade) da superfície, carga superficial e composição de nanopartículas influenciam diretamente a endocitose.2020 Champion, J. A.; Katare, Y. K.; Mitragotri, S.; Proc. Natl. Acad. Sci. U. S. A. 2007, 104, 11901. De fato, é sabido que, no caso de entrega seletiva de ativos às células, são acopladas moléculas às nanopartículas intencionalmente que interagem com receptores específicos da superfície celular. Isso evita danos às células que não expressam esses receptores ou o fazem em menor número. Mas, apesar das propriedades físico-químicas dos nanomateriais influenciarem a sua via de internalização, um mesmo material pode ser internalizados por vias diferentes em diferentes linhagens celulares, demonstrando que esse é um processo célula-dependente.2121 Doherty, G. J.; McMahon, H. T.; Annu. Rev. Biochem. 2009, 78, 857.
A análise da endocitose em células vivas (tanto in vitro quanto in vivo) é normalmente feita com inibidores químicos (farmacológicos) de endocitose,2222 Ivanov, A. I. Em Pharmacological Inhibition of Endocytic Pathways: Is It Specific Enough to Be Useful?; Ivanov, A. I., ed.; Humana Press: New York, 2008, cap. 2; Vercauteren, D.; Vandenbroucke, R. E.; Jones, A. T.; Rejman, J.; Demeester, J.; De Smedt, S. C.; Sanders, N. N.; Braeckmans, K.; Mol. Ther. 2010, 18, 561. células mutantes que têm algumas proteínas das principais vias de endocitose não funcionais2323 Benmerah, A.; Bayrou, M.; Cerf-Bensussan, N.; Dautry-Varsat, A.; J. Cell Sci. 1999, 112), 1303; Damke, H.; Baba, T.; Warnock, D. E.; Schmid, S. L.; J. Cell Biol. 1994, 12, 915; Hsueh, P.-Y.; Edman, M. C.; Sun, G.; Shi, P.; Xu, S.; Lin, Y.-A.; Cui, H.; Hamm-Alvarez, S. F.; MacKay, J. A.; J. Controlled Release 2015, 208, 2. ou, ainda, células cujas proteínas chave das vias de endocitose têm sua expressão diminuída por ferramentas de biologia molecular (ex. RNA de interferência).2424 van den Broeck, D.; de Wolf, M. J. S.; Biotechnology 2006, 41, 475.
Inibidores são ferramentas valiosas para determinar a via de endocitose utilizada, pois são simples, confiáveis e de custo acessível.1111 Ivanov, A. I.; Exocytosis and Endocytosis, 1st ed., Springer: New York, 2008. Entretanto, é necessário que os experimentos sejam realizados cuidadosamente, como discutido a seguir, e suportados por outros experimentos (ex. microscopia confocal).2222 Ivanov, A. I. Em Pharmacological Inhibition of Endocytic Pathways: Is It Specific Enough to Be Useful?; Ivanov, A. I., ed.; Humana Press: New York, 2008, cap. 2; Vercauteren, D.; Vandenbroucke, R. E.; Jones, A. T.; Rejman, J.; Demeester, J.; De Smedt, S. C.; Sanders, N. N.; Braeckmans, K.; Mol. Ther. 2010, 18, 561. Uma vez confirmada a internalização do nanomaterial, o próximo passo é avaliar a translocação do material exógeno para o destino específico na célula, processo denominado de tráfego intracelular.
Tráfego intracelular
Após internalização, independentemente da via utilizada, a partícula é englobada em uma vesícula (e.g., endossomo), sem acesso direto ao citosol ou a organelas citoplasmáticas e, portanto, fica impedida de exercer sua função biológica, particularmente crítico para carreamento de material genético, ou de fármacos que têm alvos intracelulares.1111 Ivanov, A. I.; Exocytosis and Endocytosis, 1st ed., Springer: New York, 2008.
Caso não aconteça o escape do endossomo, os materiais endocitados podem: i. ser degradados nos lisossomos; ii. redirecionados para o meio exterior através de endossomos de reciclagem; iii. encaminhados para outras organelas; ou iv. exocitados.2525 Segev, N.; Alfonso, A.; Payne, G. S.; Donaldson, J.; Trafficking Inside Cells - Pathways, Mechanisms and Regulation, 1st ed., Landes Bioscience: New York, 2010. A literatura mostra que a maior parte das nanopartículas, após internalização, são direcionadas para os lisossomos, nos quais são degradadas.2626 Behr, J. P.; Chimia 1997, 51, 34.
A degradação lisossomal pode limitar a aplicação de diversos nanomateriais, em especial daqueles que carreiam biomoléculas, como oligonucleotídeos (DNA, RNA, entre outros). Portanto, o estudo das estratégias de escape endossomal pode auxiliar na otimização de aplicações terapêuticas envolvendo nanopartículas.2727 Martens, T. F.; Remaut, K.; Demeester, J.; De Smedt, S. C.; Braeckmans, K.; Nano Today 2014, 9, 344.
Em geral, a formação de complexos de DNA com lipossomas catiônicos tem como motor propulsor um grande aumento de entropia, dada a liberação de contra-íons para a solução. Sendo assim, a simples mistura de DNA e lipossomas catiônicos na solução leva a reorganização de suas estruturas formando agregados em múltiplas camadas podendo estar organizadas no estado líquido-cristalinas a depender, entre outras coisas, da relação entre as cargas positivas e negativas dos eletrólitos, das características estruturais dos lipossomas (como raio de curvatura), do tamanho do fragmento de nucleotídeo, pH, etc.2828 de Jesus, M. B.; Ferreira, C. V.; de Paula, E.; Hoekstra, D.; Zuhorn, I. S.; J. Controlled Release 2010, 148, e89; Ewert, K.; Evans, H. M.; Ahmad, A.; Slack, N. L.; Lin, A. J.; Martin-Herranz, A.; Safinya, C. R. Em Lipoplex structures and their distinct cellular pathways.; Leaf, H.; Mien-Chie, H.; Earnest, W., eds.; Academic Press: San Diego, 2005, cap. 5.
Lin et al2929 Lin, A. J.; Slack, N. L.; Ahmad, A.; George, C. X.; Samuel, C. E.; Safinya, C. R.; Biophys. J. 2003, 84, 3307. desenvolveram um estudo comparativo em que foram preparados dois complexos entre DNA e lipossomas com estruturas distintas: lamelar (Lcα), utilizando os lipídios DOTAP/DOPC e hexagonal invertida (HIIc) e os lipídios DOTAP/DOPE. Essas estruturas, ao serem colocadas em contato com células de fibroblastos de rato, apresentaram perfis de transfecção distintos. Os complexos com estrutura hexagonal invertida obtiveram eficiência de transfecção aproximadamente 100 vezes superior em comparação com os complexos com estrutura lamelar. Segundo os autores, a fase hexagonal invertida (HIIc) leva a um mecanismo fusão dos lipoplexos com a membrana do endossomo, isso levaria a uma maior liberação do DNA no citoplasma da célula e, consequentemente, resultaria em maiores níveis de transfecção.
Para estudar o papel da acidificação sobre o processamento de um dado nanomaterial é possível empregar estratégias que utilizam ensaios com inibidores da acidificação endossomal, denominados de lisossomotrópicos (ex. cloroquina, bafilomicina, metilaminas e ionóforos carboxílicos) podem ser bastante úteis.3030 de Duve, C.; de Barsy, T.; Poole, B.; Trouet, A.; Tulkens, P.; van Hoof, F.; Biochem. Pharmacol. (Amsterdam, Neth.) 1974, 23, 2495. Inibidores de acidificação endomossoal são bases fracas que, ao entrarem em ambiente ácido, ficam protonadas e reduzem a acidificação endossomal e lisossomal. A não acidificação do endossomo impede a fusão do endossomo com o lisossomo, evitando que enzimas líticas presentes no interior dos lisossomos degradem o material internalizado. Isso pode ser benéfico para nanomateriais que seriam prontamente degradados em ambiente mais ácido, mas pode ser prejudicial para nanomateriais que dependem da acidificação para a liberação de seu conteúdo, ou para desempenhar sua função biológica.
Por exemplo, Behr,2626 Behr, J. P.; Chimia 1997, 51, 34. em 1997, hipotetizou que nanopartículas poliméricas de poli (etilenoimina) (PEI), utilizadas para transfecção, poderiam escapar de endossomos através da ruptura osmótica da membrana endossomal. Esse efeito poderia estar associado com a grande quantidade de grupos amina protonáveis na estrutura do polímero. Neste caso, à medida em que o interior do endossomo vai se tornando mais ácido, até chegar aos valores de pH lisossomais (pH ~5,5), o equilíbrio osmótico desses compartimentos é desfeito, e culmina na ruptura da vesícula com liberação do material carreado.2626 Behr, J. P.; Chimia 1997, 51, 34. Atualmente os detalhes sobre o exato mecanismo associados a esta hipótese estão em debate.3131 Benjaminsen, R. V.; Mattebjerg, M. A.; Henriksen, J. R.; Moghimi, S. M.; Andresen, T. L.; Mol. Ther. 2013, 21, 149.
Para sistemas que não possuem estas propriedades químicas, há possibilidade do uso de moléculas lisossomotrópicas, que inibem a acidificação lisossomal, aumentando o tempo de permanência das partículas em seu interior e com isso as chances de ocorrer a liberação do conteúdo para o citoplasma.3232 Silva, A. T.; Nguyen, A.; Ye, C.; Verchot, J.; Moon, J. H.; BMC Plant Biol. 2010, 10, 1.
TÉCNICAS E ESTRATÉGIAS PARA ESTUDO DA INTERAÇÃO NANOPARTÍCULA-CÉLULAS
Para obter maiores detalhes da interação nanopartícula-células discutiremos a seguir detalhes da aplicação das três técnicas mais utilizadas para estudar esse fenômeno: citometria de fluxo, uso de inibidores de endocitose e microscopia confocal. Para estudos visando a caracterização do nanomaterial, em especial nanopartículas lipídicas sólidas, e técnicas relacionadas sugerimos a leitura da referência3333 Radaic, A.; Barbosa, L. R. S.; Jaime, C.; Kapila, Y. L.; Pessine, F. B. T.; de Jesus, M. B. Em How lipid cores affect lipid nanopaticles as drug and gene delivery systems; Iglic, A.; Kulkarni, C. V.; Rappol, M., eds.; Elsevier: in press, 2016, cap. 1. e para técnicas físicas para estudar a interação nanopartículas-membrana celular, sugerimos a leitura da referência.3434 Tada, D. B.; Suraniti, E.; Rossi, L. M.; Leite, C. A. P.; Oliveira, C. S.; Tumolo, T. C.; Calemczuk, R.; Livache, T.; Baptista, M. S.; J. Biomed. Nanotechnol. 2014, 10, 519.
Citometria de fluxo
Citometria de fluxo é uma técnica que permite avaliar individualmente células e até mesmo partículas de naturezas diversas,3535 Givan, A. L. Em Flow Cytometry: An Introduction; Hawley, T. S.; Hawley, R. G., eds.; Humana Press: New York, 2004, cap. 1. através da focalização hidrodinâmica em um capilar. Ao passar pelo capilar, as células são irradiadas por lasers que excitam diferentes fluoróforos, possibilitando análises bastante detalhadas de eventos celulares.3636 Pozarowski, P.; Holden, E.; Darzynkiewicz, Z.; Methods in Molecular Biology, 1st ed., Humana Press: New York, 2013.
Para isso, são usados marcadores específicos, como anticorpos ligados a fluoróforos ou fluoróforos que têm afinidade por estruturas celulares específicas ou respondem a processos celulares (ex. a sonda DCFDA - 2',7'-diclorofluoresceína diacetato - que responde a formação de espécies reativas de oxigênio), e possibilitam a análise qualitativa e quantitativa de eventos celulares, por exemplo ciclo celular, estresse oxidativo, fosforilação de proteínas, apoptose, etc. Análises corriqueiras podem ser realizadas com um a três fluoróforos e testes mais avançados chegam a combinar até 18 fluoróforos. Quando acoplada à espectrometria de massas, esse número pode passar de 36.3737 Bendall, S. C.; Nolan, G. P.; Roederer, M.; Chattopadhyay, P. K.; Trends Immunol. 2012, 33, 323. Em geral, citômetros de fluxo descartam as amostras após a análise, ainda que alguns possam utilizar a técnica de separação de células cuja fluorescência foi previamente ativada (Fuorescence-Activated Cell Sorting, FACS) permitindo a coleta e análise de informações sobre subpopulações, separadamente. Nesse caso, as células das subpopulações podem ser cultivadas isoladamente, ampliando o leque de análises possíveis.3838 Barnard, R. M.; Bioanalysis 2012, 4, 2471.
A citometria de fluxo permite analisar milhares de células por segundo e obter resultados dependentes das propriedades intrínsecas das amostras, como tamanho das células, granulosidade, intensidade da fluorescência, etc. Pode ser empregada também para estudar eventos celulares como a eficiência de transfecção de um plasmídeo contendo um gene codificante para uma proteína modelo, usualmente eGFP (enhanced Green Fluorescent Protein), transportado por nanopartículas. Isso pode ser medido avaliando a frequência de células que respondem positivamente à excitação do laser no comprimento de onda de fluorescência da proteína, com excitação em 488 nm e emissão 509 nm. A análise de 10 mil células é considerada padrão para a maioria dos testes.3939 Cunningham, R. E. Em Overview of Flow Cytometry and Fluorescent Probes for Flow Cytometry; Oliver, C.; Jamur, M. C., eds.; Humana Press: New Jersey, 2009, cap. 31. Entretanto, esse número pode variar dependendo das características de cada amostra. Se, por exemplo, o evento celular estudado é raro, então milhares de células devem ser analisadas, desde que haja um tratamento estatístico adequado ao evento estudado.
A Citometria de fluxo tem se mostrado uma técnica bastante poderosa para investigar eventos celulares, sendo empregada no estudo entre células eucarióticas e procarióticas, como bactérias3232 Silva, A. T.; Nguyen, A.; Ye, C.; Verchot, J.; Moon, J. H.; BMC Plant Biol. 2010, 10, 1. e vírus.4040 McSharry, J. J.; Methods 2000, 21, 249. No processo de internalização celular, é cada vez mais utilizada para estudar interações de células e nanopartículas4141 Ibuki, Y.; Toyooka, T. Em Nanoparticle Uptake Measured by Flow Cytometry; Reineke, J., ed.; Humana Press: New York, 2012, cap. 11. e também propriedades de sistemas nanoparticulados, como tamanho de complexos nanoestruturados que transportam material genético em fluidos biológicos.4242 van Gaal, E. V. B.; Spierenburg, G.; Hennink, W. E.; Crommelin, D. J. A.; Mastrobattista, E.; J. Controlled Release 2010, 141, 328. A seguir será discutido em detalhes como essa técnica pode ser utilizada para estudar as principais etapas do processo de interação entre nanopartículas e células eucarióticas: internalização, tráfego intracelular e resposta biológica.
Internalização: a análise, via citometria de fluxo, da interação de nanomateriais e células eucarióticas pode ser feita de duas maneiras, que fornecem informações complementares: a primeira, quando o material adsorvido à membrana celular é incluído na análise (em inglês referido como total cell association) e, a segunda, quando esse material é excluído da análise (em inglês referido como internalization ou uptake). Em princípio, o material adsorvido deveria ser excluído para que o resultado possa ser correlacionado somente com as partículas internalizadas. Todavia, isso nem sempre é possível, pois, tecnicamente, a exclusão das partículas externas às células nem sempre é possível. Alguns fluoróforos, como os derivados de fluoresceína, podem ter a fluorescência suprimida por outros compostos, como o azul de tripano,4343 Hed, J.; FEMS Microbiol. Lett. 1977, 1, 357. corante incapaz de penetrar células saudáveis. Nesse caso, seu uso para extinguir a fluorescência das partículas aderidas à membrana da célula se limita a ensaios com células vivas, pois se estiverem fixadas, as partículas dentro das células teriam também a fluorescência suprimida.4343 Hed, J.; FEMS Microbiol. Lett. 1977, 1, 357. O emprego de azul de tripano em citometria de fluxo foi validado para o estudo da interação entre leveduras e granulócitos4444 Hed, J.; Hallden, G.; Johansson, S. G. O.; Larsson, P.; J. Immunol. Methods 1987, 101, 119. e, atualmente, é bastante empregado para investigar interações entre nanomateriais e células eucarióticas em geral.4545 Xue, H.-Y.; Wong, H.-L.; ACS Nano 2011, 5, 7034.
Shimoni et al investigaram a internalização de nano-cápsulas de hidrogel de ácido polimetacrílico em células HeLa, concluindo que cápsulas esféricas eram internalizadas em maior quantidade que outras com mesma composição, mas com formato alongado.4646 Shimoni, O.; Yan, Y.; Wang, Y.; Caruso, F.; ACS Nano 2012, 7, 522. Martins et al. demonstraram que nanopartículas lipídicas sólidas apresentavam taxa de internalização preferencial em linhagens de glioma humano (A172 e U87) em relação à macrófagos, fato que aumenta a relevância dessas nanopartículas como carreadores de fármacos anti-neoplásicos.4747 Martins, S.; Costa-Lima, S.; Carneiro, T.; Cordeiro-da-Silva, A.; Souto, E. B.; Ferreira, D. C.; Int. J. Pharm. (Amsterdam, Neth.) 2012, 430, 216. Silva et al empregaram nanopartículas poliméricas para carrear siRNA em protoplastos de tabaco BY-2 e demonstraram que esse processo depende do tempo e da concentração.3232 Silva, A. T.; Nguyen, A.; Ye, C.; Verchot, J.; Moon, J. H.; BMC Plant Biol. 2010, 10, 1. Esses são apenas alguns exemplos da aplicação da citometria de fluxo para estudar a internalização de nanomateriais em células eucarióticas.
Tráfego intracelular: citometria de fluxo não é comumente utilizada para estudos sobre tráfego intracelular de nanopartículas. Contudo, é possível obter informações indiretas, como sobre interação com organelas (via alterações no potencial mitocondrial) ou alterações nas células (via fragmentação do DNA), que podem servir de guia para experimentos mais específicos a serem realizados mediante outras técnicas.
Resposta biológica: após internalização e processamento celular, muitas nanopartículas ativam respostas celulares, que vão desde alterações com efeitos terapêuticos ou de entrega de fármacos e genes, até efeitos citotóxicos como pequenas alterações no citoesqueleto celular, estresse, etc.4848 Jesus, M. B.; Kapila, Y. L. Em Cellular Mechanisms in Nanomaterial Internalization, Intracellular Trafficking, and Toxicity; Durán, N.; Guterres, S. S.; Alves, O. L., eds.; Humana Press: New York, 2014, cap. 9. A citometria é uma técnica estabelecida para estudar eventos intracelulares como estresse oxidativo,4949 Eruslanov, E.; Kusmartsev, S. Em Identification of ROS Using Oxidized DCFDA and Flow-Cytometry; Armstrong, D., ed.; Humana Press: New York, 2010, cap. 4. autofagia,5050 Hansen, T. E.; Johansen, T.; BMC Biol. 2011, 9, 1. alterações no ciclo celular,5151 Pozarowski, P.; Darzynkiewicz, Z. Em Analysis of Cell Cycle by Flow Cytometry; Schönthal, A. H., ed.; Humana Press: New York, 2004, cap. 22. apoptose,5252 Vermes, I.; Haanen, C.; Steffens-Nakken, H.; Reutellingsperger, C.; J. Immunol. Methods 1995, 184, 39. entre outros. Mesmo alterações como fragmentação do DNA celular, antes analisada por eletroforese ou microscopia, também pode ser investigada por citometria de fluxo.
Zhang et al observaram alto nível de fragmentação de DNA, uma característica de apoptose, em tratamentos de 24 h em hepatócitos humanos de Chang e fibroblastos de pulmão de hamster chinês (V79-4).5353 Zhang, R.; Piao, M. J.; Kim, K. C.; Kim, A. D.; Choi, J.-Y.; Choi, J.; Hyun, J. W.; Int. J. Biochem. Cell Biol. 2012, 44, 224.
Uso de inibidores de internalização
Considerando que as várias vias de endocitose de nanomateriais são diretamente relacionadas aos efeitos causados,2121 Doherty, G. J.; McMahon, H. T.; Annu. Rev. Biochem. 2009, 78, 857. é crucial que elas sejam determinadas. O estudo da inibição dessas vias constitui estratégia largamente usada para compreensão desse fenômeno.5454 Subtil, A.; Hémar, A.; Dautry-Varsat, A.; J. Cell Sci. 1994, 107), 3461. A inibição de endocitose pode ser abordada por métodos seletivos e não seletivos para cada tipo de endocitose, mediante emprego de inibidores químicos ou através de regulação da expressão gênica.3232 Silva, A. T.; Nguyen, A.; Ye, C.; Verchot, J.; Moon, J. H.; BMC Plant Biol. 2010, 10, 1. O uso de inibidores químicos é comum na caracterização da via de internalização, principalmente de nanomateriais,5555 Macia, E.; Ehrlich, M.; Massol, R.; Boucrot, E.; Brunner, C.; Kirchhausen, T.; Dev. Cell 2006, 10, 839. como ocorre com carreadores de genes.5656 Vercauteren, D.; Vandenbroucke, R. E.; Jones, A. T.; Rejman, J.; Demeester, J.; De Smedt, S. C.; Sanders, N. N.; Braeckmans, K.; Mol. Ther. 2010, 18, 561. Os protocolos que utilizam estes inibidores são vantajosos, pois são de baixo custo e rápidos. Como exemplos podem ser citados o uso de clorpromazina, que atua na inibição de endocitose mediada por clatrina5757 Callegari, F.; Rossini, G. P.; Toxicology 2008 244, 133. e o de metil-β-ciclodextrina, que atua na inibição de endocitose mediada por flotilina e caveolina.5858 Rodal, S. K.; Skretting, G.; Garred, Ø.; Vilhardt, F.; van Deurs, B.; Sandvig, K.; Mol. Biol. Cell 1999, 10, 961. Outro ponto favorável aos ensaios com inibidores com atividade farmacológica é que eles afetam todas as células em uma população, e os efeitos são facilmente quantificados.1111 Ivanov, A. I.; Exocytosis and Endocytosis, 1st ed., Springer: New York, 2008. A Tabela 1S apresenta uma relação dos inibidores de endocitose mais utilizados, organizados pelas vias de endocitose, além de uma breve descrição de seu mecanismo de ação e concentrações de uso, que podem servir como guia na padronização dos experimentos e a estrutura de cada um dos inibidores.
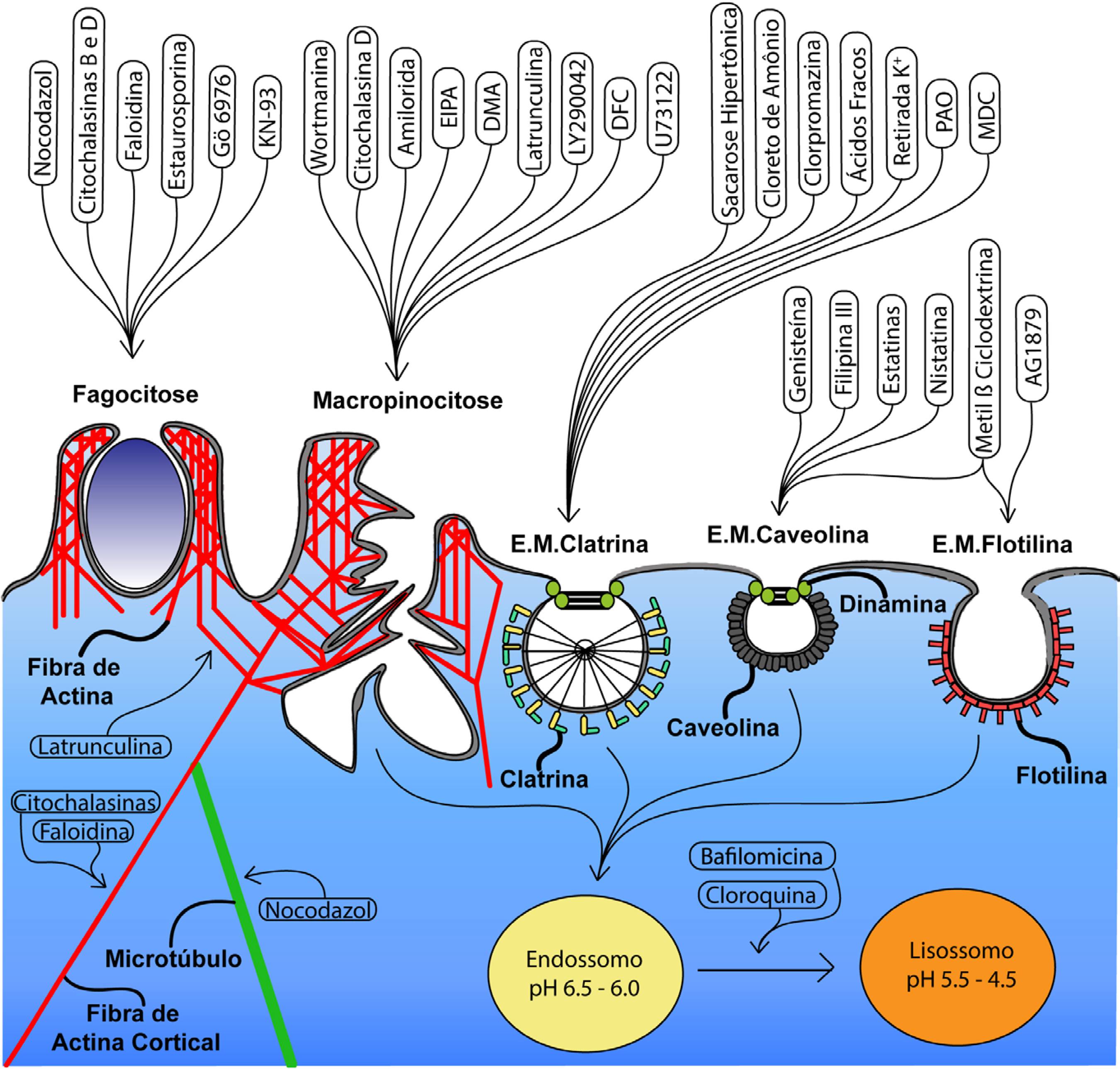
A Figura 2 apresenta uma visão geral dos principais inibidores de endocitose e as respectivas vias em que atuam. Além disso, como o tráfego de vesículas intracelulares no interior das células é mediado por fibras do citoesqueleto (actina ou miosina) representamos moléculas (como latranculina, citocalasinas, faloidina e nocodazol) que atuam no citoesqueleto. Elas são importantes para determinar quais fibras do citoesqueleto são relevantes para o papel biológico de um determinado nanomaterial. Agentes lisossomotrópicos que, em sua maioria, são bases fracas inibidoras da acidificação de vesículas intracelulares, auxiliam também na determinação do papel desse efeito para o papel biológico de um dado nanomaterial. Como discutido acima, alguns nanomateriais dependem da acidificação para desempenhar seu papel biológico.
Local de atuação de inibidores de endocitose nas vias clássicas: Fagocitose, Macropinocitose, Endocitose mediada por Clatrina (E.M. Clatrina), Endocitose mediada por Caveolina (E.M. Caveolina) e Endocitose mediada por Flotilina (E.M. Flotilina) e tráfego intracelular
Precauções no uso de inibidores
Para evitar problemas com a inespecificidade dos inibidores é necessário a padronização de seu uso para cada tipo celular, o primeiro passo é determinar a concentração tóxica do inibidor. A concentração utilizada não pode apresentar efeitos tóxicos significativos, pois nessa condição não é possível distinguir se o efeito observado ocorre devido à inibição da via de interesse ou se é consequência da toxicidade do inibidor.5959 Joshi, P.; Chakraborti, S.; Ramirez-Vick, J. E.; Ansari, Z. A.; Shanker, V.; Chakrabarti, P.; Singh, S. P.; Colloids Surf., B 2012, 95, 195. Via de regra, ensaios de viabilidade padrão (ex. com MTT) devem apresentar, no máximo, 15% de redução de viabilidade, sendo o ideal utilizar concentrações que não apresentam nenhuma toxicidade. Além disso, é importante levar em consideração o tempo de exposição das células aos inibidores. Exposições muito longas (> 8 h, dependendo do inibidor e da célula) devem ser evitadas, pois as células podem metabolizar os inibidores, que perdem seu efeito. Caso sejam encontrados resultados contraditórios deve-se validar a atividade do inibidor naquele determinado tempo.
Cumprida esta etapa, o próximo passo é validar as concentrações utilizadas do inibidor. Faz-se necessário padronizar as concentrações dos inibidores utilizando marcadores específicos de cada via que depende do inibidor, do tipo celular e do tempo de exposição. Os marcadores mais utilizados são a transferrina para endocitose mediada por clatrina,6060 Pozzi, D.; Marchini, C.; Cardarelli, F.; Salomone, F.; Coppola S, M. M., Zabaleta M. E., Digman M. A., Gratton E, Colapicchioni V, Caracciolo G.; Biochim. Biophys. Acta 2014, 1838,957. toxina da cólera para a endocitose mediada por caveolina6161 Wittrup, A.; Zhang, S.-H.; Svensson, K. J.; Kucharzewska, P.; Johansson, M. C.; Mörgelin, M.; Belting, M.; Proc. Natl. Acad. Sci. U. S. A. 2010, 107, 13342. e dextrana para macropinocitose.6262 Bang, C.; Batkai, S.; Dangwal, S.; Gupta, S. K.; Foinquinos, A.; Holzmann, A.; Just, A.; Remke, J.; Zimmer, K.; Zeug, A.; Ponimaskin, E.; Schmiedl, A.; Yin, X.; Mayr, M.; Halder, R.; Fischer, A.; Engelhardt, S.; Wei, Y.; Schober, A.; Fiedler, J.; Thum, T.; J. Clin. Invest. 2014, 124, 2136. Os marcadores geralmente são disponíveis comercialmente já associados a um fluoróforo. A validação da concentração usada do inibidor pode ser obtida através de citometria de fluxo (quantitativo) ou microscopia confocal (qualitativo). Neste caso, determina-se a concentração de inibidor capaz de causar redução significativa da internalização do marcador de cada via. Para auxiliar na determinação das concentrações iniciais de inibidor compilamos as concentrações mais utilizadas na literatura (Tabela 1S). Caso as células de interesse sejam diferentes daquela utilizadas nos trabalhos citados, será necessária uma busca em literatura pela célula específica.
Por fim, é importante ressaltar que células em geral utilizam mais de uma via para internalização de moléculas ou nanoestruturas. Em presença de inibidor, a célula pode apresentar um efeito compensatório, utilizando outra via endocítica para suprir a internalização. Isso pode, por exemplo, aumentar a resposta biológica (ex. transfecção ou citotoxicidade) e não diminuí-la, como poderia ser pensado inicialmente,6363 Rejman, J.; Bragonzi, A.; Conese, M.; Mol. Ther. 2005, 12, 468. ou, então, suprimir parcialmente as respostas biológicas frente ao uso de inibidor.6363 Rejman, J.; Bragonzi, A.; Conese, M.; Mol. Ther. 2005, 12, 468.,6464 Damke, H.; Baba, T.; van der Bliek, A. M.; Schmid, L.; J. Cell Biol. 1995, 131, 69.
Microscopia confocal
A observação direta de nanomateriais exige o uso de microscópios e por isso a microscopia tem se tornado a técnica indispensável em nanotecnologia. Diversos tipos de microscopia são usadas para caracterizar nanopartículas: microscopia eletrônica de varredura, microscopia eletrônica de transmissão, microscopia de varredura por tunelamento, microscopia por difração de elétrons, microscopia de força atômica, microscopia de fluorescência (em suas várias modalidades), microscopia confocal Raman, etc.6565 Rao, A.; Schoenenberger, M.; Gnecco, E.; Glatzel, T.; Meyer, E.; Brändlin, D.; Scandella, L.; J. Phys.: Conf. Ser. 2007, 61, 971.
Em particular, a microscopia de fluorescência vem sendo empregada em investigações sobre interações entre nanopartículas e meios biológicos. A evolução dessa técnica nas últimas décadas originou diversas variantes da mesma. Dentre as mais importantes há a microscopia de varredura confocal à laser, microscopia de fluorescência de 2 fótons, microscopia confocal com super-resolução óptica com depleção via emissão estimulada (STED), microscopia de fluorescência em campo e com reflexão interna total (TIRF) entre outras.6666 Haustein, E.; Schwille, P.; HFSP J. 2007, 1, 169. As técnicas mais avançadas de microscopia de fluorescência possibilitam elucidar processos celulares em nível molecular. Com maior poder de resolução revelam, com resolução espacial e temporal, detalhes do processamento de nanomateriais por células eucarióticas. Algumas técnicas baseadas em transferência de energia de ressonância por fluorescência (FRET), tempo de vida de fluorescência, medidas de grau de anisotropia, de variação temporal do grau de anisotropia e espectroscopia de correlação de fluorescência têm alcançado limite de resolução abaixo de 30 nm, aproximando o poder de resolução ao da microscopia eletrônica (0,1 nm). Entre as vantagens adicionais podem ser citadas: técnica não destrutiva, menos laboriosa, menos dispendiosa, necessidade de uma menor quantidade de amostra e possibilidade de analisar amostras vivas.6666 Haustein, E.; Schwille, P.; HFSP J. 2007, 1, 169.
A microscopia de fluorescência é fundamentada na emissão de luz por um fluoróforo endógeno ou exógeno da amostra e tem se tornado bastante comum, em virtude também da facilidade de manuseio do equipamento. Todavia, o preparo, marcação, análise e processamento das imagens devem ser feitos com o devido cuidado para não levar a interpretações equivocadas dos resultados. A análise dos dados obtidos por microscopia pode ser feita qualitativa e quantitativamente, e é fundamental o conhecimento do sistema para que essa análise seja feita corretamente.6767 North, A. J.; J. Cell Biol. 2006, 172, 9. Além disso, as limitações intrínsecas de cada uma dessas técnicas devem ser sempre ser consideradas. Por exemplo, microscópios de epifluorescência não permitem concluir se nanopartículas estão dentro ou fora de células.
A marcação utilizada em microscopia de fluorescência é semelhante à da citometria. Proteínas de interesse podem ser etiquetadas através de técnicas de biologia molecular como associação com proteínas fluorescentes (GFP, RFP, BFP) ou com anticorpos fluorescentes (ou, ainda, um anticorpo secundário fluorescente). Assim, estruturas e organelas celulares podem ser identificadas juntamente com a nanopartícula de interesse. A Tabela 1 apresenta um resumo das principais proteínas e marcadores fluorescentes utilizados para identificação de organelas e vesículas intracelulares. É pré-requisito que essas proteínas ou estruturas reconhecidas na identificação de vesículas ou organelas não deixem a estrutura de origem, mesmo após a fixação do material estudado. A mesma regra deve ser aplicada à marcação de nanopartículas, sendo imperativo garantir que, mesmo após a fixação, o material e o fluoróforo utilizado para marcação não se separem. Para isso, quando o nanomaterial não apresenta fluorescência intrínseca, é sempre desejável que o fluoróforo esteja ligado por ligação covalente; fluoróforos que têm coeficiente de partição com nanomaterial não devem ser utilizados. A marcação de organelas como núcleo, mitocôndria e lisossomos pode ser feita com moléculas pequenas que exibem interações específicas com essas estruturas (Tabela 1). Os marcadores fluorescentes de núcleos de células vivas e fixadas são empregados por serem confiáveis, práticos e mais baratos que anticorpos. É preciso destacar que alguns desses marcadores podem interferir na proliferação celular, pois muitos deles interagem diretamente com o DNA. Em experimentos de longa duração com células vivas são empregadas por exemplo histonas, proteínas responsáveis pelo empacotamento do DNA no núcleo, associadas a proteínas fluorescentes (ex. GFP).
A seguir serão apresentados exemplos de aplicações da microscopia de florescência no estudo das principais etapas do processo de interação de nanopartículas com células eucarióticas: internalização, tráfego intracelular e resposta biológica.
Internalização: são frequentes estudos de colocalização entre partículas marcadas e alguns marcadores ou proteínas envolvidas com o processo de internalização.6868 Fujimoto, K.; Kitamura, M.; Yokokawa, M.; Kanno, I.; Kotera, H.; Yokokawa, R.; ACS Nano 2013, 7, 447; Fichter, K. M.; Ingle, N. P.; McLendon, P. M.; ACS Nano 2013, 7, 347; Vranic S.; Boggetto N.; Contremoulins V.; Mornet S.; Reinhardt N.; Marano F.; Baeza-Squiban A.; S., B.; Part. Fibre Toxicol. 2013, 10, 2; Kasper J.; Hermanns M. I.; Bantz C.; Koshkina O.; Lang T.; Maskos M.; Pohl C.; Unger R. E.; Kirkpatrick C. J.; Arch. Toxicol. 2013, 87, 1052. Tais estudos, juntamente com o uso de inibidores, são fundamentais para demonstrar a via de internalização de nanomateriais. A internalização via clatrina geralmente leva nanomateriais à degradação lisossomal. Isso pode ser utilizado para racionalizar a entrega intracelular de fármacos ou ativos. Por exemplo, em um estudo sobre internalização de nanopartículas inorgânicas lamelares contendo metotrexato (fármaco antagonista de ácido fólico, utilizado no tratamento de algumas neoplasias malignas), internalizadas preferencialmente por clatrina em células de osteosarcoma (linhagem MNNG/HOS), foi sugerido que esta seja uma via especialmente eficiente para o transporte de quimioterápicos. Isso porque a diminuição do pH, característico do tráfego intracelular, leva à liberação gradual do quimioterápico no interior das células, graças à possibilidade da troca de íons localizados na região interlamelar dessas nanopartículas.6969 Oh, J.-M.; Choi, S.-J.; Kim, S.-T.; Choy, J.-H.; Bioconjugate Chem. 2006, 17, 1411. Em contrapartida, nanopartículas poliméricas altamente compactas, carreando DNA, apresentaram internalização mediada por caveolina, em células epiteliais de via respiratória humana (BEAS-2B), como confirmado pela alta colocalização com a toxina da cólera. Essa internalização resultou em processamento mais lento quando comparado com nanopartículas de poliestireno havendo, ainda, acúmulo na região perinuclear, benéfica para entrega de DNA.3232 Silva, A. T.; Nguyen, A.; Ye, C.; Verchot, J.; Moon, J. H.; BMC Plant Biol. 2010, 10, 1. Estudos sobre colocalização são vastamente empregados na caracterização da via de internalização de nanomateriais. Em sua maioria, esses experimentos adotam duas estratégias: colocalizar as nanopartículas com i. proteínas envolvidas na endocitose (ex. clatrina, caveolina), ou ii. com moléculas que são sabidamente internalizadas por uma via específica associadas a fluoróforos, como transferrina, para endocitose mediada por clatrina, toxina da cólera para endocitose mediada por caveolina e dextrana para endocitose por macropinocitose, para mais exemplos vide Tabela 1.
Tráfego intracelular: após internalização é importante avaliar o tráfego intracelular das nanopartículas. Apesar de fortemente influenciado pela via de internalização, nem sempre é possível prever o destino intracelular de nanopartículas. É possível que elas escapem dos endossomos ao citosol ou que seu acúmulo intracelular as leve à degradação e/ou exocitose. Os vários tipos de microscopia de fluorescência constituem as técnicas mais poderosas para investigar o tráfego intracelular de nanopartículas. Porém, neste caso é necessário considerar que ocorre acidificação dos endossomos, como já mencionado, o que pode alterar as características da fluorescência. Por exemplo, a proteína fluorescente eGFP, uma das variantes mais utilizadas da proteína fluorescente verde (GFP), apresenta brusca queda na fluorescência quando em meio ácido (pH < 6).7070 Patterson, G. H.; Knobel, S. M.; Sharif, W. D.; Kain, S. R.; Piston, D. W.; Biophys. J. 1997, 73, 2782. Organelas ácidas, como endossomos tardios e lisossomos, tendem a perder o gradiente de prótons após fixação. Essa característica constitui um inconveniente para algumas aplicações, pois a emissão de fluorescência da maioria dos marcadores é afetada pela acidez (ex. pHRodo e Lysotracker). A título de exemplo, acreditou-se por alguns anos que a internalização via caveolina levasse à estruturas especiais chamadas caveossomos,7171 Pelkmans, L.; Kartenbeck, J.; Helenius, A.; Nat. Cell Biol. 2001, 3, 473. as quais teriam pH mais próximos do fisiológico e não apresentariam enzimas líticas. Entretanto, os mesmos autores que descreveram essas estruturas demonstraram posteriormente que este resultado era um artefato. Ao analisarem as células após fixação observaram que o gradiente de pH era desfeito, e por consequência, ocorria o mesmo com a marcação das estruturas.7272 Hayer, A.; Stoeber, M.; Ritz, D.; Engel, S.; Meyer, H. H.; Helenius, A.; J. Cell Biol. 2010, 191, 615. Esse artefato levou a uma interpretação errônea sobre o tráfego intracelular subsequente à endocitose mediada por caveolina. Inclusive, até hoje é descrito em artigos científicos e de revisão que essa via era mais apropriada para entrega de biomoléculas a células eucarióticas.
O destino intracelular de nanopartículas pode ser fundamental para seu papel biológico ou terapêutico. Consequentemente, o direcionamento intracelular do nanomaterial pode ser uma estratégia para melhorar seu desempenho. Nanopartículas de óxido de ferro foram ligadas ao peptídeo D[KLAKLAK]2, inicialmente sintetizado como antibacteriano e que apresenta alta afinidade pela mitocôndria quando internalizado por células eucarióticas, e direcionaram essas nanopartículas até as mitocôndrias não só de células em cultura, mas também in vivo, conforme demonstrado pela colocalização da partícula e do marcador MitoTracker®.7373 Agemy, L.; Friedmann-Morvinski, D.; Kotamraju, V. R.; Roth, L.; Sugahara, K. N.; Girard, O. M.; Mattrey, R. F.; Verma, I. M.; Ruoslahti, E.; Proc. Natl. Acad. Sci. U. S. A. 2011, 108, 17450.
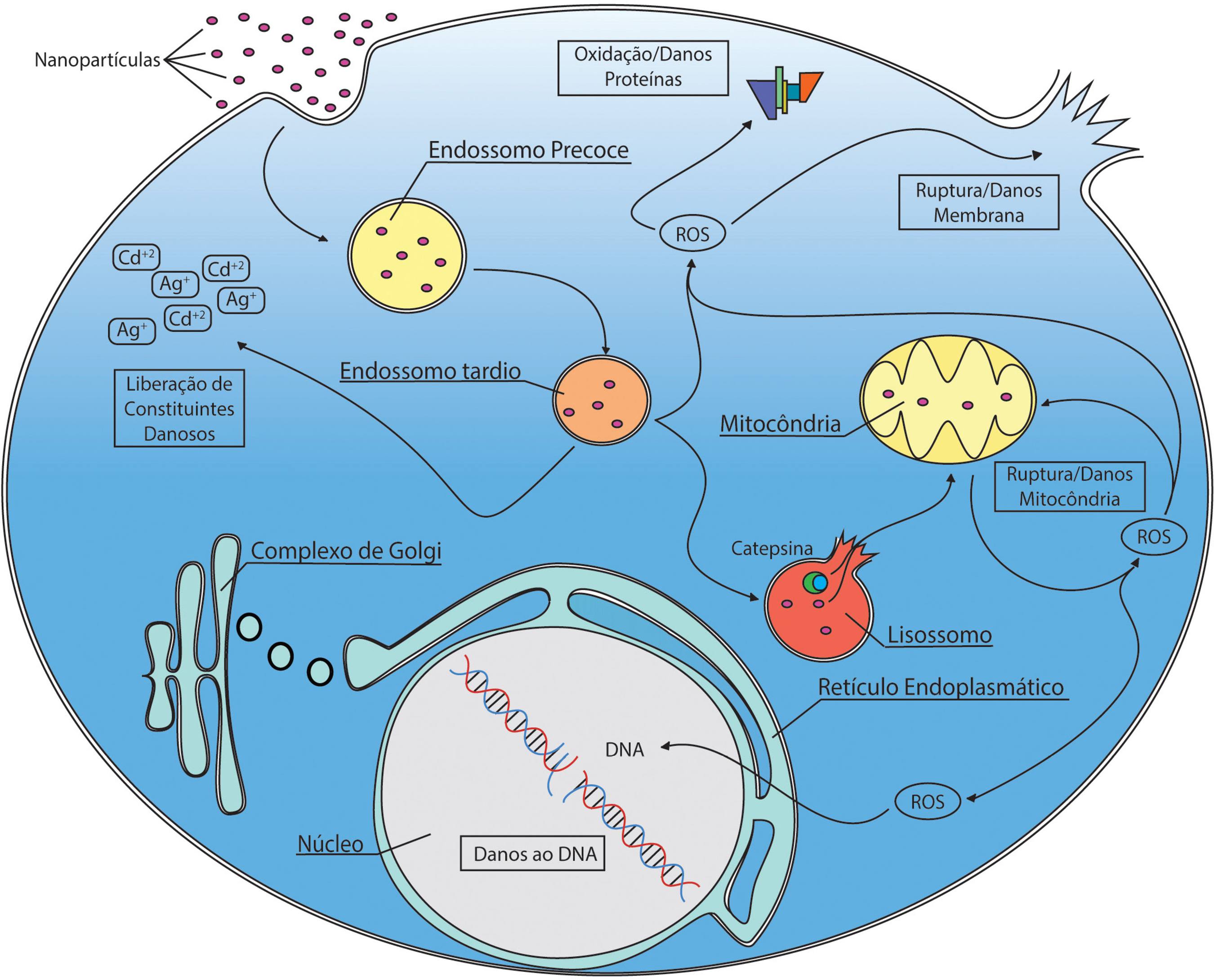
Ao entrarem em contato com células, nanopartículas podem ocasionar efeitos indesejados, apresentados resumidamente na Fig. 3.
Ao serem internalizadas e passarem ao sistema endossomo-lisossomo, nanopartículas podem ser degradadas, liberando constituintes que podem, por exemplo, gerar espécies reativas de oxigênio (ROS). Estas causam danos à proteínas, à membrana celular e ao DNA.8181 Berra, C. M.; Menck, C. F. M.; Martinez, G. R.; de Oliveira, C. S.; Baptista, M. S.; di Mascio, P.; Quim. Nova 2010, 33, 279; Rotilio, G.; Rossi, L.; de Martino, A.; Ferreira, A. M. C.; Ciriolo, M. R.; J. Braz. Chem. Soc. 1995, 6, 221. Danos à membrana dos lisossomos pode liberar enzimas líticas, como a Catepsina (enzima degradadora de polipeptídios), que uma vez no citosol, pode atacar a membrana da mitocôndria, provocando danos e até ruptura desta organela. Ao danificar a mitocôndria, são produzidas mais ROS, entrando em um processo cíclico, no qual ROS é produzida, induzindo mais danos à mitocôndria, gerando ainda mais ROS. Esse processo, na maioria dos casos, pode levar à mutagenicidade do material genético (DNA) e morte celular.8282 Meneghini, R.; Martins, E. A. L.; Calderaro, M.; Quim. Nova 1993, 16, 377.
O tráfego intracelular de nanopartículas é um processo dinâmico e bastante complexo. Por isso nos últimos anos as técnicas para imageamento nessa área têm evoluído e criado uma expectativa de melhor compreensão desse processo.8383 Watson, P.; Jones, A. T.; Stephens, D. J.; Adv. Drug Delivery Rev. 2005, 57, 43. Essas técnicas permitem observação direta de nanopartículas individuais no interior das células e oferecem avanços significativos na resolução espacial (10-20 nm) e temporal (1-2 s) para imagens subcelulares.8484 Vercauteren, D.; Deschout, H.; Remaut, K.; Engbersen, J. F. J.; Jones, A. T.; Demeester, J.; De Smedt, S. C.; Braeckmans, K.; ACS Nano 2011, 5, 7874. Por isso, para os próximos anos, com o desenvolvimento e custos mais acessíveis dessas ferramentas espera-se elucidar melhor esses eventos, o que certamente auxiliaria no entendimento do processo de interação nanopartículas-células.
CONCLUSÃO
A internalização, o tráfego e o processamento intracelular de nanopartículas são processos bastante complexos. A evolução e aplicação de técnicas para imageamento têm contribuído para investigação desses fenômenos possibilitando melhor entendimento dos mesmos.8383 Watson, P.; Jones, A. T.; Stephens, D. J.; Adv. Drug Delivery Rev. 2005, 57, 43. Essas técnicas permitem observação direta de nanopartículas individuais no interior de células e de organelas intracelulares, proporcionando avanços significativos na resolução espacial (10-20nm) e temporal (1-2 s) de imagens subcelulares.8484 Vercauteren, D.; Deschout, H.; Remaut, K.; Engbersen, J. F. J.; Jones, A. T.; Demeester, J.; De Smedt, S. C.; Braeckmans, K.; ACS Nano 2011, 5, 7874.
A utilização de nanopartículas já é uma realidade crescente a cada dia nas mais diversas áreas. Atualmente as três técnicas descritas aqui são as mais utilizadas na caracterização da internalização e tráfego intracelular de nanopartículas em células eucarióticas. Apesar de muitos dos métodos descritos aqui serem utilizados in vivo, em sua maioria essas técnicas são utilizadas em sistemas in vitro. As limitações e a utilidade desses métodos ainda estão sendo determinados. Entretanto, a importância desse tema cresce a cada dia e tem despertado grande preocupação em relação à segurança na utilização de nanomateriais. Nos últimos anos vemos que a nanotoxicologia apresenta importância crescente e o desenvolvimento dessa área depende do desenvolvimento de novas ferramentas. Cresce a conscientização para uso de experimentação em animais; portanto, o desenvolvimento de ferramentas para o entendimento das interações nanopartícula-célula é vital para o futuro desses sistemas.
MATERIAL SUPLEMENTAR
Uma tabela contendo os principais inibidores de endocitose, conjuntamente com breve descrição de seus mecanismos de ação, concentração de uso e estrutura química estão disponíveis em http://quimicanova.sbq.org.br, na forma de arquivo PDF, com acesso livre.
https://minio.scielo.br/documentstore/1678-7064/MwRyWQWcFckqBVyWyZx6Nzt/c58a349b9a077258fbc66c324569d8a5281ca375.pdfAGRADECIMENTOS
Os autores agradecem à FAPESP (Proc. 2014/03002-7 e 2013/05432-6) e CNPq (Proc 140802/2014-2) pelo apoio financeiro.
REFERÊNCIAS
-
1Martins, M. A.; Trindade, T.; Quim. Nova 2012, 35, 1434.
-
2Perugini, P.; Simoni, S.; Scalia, S.; Genta, I.; Modena, T.; Conti, B.; Pavanetto, F.; Int. J. Pharm. (Amsterdam, Neth.) 2002, 246, 37.
-
3Brown, D. M.; Wilson, M. R.; MacNee, W.; Stone, V.; Donaldson, K.; Toxicol. Appl. Pharmacol. 2001, 175, 191.
-
4Asharani, P. V.; Lian Wu, Y.; Gong, Z.; Valiyaveettil, S.; Nanotechnology 2008, 19, 255102.
-
5Arora, S.; Rajwade, J. M.; Paknikar, K. M.; Toxicol. Appl. Pharmacol. 2012, 258, 151.
-
6Oberdörster, G.; Oberdörster, E.; Oberdörster, J.; Environ. Health Perspect. 2005, 113, 823; Franchi, L. P.; Santos, R. A.; Matsubara, E. Y.; de Lima, J. C.; Rosolen, J. M.; Takahashi, C. S.; Quim. Nova 2012, 35, 571.
-
7Paschoalino, M. P.; Marcone, G. P. S.; Jardim, W. F.; Quim. Nova 2010, 33, 421; Dimer, F. A.; Friedrich, R. B.; Beck, R. C. R.; Guterres, S. S.; Quim. Nova 2013, 36, 1520.
-
8Cancino, J.; Marangoni, V. S.; Zucolotto, V.; Quim. Nova 2014, 37, 521.
-
9Stern, S. T.; Adiseshaiah, P. P.; Crist, R. M.; Part. Fibre Toxicol. 2012, 9, 1.
-
10Sahu, S. C.; Casciano, D. A.; Nanotoxicity: from in vivo and in vitro models to health risks, 1st ed., Wiley: Wiltshire, 2009.
-
11Ivanov, A. I.; Exocytosis and Endocytosis, 1st ed., Springer: New York, 2008.
-
12Alberts, B.; Johnson, A.; Lewis, J.; Raff, M.; Roberts, K.; Walter, P.; Molecular Biology of the Cell, 5th ed., Garland Science: New York, 2007.
-
13Marquis, B. J.; Love, S. A.; Braun, K. L.; Haynes, C. L.; RSC Adv. 2009, 134, 425.
-
14Nogueira, D. R.; Mitjans, M.; Rolim, C. M. B.; Pilar Vinardell, M.; Nanomaterials 2014, 4, 454.
-
15Carlson, C.; Hussain, S. M.; Schrand, A. M.; K. Braydich-Stolle, L.; Hess, K. L.; Jones, R. L.; Schlager, J. J.; J. Phys. Chem. B 2008, 112, 13608.
-
16Dijkstra, J.; van Galen, W. J. M.; Hulstaert, C. E.; Kalicharan, D.; Roerdink, F. H.; Scherphof, G. L.; Exp. Cell Res. 1984, 150, 161.
-
17Bhattacharjee, S.; Ershov, D.; Fytianos, K.; van der Gucht, J.; Alink, G. M.; Rietjens, I. M. C. M.; Marcelis, A. T. M.; Zuilhof, H.; Part. Fibre Toxicol. 2012, 9, 1.
-
18Saraste, J.; Palade, G. E.; Farquhar, M. G.; Cell 1986, 83, 6425.
-
19Ferreira, L. A. B.; Radaic, A.; Pugliese, G. O.; Valentini, M. B.; Oliveira, M. R.; Jesus, M. B.; Acta Farmacêutica Portuguesa 2014, 3, 149.
-
20Champion, J. A.; Katare, Y. K.; Mitragotri, S.; Proc. Natl. Acad. Sci. U. S. A. 2007, 104, 11901.
-
21Doherty, G. J.; McMahon, H. T.; Annu. Rev. Biochem. 2009, 78, 857.
-
22Ivanov, A. I. Em Pharmacological Inhibition of Endocytic Pathways: Is It Specific Enough to Be Useful?; Ivanov, A. I., ed.; Humana Press: New York, 2008, cap. 2; Vercauteren, D.; Vandenbroucke, R. E.; Jones, A. T.; Rejman, J.; Demeester, J.; De Smedt, S. C.; Sanders, N. N.; Braeckmans, K.; Mol. Ther. 2010, 18, 561.
-
23Benmerah, A.; Bayrou, M.; Cerf-Bensussan, N.; Dautry-Varsat, A.; J. Cell Sci. 1999, 112), 1303; Damke, H.; Baba, T.; Warnock, D. E.; Schmid, S. L.; J. Cell Biol. 1994, 12, 915; Hsueh, P.-Y.; Edman, M. C.; Sun, G.; Shi, P.; Xu, S.; Lin, Y.-A.; Cui, H.; Hamm-Alvarez, S. F.; MacKay, J. A.; J. Controlled Release 2015, 208, 2.
-
24van den Broeck, D.; de Wolf, M. J. S.; Biotechnology 2006, 41, 475.
-
25Segev, N.; Alfonso, A.; Payne, G. S.; Donaldson, J.; Trafficking Inside Cells - Pathways, Mechanisms and Regulation, 1st ed., Landes Bioscience: New York, 2010.
-
26Behr, J. P.; Chimia 1997, 51, 34.
-
27Martens, T. F.; Remaut, K.; Demeester, J.; De Smedt, S. C.; Braeckmans, K.; Nano Today 2014, 9, 344.
-
28de Jesus, M. B.; Ferreira, C. V.; de Paula, E.; Hoekstra, D.; Zuhorn, I. S.; J. Controlled Release 2010, 148, e89; Ewert, K.; Evans, H. M.; Ahmad, A.; Slack, N. L.; Lin, A. J.; Martin-Herranz, A.; Safinya, C. R. Em Lipoplex structures and their distinct cellular pathways.; Leaf, H.; Mien-Chie, H.; Earnest, W., eds.; Academic Press: San Diego, 2005, cap. 5.
-
29Lin, A. J.; Slack, N. L.; Ahmad, A.; George, C. X.; Samuel, C. E.; Safinya, C. R.; Biophys. J. 2003, 84, 3307.
-
30de Duve, C.; de Barsy, T.; Poole, B.; Trouet, A.; Tulkens, P.; van Hoof, F.; Biochem. Pharmacol. (Amsterdam, Neth.) 1974, 23, 2495.
-
31Benjaminsen, R. V.; Mattebjerg, M. A.; Henriksen, J. R.; Moghimi, S. M.; Andresen, T. L.; Mol. Ther. 2013, 21, 149.
-
32Silva, A. T.; Nguyen, A.; Ye, C.; Verchot, J.; Moon, J. H.; BMC Plant Biol. 2010, 10, 1.
-
33Radaic, A.; Barbosa, L. R. S.; Jaime, C.; Kapila, Y. L.; Pessine, F. B. T.; de Jesus, M. B. Em How lipid cores affect lipid nanopaticles as drug and gene delivery systems; Iglic, A.; Kulkarni, C. V.; Rappol, M., eds.; Elsevier: in press, 2016, cap. 1.
-
34Tada, D. B.; Suraniti, E.; Rossi, L. M.; Leite, C. A. P.; Oliveira, C. S.; Tumolo, T. C.; Calemczuk, R.; Livache, T.; Baptista, M. S.; J. Biomed. Nanotechnol. 2014, 10, 519.
-
35Givan, A. L. Em Flow Cytometry: An Introduction; Hawley, T. S.; Hawley, R. G., eds.; Humana Press: New York, 2004, cap. 1.
-
36Pozarowski, P.; Holden, E.; Darzynkiewicz, Z.; Methods in Molecular Biology, 1st ed., Humana Press: New York, 2013.
-
37Bendall, S. C.; Nolan, G. P.; Roederer, M.; Chattopadhyay, P. K.; Trends Immunol. 2012, 33, 323.
-
38Barnard, R. M.; Bioanalysis 2012, 4, 2471.
-
39Cunningham, R. E. Em Overview of Flow Cytometry and Fluorescent Probes for Flow Cytometry; Oliver, C.; Jamur, M. C., eds.; Humana Press: New Jersey, 2009, cap. 31.
-
40McSharry, J. J.; Methods 2000, 21, 249.
-
41Ibuki, Y.; Toyooka, T. Em Nanoparticle Uptake Measured by Flow Cytometry; Reineke, J., ed.; Humana Press: New York, 2012, cap. 11.
-
42van Gaal, E. V. B.; Spierenburg, G.; Hennink, W. E.; Crommelin, D. J. A.; Mastrobattista, E.; J. Controlled Release 2010, 141, 328.
-
43Hed, J.; FEMS Microbiol. Lett. 1977, 1, 357.
-
44Hed, J.; Hallden, G.; Johansson, S. G. O.; Larsson, P.; J. Immunol. Methods 1987, 101, 119.
-
45Xue, H.-Y.; Wong, H.-L.; ACS Nano 2011, 5, 7034.
-
46Shimoni, O.; Yan, Y.; Wang, Y.; Caruso, F.; ACS Nano 2012, 7, 522.
-
47Martins, S.; Costa-Lima, S.; Carneiro, T.; Cordeiro-da-Silva, A.; Souto, E. B.; Ferreira, D. C.; Int. J. Pharm. (Amsterdam, Neth.) 2012, 430, 216.
-
48Jesus, M. B.; Kapila, Y. L. Em Cellular Mechanisms in Nanomaterial Internalization, Intracellular Trafficking, and Toxicity; Durán, N.; Guterres, S. S.; Alves, O. L., eds.; Humana Press: New York, 2014, cap. 9.
-
49Eruslanov, E.; Kusmartsev, S. Em Identification of ROS Using Oxidized DCFDA and Flow-Cytometry; Armstrong, D., ed.; Humana Press: New York, 2010, cap. 4.
-
50Hansen, T. E.; Johansen, T.; BMC Biol. 2011, 9, 1.
-
51Pozarowski, P.; Darzynkiewicz, Z. Em Analysis of Cell Cycle by Flow Cytometry; Schönthal, A. H., ed.; Humana Press: New York, 2004, cap. 22.
-
52Vermes, I.; Haanen, C.; Steffens-Nakken, H.; Reutellingsperger, C.; J. Immunol. Methods 1995, 184, 39.
-
53Zhang, R.; Piao, M. J.; Kim, K. C.; Kim, A. D.; Choi, J.-Y.; Choi, J.; Hyun, J. W.; Int. J. Biochem. Cell Biol. 2012, 44, 224.
-
54Subtil, A.; Hémar, A.; Dautry-Varsat, A.; J. Cell Sci. 1994, 107), 3461.
-
55Macia, E.; Ehrlich, M.; Massol, R.; Boucrot, E.; Brunner, C.; Kirchhausen, T.; Dev. Cell 2006, 10, 839.
-
56Vercauteren, D.; Vandenbroucke, R. E.; Jones, A. T.; Rejman, J.; Demeester, J.; De Smedt, S. C.; Sanders, N. N.; Braeckmans, K.; Mol. Ther. 2010, 18, 561.
-
57Callegari, F.; Rossini, G. P.; Toxicology 2008 244, 133.
-
58Rodal, S. K.; Skretting, G.; Garred, Ø.; Vilhardt, F.; van Deurs, B.; Sandvig, K.; Mol. Biol. Cell 1999, 10, 961.
-
59Joshi, P.; Chakraborti, S.; Ramirez-Vick, J. E.; Ansari, Z. A.; Shanker, V.; Chakrabarti, P.; Singh, S. P.; Colloids Surf., B 2012, 95, 195.
-
60Pozzi, D.; Marchini, C.; Cardarelli, F.; Salomone, F.; Coppola S, M. M., Zabaleta M. E., Digman M. A., Gratton E, Colapicchioni V, Caracciolo G.; Biochim. Biophys. Acta 2014, 1838,957.
-
61Wittrup, A.; Zhang, S.-H.; Svensson, K. J.; Kucharzewska, P.; Johansson, M. C.; Mörgelin, M.; Belting, M.; Proc. Natl. Acad. Sci. U. S. A. 2010, 107, 13342.
-
62Bang, C.; Batkai, S.; Dangwal, S.; Gupta, S. K.; Foinquinos, A.; Holzmann, A.; Just, A.; Remke, J.; Zimmer, K.; Zeug, A.; Ponimaskin, E.; Schmiedl, A.; Yin, X.; Mayr, M.; Halder, R.; Fischer, A.; Engelhardt, S.; Wei, Y.; Schober, A.; Fiedler, J.; Thum, T.; J. Clin. Invest. 2014, 124, 2136.
-
63Rejman, J.; Bragonzi, A.; Conese, M.; Mol. Ther. 2005, 12, 468.
-
64Damke, H.; Baba, T.; van der Bliek, A. M.; Schmid, L.; J. Cell Biol. 1995, 131, 69.
-
65Rao, A.; Schoenenberger, M.; Gnecco, E.; Glatzel, T.; Meyer, E.; Brändlin, D.; Scandella, L.; J. Phys.: Conf. Ser. 2007, 61, 971.
-
66Haustein, E.; Schwille, P.; HFSP J. 2007, 1, 169.
-
67North, A. J.; J. Cell Biol. 2006, 172, 9.
-
68Fujimoto, K.; Kitamura, M.; Yokokawa, M.; Kanno, I.; Kotera, H.; Yokokawa, R.; ACS Nano 2013, 7, 447; Fichter, K. M.; Ingle, N. P.; McLendon, P. M.; ACS Nano 2013, 7, 347; Vranic S.; Boggetto N.; Contremoulins V.; Mornet S.; Reinhardt N.; Marano F.; Baeza-Squiban A.; S., B.; Part. Fibre Toxicol. 2013, 10, 2; Kasper J.; Hermanns M. I.; Bantz C.; Koshkina O.; Lang T.; Maskos M.; Pohl C.; Unger R. E.; Kirkpatrick C. J.; Arch. Toxicol. 2013, 87, 1052.
-
69Oh, J.-M.; Choi, S.-J.; Kim, S.-T.; Choy, J.-H.; Bioconjugate Chem. 2006, 17, 1411.
-
70Patterson, G. H.; Knobel, S. M.; Sharif, W. D.; Kain, S. R.; Piston, D. W.; Biophys. J. 1997, 73, 2782.
-
71Pelkmans, L.; Kartenbeck, J.; Helenius, A.; Nat. Cell Biol. 2001, 3, 473.
-
72Hayer, A.; Stoeber, M.; Ritz, D.; Engel, S.; Meyer, H. H.; Helenius, A.; J. Cell Biol. 2010, 191, 615.
-
73Agemy, L.; Friedmann-Morvinski, D.; Kotamraju, V. R.; Roth, L.; Sugahara, K. N.; Girard, O. M.; Mattrey, R. F.; Verma, I. M.; Ruoslahti, E.; Proc. Natl. Acad. Sci. U. S. A. 2011, 108, 17450.
-
74Mu, F.-T.; Callaghan, J. M.; Steele-Mortimer, O.; Stenmark, H.; Parton, R. G.; Campbell, P. L.; McCluskey, J.; Yeo, J.-P.; Tock, E. P. C.; Toh, B.-H.; J. Biol. Chem. 1995, 270, 13503; Simonsen, A.; Lippe, R.; Christoforidis, S.; Gaullier, J.-M.; Brech, A.; Callaghan, J.; Toh, B.-H.; Murphy, C.; Zerial, M.; Stenmark, H.; Nature 1998, 394, 494; van der Sluijs, P.; Hull, M.; Webster, P.; Mâle, P.; Goud, B.; Mellman, I.; Cell 1992, 70, 729; Barriocanal, J. G.; Bonifacino, J. S.; Yuan, L.; Sandoval, I. V.; J. Biol. Chem. 1986, 261, 16755; Lippincott-Schwartz, J.; Fambrough, D. M.; J. Cell Biol. 1986, 102, 1593.
-
75Chen, J. W.; Murphy, T. L.; Willingham, M. C.; Pastan, I.; August, J. T.; J. Cell Biol. 1985, 101, 85; Lewis, V.; Green, S. A.; Marsh, M.; Vihko, P.; Helenius, A.; Mellman, I.; J. Cell Biol. 1985, 100, 1839; Ullrich, O.; Reinsch, S.; Urbé, S.; Zerial, M.; Parton, R. G.; J. Cell Biol. 1996, 135, 913; Urbe, S.; Huber, L. A.; Zerial, M.; Tooze, S. A.; Parton, R. G.; FEBS lett. 1993, 334, 175.
-
76Sanders, E. J.; Parker, E.; J. Anat. 2002, 201, 121; Bossy-Wetzel, E.; Newmeyer, D. D.; Green, D. R.; EMBO J. 1998, 17, 37.
-
77Chung, H.-E.; Park, D.-H.; Choy, J.-H.; Choi, S.-J.; Appl. Clay Sci. 2012, 65, 24; Alvarez, C.; J. Biol. Chem. 2000, 276, 2693.
-
78Nakamura, N.; Rabouille, C.; Watson, R.; Nilsson, T.; Hui, N.; Slusarewicz, P.; Kreis, T. E.; Warren, G.; J. Cell Biol. 1995, 131, 1715; Linstedt, A. D.; Hauri, H.-P.; Mol. Biol. Cell 1993, 4, 679; Martin, R. M.; Leonhardt, H.; Cardoso, M. C.; Cytometry, Part A 2005, 67A, 45; Kanda, T.; Sullivan, K. F.; Wahl, G. M.; Curr. Biol. 1998, 8, 377.
-
79Valapala, M.; Vishwanatha, J. K.; J. Biol. Chem. 2011, 286, 30911; Pols, M. S.; Klumperman, J.; Exp. Cell Res. 2009, 315, 1584.
-
80Geminard, C.; J. Biol. Chem. 2000, 276, 9910; Escola, J.-M.; Kleijmeer, M. J.; Stoorvogel, W.; Griffith, J. M.; Yoshie, O.; Geuze, H. J.; J. Biol. Chem. 1998, 273, 20121; Rajagopalan, S.; Xu, Y.; Brenner, M. B.; Science (New York, N.Y.) 1994, 263, 387; Hochstenbach, F.; David, V.; Watkins, S.; Brenner, M. B.; Proc. Natl. Acad. Sci. U. S. A. 1992, 89, 4734.
-
81Berra, C. M.; Menck, C. F. M.; Martinez, G. R.; de Oliveira, C. S.; Baptista, M. S.; di Mascio, P.; Quim. Nova 2010, 33, 279; Rotilio, G.; Rossi, L.; de Martino, A.; Ferreira, A. M. C.; Ciriolo, M. R.; J. Braz. Chem. Soc. 1995, 6, 221.
-
82Meneghini, R.; Martins, E. A. L.; Calderaro, M.; Quim. Nova 1993, 16, 377.
-
83Watson, P.; Jones, A. T.; Stephens, D. J.; Adv. Drug Delivery Rev. 2005, 57, 43.
-
84Vercauteren, D.; Deschout, H.; Remaut, K.; Engbersen, J. F. J.; Jones, A. T.; Demeester, J.; De Smedt, S. C.; Braeckmans, K.; ACS Nano 2011, 5, 7874.
-
85Maxfield, F. R.; Wustner, D.; Methods Cell Biol. 2012, 108, 367.
-
86Maréchal, V.; Prevost, M.-C.; Petit, C.; Perret, E.; Heard, J.-M.; Schwartz, O.; J. Virol. 2001, 75, 11166.
-
87Cannon, G. J.; Swanson, J. A.; J. Cell Sci. 1992, 101, 907.
-
88West, M. A.; Bretscher, M. S.; Watts, C.; J. Cell Biol. 1989, 109, 2731.
-
89Nakase, I.; Niwa, M.; Takeuchi, T.; Sonomura, K.; Kawabata, N.; Koike, Y.; Takehashi, M.; Tanaka, S.; Ueda, K.; Simpson, J. C.; Jones, A. T.; Sugiura, Y.; Futaki, S.; Mol. Ther. 2004, 10, 1011.
-
90Loike, J. D.; Shabtai, D. Y.; Neuhut, R.; Malitzky, S.; Lu, E.; Husemann, J.; Goldberg, I. J.; Silverstein, S. C.; Arterioscler., Thromb., Vasc. Biol. 2004, 24, 2051.
-
91Fretz, M.; Jin, J.; Conibere, R.; Penning, N. A.; Al-Taei, S.; Storm, G.; Futaki, S.; Takeuchi, T.; Nakase, I.; Jones, A. T.; J. Controlled Release 2006, 116, 247; Meier, O.; J. Cell Biol. 2002, 158, 1119.
-
92Dharmawardhane, S.; Schürmann, A.; Sells, M. A.; Chernoff, J.; Schmid, S. L.; Bokoch, G. M.; Mol. Biol. Cell 2000, 11, 3341.
-
93Peterson, J. R.; Mitchison, T. J.; Chemistry & Biology 2002, 9, 1275.
-
94Torgersen, M. L.; Skretting, G.; Deurs, B. v.; Sandvig, K.; J. Cell Sci. 2001, 114, 3737.
-
95Araki, N.; Johnson, M. T.; Swanson, J. A.; J. Cell Biol. 1996, 135, 1249.
-
96Pol, A.; Lu, A.; Pons, M.; Peiró, S.; Enrich, C.; J. Biol. Chem. 2000, 275, 30566; Mettlen, M.; Platek, A.; Van Der Smissen, P.; Carpentier, S.; Amyere, M.; Lanzetti, L.; de Diesbach, P.; Tyteca, D.; Courtoy, P. J.; Traffic 2006, 7, 589.
-
97Ford, M. G. J.; Pearse, B. M. F.; Higgins, M. K.; Vallis, Y.; Owen, D. J.; Gibson, A.; Hopkins, C. R.; Evans, P. R.; McMahon, H. T.; Science (New York, N.Y.) 2001, 291, 1051.
-
98Wang, L.-H.; Rothberg, K. G.; Anderson, R. G.; J. Cell Biol. 1993, 123, 1107.
-
99Tulapurkar, M. E.; Schäfer, R.; Hanck, T.; Flores, R. V.; Weisman, G. A.; González, F. A.; Reiser, G.; Cell. Mol. Life Sci. 2005, 62, 1388; Yao, D.; Ehrlich, M.; Henis, Y. I.; Leof, E. B.; Mol. Biol. Cell 2002, 13, 4001; Liu, J.; Kesiry, R.; Periyasamy, S. M.; Malhotra, D.; Xie, Z.; Shapiro, J. I.; Kidney Int. 2004, 66, 227; Larkin, J. M.; Brown, M. S.; Goldstein, J. L.; Anderson, R. G. W.; Cell 1983, 33, 273.
-
100Carpentier, J. L.; Sawano, F.; Geiger, D.; Gorden, P.; Perrelet, A.; Orci, L.; J. Cell Physiol. 1989, 138, 519; Hansen, S. H.; Sandvig, K.; Van Deurs, B.; J. Cell Biol. 1993, 121, 61.
-
101Page, E.; Winterfield, J.; Goings, G.; Bastawrous, A.; Upshaw-Earley, J.; Doyle, D.; Am. J. Physiol.: Heart Circ. Physiol. 1998, 274, H1988.
-
102Heuser, J. E.; Anderson, R. G.; J. Cell Biol. 1989, 108, 389; Ivanov, A. I.; Nusrat, A.; Parkos, C. A.; Mol. Biol. Cell 2004, 15, 176; Sandvig, K.; Olsnes, S.; Petersen, O. W.; Deurs, B. v.; J. Cell Biol. 1987, 105, 679.
-
103Yumoto, R.; Nishikawa, H.; Okamoto, M.; Katayama, H.; Nagai, J.; Takano, M.; Am. J. Physiol.: Lung Cell Mol. Physiol. 2006, 290, L946.
-
104Nandi, P. K.; Van Jaarsveld, P. P.; Lippoldt, R. E.; Edelhoch, H.; Biochemistry 1981, 20, 6706.
-
105Schnitzer, J. E.; Oh, P.; Pinney, E.; Allard, J.; J. Cell Biol. 1994, 127, 1217.
-
106Ros-Baró, A.; López-Iglesias, C.; Peiró, S.; Bellido, D.; Palacín, M.; Zorzano, A.; Camps, M.; Proc. Natl. Acad. Sci. U. S. A. 2001, 98, 12050.
-
107Orlandi, P. A.; Fishman, P. H.; J. Cell Biol. 1998, 141, 905; Rothberg, K. G.; Ying, Y. S.; Kamen, B. A.; Anderson, R. G.; J. Cell Biol. 1990, 111, 2931.
-
108Liu, P.; Anderson, R. G. W.; Biochem. Biophys. Res. Commun. 1999, 261, 695; Aoki, T.; Nomura, R.; Fujimoto, T.; Exp. Cell Res. 1999, 253, 629.
-
109Keller, P.; Simons, K.; J. Cell Biol. 1998, 140, 1357; Subtil, A.; Gaidarov, I.; Kobylarz, K.; Lampson, M. A.; Keen, J. H.; McGraw, T. E.; Proc. Natl. Acad. Sci. U. S. A. 1999, 96, 6775.
-
110Banquy, X.; Suarez, F.; Rabanel, J. M.; Argaw, A.; Grütter, P.; Bouchard, J. F.; Hildgen, P.; Giasson, S.; Soft Matter 2009, 20, 3984; MacLachlan, J.; Wotherspoon, A. T. L.; Ansell, R. O.; Brooks, C. J. W.; J. Steroid. Biochem. Mol. Biol. 2000, 72, 169.
-
111Diakonova, M.; Bokoch, G.; Swanson, J. A.; Mol. Biol. Cell 2002, 13, 402.
-
112Riento, K.; Frick, M.; Schafer, I.; Nichols, B. J.; J. Cell Biol. 2009, 122, 912.
-
113Vercauteren, D.; Piest, M.; van der Aa, L. J.; Al Soraj, M.; Jones, A. T.; Engbersen, J. F. J.; De Smedt, S. C.; Braeckmans, K.; Biomaterials 2011, 32, 3072; DeLoid, G. M.; Sulahian, T. H.; Imrich, A.; Kobzik, L.; PLoS ONE 2009, 4, e6209.
-
114Pozarowski, P.; Holden, E.; Darzynkiewicz, Z. Em Laser scanning cytometry: principles and applications-an update; Taatjes, D. J.; Roth, J., eds.; Humana Press: New York, 2014, cap. 8.
Datas de Publicação
-
Publicação nesta coleção
Dez 2016
Histórico
-
Recebido
02 Maio 2016 -
Aceito
15 Jun 2016