Resumos
O meloeiro (Cucumis melo L.) é uma frutífera largamente cultivada no Brasil, principalmente no nordeste brasileiro, onde é produzida principalmente para a exportação. Plantas da família do meloeiro, como pepino e abóbora, podem ser severamente afetadas pelo oídio, causado por Podosphaera xanthii.. Este fungo apresenta diversas raças fisiológicas cuja correta identificação é importante para o manejo da doença, já que o uso de variedades resistentes é o método mais eficaz de seu controle. No entanto, a identificação destas raças por meio da prática tradicional de inoculações em uma série diferenciadora de variedades de meloeiro é laboriosa e passível de erros. Devido a isso, um método alternativo seria o uso de marcadores moleculares para determinar de forma rápida a identidade das raças. O objetivo deste estudo foi o de analisar a variabilidade entre isolados de P. xanthii previamente classificados em raças através da técnica de AFLP e do seqüenciamento da região ITS 5.8S do rDNA. A partir dos marcadores AFLP obteve-se um dendrograma no qual não houve separação dos isolados quanto às suas raças, origem geográfica ou hospedeiro de origem. Com esta técnica verificou-se alta variabilidade entre isolados, com similaridade genética máxima de 69% e similaridade mínima de 23%. Ao contrário da informação gerada por AFLP, não foi observada variação na sequência da região ITS 5.8S entre isolados. Desta forma, a análise por AFLP indicou que os isolados tem composição genética heterogênea muito embora este fato não tenha sido evidenciado pelo sequenciamento da região ITS.
Cucumis melo; raças; diversidade genética
Melon crop (Cucumis melo L.) is a fruit largely cultivated in Brazil, especially in the Northwestern region, where it is produced mainly for export. Plants of the melon family, such as cucumber and pumpkin, can be severely affected by powdery mildew, caused by Podosphaera xanthii. This fungus has several physiological races and their correct identification is important for managing the disease, since the use of resistant cultivars is the most effective method for its control. However, identification of races by the traditional method of inoculation in a differential series of melon varieties is laborious and error prone. Due to this, an alternative method would be the use of molecular markers to quickly determine the race identity. The objective of this study was to analyze the variability among P. xanthii isolates previously classified into races through the AFLP technique and sequencing of the rDNA's ITS 5.8S region. With the AFLP markers a dendrogram was generated, where the isolates could not be grouped according to their races, geographic region or host. With this technique, a high variability among isolates was verified, with maximum genetic similarity of 69% and minimum similarity of 23%. In contrast to the AFLP data, there was no sequence variation in the ITS 5.8S region among isolates. Thus the AFLP data indicated that the isolates have a heterogeneous genetic composition even though this was not evidenced by the sequencing of the ITS region.
Powdery mildew; Cucumis melo; races; genetic diversity
ARTIGOS
Estudo da diversidade genética de Podosphaera xanthii através de marcadores AFLP e seqüências ITS
Genetic diversity of Podosphaera xanthii assessed by AFLP markers and ITS sequences
Erika Sayuri NaruzawaI; Raphaelle Komatsu Dalla ValeII; Cynthia Maria SilvaII; Luis Eduardo Aranha CamargoII,* * Autor para correspondência: Luis Eduardo Aranha Camargo ( leacamar@esalq.usp.br)
IUniversité Laval, Faculté de foresterie et géomatique, Département des sciences du bois et de la forêt, Pavillon Charles-Eugène Marchand, G1V 0A6, Québec, Qc, Canada
IIUniversidade de São Paulo, Escola Superior de Agricultura "Luiz de Queiroz", Depto. de Fitopatologia e Nematologia, CEP 13418-900, Piracicaba , SP. Parte da dissertação de mestrado da primeira autora.
RESUMO
O meloeiro (Cucumis melo L.) é uma frutífera largamente cultivada no Brasil, principalmente no nordeste brasileiro, onde é produzida principalmente para a exportação. Plantas da família do meloeiro, como pepino e abóbora, podem ser severamente afetadas pelo oídio, causado por Podosphaera xanthii.. Este fungo apresenta diversas raças fisiológicas cuja correta identificação é importante para o manejo da doença, já que o uso de variedades resistentes é o método mais eficaz de seu controle. No entanto, a identificação destas raças por meio da prática tradicional de inoculações em uma série diferenciadora de variedades de meloeiro é laboriosa e passível de erros. Devido a isso, um método alternativo seria o uso de marcadores moleculares para determinar de forma rápida a identidade das raças. O objetivo deste estudo foi o de analisar a variabilidade entre isolados de P. xanthii previamente classificados em raças através da técnica de AFLP e do seqüenciamento da região ITS 5.8S do rDNA. A partir dos marcadores AFLP obteve-se um dendrograma no qual não houve separação dos isolados quanto às suas raças, origem geográfica ou hospedeiro de origem. Com esta técnica verificou-se alta variabilidade entre isolados, com similaridade genética máxima de 69% e similaridade mínima de 23%. Ao contrário da informação gerada por AFLP, não foi observada variação na sequência da região ITS 5.8S entre isolados. Desta forma, a análise por AFLP indicou que os isolados tem composição genética heterogênea muito embora este fato não tenha sido evidenciado pelo sequenciamento da região ITS.
Palavras-chave adicionais: Cucumis melo, raças, diversidade genética.
ABSTRACT
Melon crop (Cucumis melo L.) is a fruit largely cultivated in Brazil, especially in the Northwestern region, where it is produced mainly for export. Plants of the melon family, such as cucumber and pumpkin, can be severely affected by powdery mildew, caused by Podosphaera xanthii. This fungus has several physiological races and their correct identification is important for managing the disease, since the use of resistant cultivars is the most effective method for its control. However, identification of races by the traditional method of inoculation in a differential series of melon varieties is laborious and error prone. Due to this, an alternative method would be the use of molecular markers to quickly determine the race identity. The objective of this study was to analyze the variability among P. xanthii isolates previously classified into races through the AFLP technique and sequencing of the rDNA's ITS 5.8S region. With the AFLP markers a dendrogram was generated, where the isolates could not be grouped according to their races, geographic region or host. With this technique, a high variability among isolates was verified, with maximum genetic similarity of 69% and minimum similarity of 23%. In contrast to the AFLP data, there was no sequence variation in the ITS 5.8S region among isolates. Thus the AFLP data indicated that the isolates have a heterogeneous genetic composition even though this was not evidenced by the sequencing of the ITS region.
Keywords: Powdery mildew, Cucumis melo, races, genetic diversity
Cucumis melo L., conhecido como meloeiro, é uma olerícola muito apreciada e atualmente popularizada para consumo in natura no Brasil. É também altamente consumida na Europa, Estados Unidos e Japão, de acordo com Silva & Costa (16).
O oídio é uma das doenças foliares mais destrutivas e freqüentes de cucurbitáceas, afetando a fotossíntese e prejudicando a produção dos frutos. O pepino, melão, melancia, abóbora, cabaça, chuchu e bucha são as espécies cultivadas desta família que são mais afetadas por esta doença (12). O patógeno infecta toda a parte aérea da planta hospedeira exceto frutos (17). Sinais compreendem crescimento branco pulverulento que corresponde a conídios, conidióforos e micélio do fungo em ambas as faces da folha. No entanto, a superfície adaxial (17) geralmente é a mais afetada. Estes crescimentos pulverulentos podem coalescer.
Podosphaera (sect. Sphaerotheca) xanthii (Castag.) U. Braun & N. Shish. (Sphaerotheca fuliginea (Schlechtend.:Fr.) Pollaci) e Golovinomyces cichoracearum (Erysiphe cichoracearum DC. ex Merat) são as espécies mais freqüentes de oídio em cucurbitáceas (12, 17) e as de maior importância econômica. Em regiões de climas tropicais e subtropicais, como o Brasil, P. xanthii é a mais freqüente, já que está mais adaptada a climas úmidos, enquanto que G. cichoracearum ocorre em clima seco e com mais freqüência em regiões de clima temperado (12, 17).
Este patógeno apresenta várias raças fisiológicas. A mudança na freqüência de raças ao longo do tempo tem impacto negativo em programas de melhoramento baseados em resistência do tipo raça-específica. Nestes casos, informações sobre a genética e mecanismos de genes de resistência são importantes antes que os genes sejam incorporados em cultivares comerciais. A correta identificação das raças de P. xanthi e informações sobre a ocorrência e distribuição destas são de elevada importância para recomendação de linhagens adequadas em programas de melhoramento e para a implementação de estratégias de controle (19), já que estas diferem em virulência segundo a variedade de melão (13, 6). A identificação de raças é muito importante também para a pesquisa aplicada, principalmente para indústria de sementes, que requer acurácia ao declarar o tipo e o nível de resistência a oídio em seus produtos (3).
A identificação destas raças dá-se através do método tradicional de inoculação do fungo em uma série diferenciadora de cultivares de meloeiro. Apesar de muito utilizada, a identificação de raças de P. xanthii através deste método pode ser dificultada por vários fatores, como diferenças na resposta da planta à inoculação devido a variações nas condições do ambiente, variabilidade genética dentro da população do patógeno e idade e estado nutricional da planta (3). A fim de obter informações mais rápidas e confiáveis sobre raças, o uso de marcadores moleculares ligados a genes de virulência pode representar uma estratégia interessante para a identificação de raças.
O presente trabalho teve por objetivo obter informações que auxiliem no melhoramento genético de meloeiro contra o ataque de P. xanthii, através da avaliação da diversidade genética entre isolados por meio de marcadores moleculares AFLP e de seqüenciamento da região ITS-5.8S do rDNA.
MATERIAL E MÉTODOS
Obtenção e manutenção dos isolados de oídio
Isolados de P. xanthii foram coletados pela Clínica Fitopatológica da empresa Sakata Seeds Sudamerica LTDA e identificados quanto à raça por Fazza (7) usando as cultivares diferenciadoras conforme Tabela 2. Estes isolados foram obtidos de plantas de melão, melancia (Citrullus lanatus L.) e abóbora (Cucurbita máxima L.) nas principais regiões produtoras do Brasil (Tabela 1).
A manutenção dos isolados foi realizada através de inoculação em cotilédones de plantas de pepino (híbrido Safira) cultivadas em bandejas contendo vermiculita. Estes foram cortados e desinfectados superficialmente em câmara asséptica por tratamento de 30 segundos em hipoclorito de sódio 0,3% e duas lavagens em água destilada estéril antes de serem acomodados em placas de petri contendo três papéis de filtro estéreis umedecidos com água. Conídios do fungo foram transferidos para os cotilédones com auxílio de um cílio fixado na ponta de uma pipeta pasteur. Os cotilédones inoculados foram armazenados em câmara de germinação sob temperatura de 22ºC e fotoperíodo 12/12 horas (luz/escuro) por aproximadamente 15 dias. Após este período, o isolado foi novamente inoculado, repetindo-se o procedimento. A manutenção dos isolados foi realizada tanto na Clínica Fitopatológica da empresa quanto no Laboratório de Genética Molecular do setor de fitopatologia da ESALQ/USP.
Caracterização molecular de isolados de Podosphaera xanthii com marcadores AFLP
O DNA dos isolados foi extraído conforme protocolo desenvolvido por Fazza (7). Aproximadamente 50 µg de conídios, conidióforos e micélio de cada isolado foram coletados de colônias mantidas em cotilédones de pepino da variedade Safira com auxílio de agulha hipodérmica, colocados em tubos eppendorf contendo 100 µl de tampão de extração (50 µM Tris HCl pH 7,5; 0,7 M NaCl; 10 µM EDTA pH 8,0; 0,5% SDS) à temperatura ambiente e agitados por 30 segundos. Logo após, as amostras foram centrifugadas a 13.000 rpm por 2 minutos em centrífuga de bancada e 90 µl do sobrenadante foram transferidos para novos tubos contendo 90 µl de isopropanol. As amostras foram homogeneizadas por 2 minutos, centrifugadas por 10 minutos a 13.000 rpm e secas à temperatura ambiente. Após descarte do sobrenadante, os pellets foram ressuspendidos em 20 µl de água milli-Q estéril contendo RNAse (1mg/µl). A quantificação foi realizada em gel de agarose a 1%, tendo como padrão fragmentos de DNA de fago γ com concentração conhecida de 10 ng/µl. A eletroforese se deu em tampão 0,5X TBE (100µM de Tris base, 100µM de ácido bórico e 2 µM de EDTA pH 8,0).
O procedimento de obtenção de marcadores AFLP foi baseado no protocolo adaptado de Vos et al.; (21). O DNA genômico foi digerido por duas enzimas de restrição, uma de corte freqüente (MseI, New England-Biolabs, Estados Unidos) e outra de corte raro (EcoRI, New England-Biolabs, Estados Unidos), em reação composta por 2,0µl de tampão OnePhorAll (OPA)10X, 0,2µl de BSA (10mg/µl), 5µl de DNA (0,6ng/µl) e 2 unidades de cada uma das enzimas em um volume final de 20µl. A incubação foi feita a 37ºC por 20 horas.
Após a digestão, procedeu-se a ligação de adaptadores em reação composta de 0,4µl de tampão da ligase (10X), 0,4µl de cada um dos adaptadores (5µM) e 0,13µl de T4 DNA ligase (3unidades/µl) em volume final de 4µl por tubo com DNA previamente digerido. A incubação foi realizada a 23ºC por três horas. Depois desta etapa foi realizada a reação de pré-amplificação com 25ng de iniciadores EcoRI mais uma base (E+A) combinado com iniciadores MseI mais uma base (M+C), 0,8µl de dNTP (10µM), 2µl de tampão 10X, 1,2µl de MgCl2 (25µM), 0,6µl de Taq DNA polimerase (5unidades/µl) e 2µl de DNA ligado (0,5 ng/µl), em volume final de 20µl. Esta reação iniciou-se a 94ºC por 2 minutos, passando a seguir por 26 ciclos compostos de 94ºC por 1 minuto, 56ºC por 1 minuto e 72ºC por 1 minuto, havendo um ciclo de extensão final a 72ºC por 5 minutos. Ao final deste processo foram acrescentados 80µl de água Milli Q a cada um dos tubos.
O procedimento para a reação da amplificação seletiva foi realizado com iniciadores com 2 nucleotídeos a mais, além do já constante nos iniciadores da reação de pré-amplificação. Assim, foram utilizadas as combinações E+AAA e M+CAA, E+AAC e M+CAC e E+AAA e M+CAT. A reação foi composta por 1,2µl de cada iniciador (25ng/µl), 0,4µl de dNTP (10µM), 2µl de tampão 10X, 1,2µl de MgCl2 (25µM), 0,2µl de Taq DNA polimerase (5u/µl) e 3µl da reação de pré-amplificação diluída, em volume final de 20µl. Esta reação iniciou-se a 94ºC por 2 minutos, passando a seguir por 12 ciclos compostos de 94ºC por 30 segundos, 65ºC por 30 segundos e 72ºC por 1 minuto. Após isto, foram realizados 23 ciclos compostos de 94ºC por 30 segundos, 56ºC por 30 segundos e 72ºC por 1 minuto, havendo um ciclo de extensão final a 72ºC por 2 minutos.
Posteriormente, foi feita uma pré-corrida por eletroforese em gel de acrilamida 6% (50,4g de uréia, 7,2g de acrilamida, 0,36g de bisacrilamida em TBE1X até volume final de 120µl), por 1 hora a 80W a 45ºC. Os produtos obtidos nas reações de amplificação seletiva foram submetidas à eletroforese neste gel nas mesmas condições da pré-corrida, por 5 horas. O gel foi revelado através de metodologia descrita por Creste et al.; (4). Os géis foram fixados em 2 litros de solução 1% ácido acético glacial e 10% de álcool etílico por 10 minutos sob agitação constante, seguido de lavagem por 1 minuto em 2 litros de água destilada sob agitação. Terminada a lavagem, procedeu-se pré-tratamento por 2 minutos e 40 segundos em ácido nítrico 1,5% e posterior lavagem com dois litros de água destilada. Os géis foram tratados com solução de nitrato de prata 0,2%, sob agitação, durante 20 minutos. Em seguida foram realizadas duas lavagens em 2 litros de água por 30 segundos cada. A revelação dos géis foi feita com dois litros de solução gelada de 30 g/l de carbonato de sódio e adicionado 600 µl de formaldeído 37%.
Os fragmentos visualizados no gel foram analisados visualmente produzindo uma tabela binária conforme a presença (1) ou ausência (0) de um determinado fragmento em cada isolado. Foram utilizados dois géis para cada combinação de iniciadores, já que não havia espaço para todos isolados no mesmo gel. Somente fragmentos reproduzíveis foram utilizados na análise. Estes foram identificados comparando-se os padrões eletroforéticos dos géis com os do um gel controle, no qual analisou-se, sob iguais condições, uma sub amostra de dez isolados que foram obtidos de nova extração, digestão, ligação, pré-amplificação e amplificação seletiva.
Os dados foram analisados empregando o programa NTSYSpc (14). A matriz de similaridade foi calculada com base no coeficiente de Jaccard GS(ij) = a / (a + b + c), onde GS(ij) é a medida da similaridade entre isolados "i" e "j"; "a" é o número de bandas polimórficas que são compartilhadas por "i" e "j"; b é o número de bandas presentes em "i" e ausentes em "j"; e "c" é o número de bandas presentes em "j" e ausentes em "i". Os dados gerados foram utilizados na análise de agrupamento pelo método UPGMA (14) para a construção de um dendrograma.
Estudo da diversidade de Podosphaera xanthii através de seqüenciamento da região ITS-5.8S do rDNA
A região ITS-5.8S do gene ribossomal foi amplificada por PCR em uma reação contendo 0,5µl dos iniciadores ITS 4 e ITS 5 (22) a 5µM cada, 0,5µl de dNTP (10µM), 2,5µl de tampão 10X, 1,5µl de MgCl2 (25µM), 0,2µl de Taq DNA polimerase (5U/µl) e 2µl de DNA de P. xanthii (2ng/µl), em volume final de 25µl. Esta reação iniciou-se a 95ºC por 5 minutos, passando a seguir por 40 ciclos a 95ºC por 30 segundos, 50ºC por 30 segundos e 72ºC por 50 segundos.
A partir de gel de agarose 1% foi verificada a qualidade da amplificação dos fragmentos utilizando como padrão fragmentos de DNA de fago l com concentração conhecida de 50 ng/µl, 100 ng/µl e 200 ng/µl. A eletroforese ocorreu em tampão 0,5X TBE (100µM de Tris base, 100µM de ácido bórico e 2 µM de EDTA pH 8,0).
Foram feitas duas reações de PCR para o seqüenciamento nas quais foram utilizados aproximadamente 200 ng de produto da primeira PCR descrita acima. Além deste foi também utilizado 1ul de iniciador forward ITS 4 (TCC TCC GCT TAT TGA TAT GC) a 3,2µM ou 1ul de iniciador reverse ITS 5 (GGA AGT AAA AGT CGT AAC AAG G) a 3,2µM, mais 2ul de tampão Save Money (300ul de Tris-HCl pH 9 1M, 300ul de MgCl2 0,025M e 900ul de água Milli-Q), 2ul do kit Big Dye Terminator (Applied Biosystem) e quantidade de água Milli-Q suficiente para completar 10 ul da reação. A reação iniciou-se a 96°C por 3 minutos e consistiu de 25 ciclos compostos de 96ºC por 10 segundos, 50ºC a 5 segundos e 60ºC por 4 minutos.
Os amplicons foram precipitados em 80ul de isopropanol a 75% para cada amostra e agitados em vortex por 10 segundos. As amostras foram deixadas no escuro por 15 minutos e foram centrifugadas por 45 minutos em centrífuga de bancada à 20°C e 4000rpm. O sobrenadante foi descartado e precipitado lavado com 150ul de etanol gelado a 70%. As amostras foram novamente centrifugadas durante 15 minutos. O sobrenadante foi novamente descartado e o DNA foi então seco durante 1 hora em temperatura ambiente.
Foram adicionados 3ul de tampão Blue Dextran (Promega, Estados Unidos) para cada amostra e agitados em vortex para ressuspender o DNA. A solução com o DNA foi desnaturada a 95°C por 5 minutos e imediatamente imersa em gelo até o momento do seqüenciamento, realizado em seqüenciador ABI 377 Prism, conforme protocolo do fabricante (Applied Biosystems, Estados Unidos).
O gel foi preparado com 18 g de uréia, 26 mL de água bidestilada e 5 mL de solução Long Ranger (Cambrex, Estados Unidos) contendo 5% de acrilamida. Foram então adicionados 5 mL de TBE 10x, 250ul de persulfato de amônio 10% simultaneamente a 35ul de TEMED. Após a polimerização, 1,5ul de cada amostra foram pipetados no gel.
As seqüências foram alinhadas e editadas pelo programa Sequencher (Gene Codes Corporation Ann Arbor, MI, Estados Unidos). Cada seqüência foi editada manualmente através de inspeção visual. Através do programa CLUSTAL X, versão 1.81 (20) foi realizado novo alinhamento. Foram utilizados os parâmetros "gap-opening penalty" de 10.0 e "gap-extention penalty" de 0.1. As seqüências foram comparadas e alinhadas com seqüências de P. xanthii e outras espécies de Podosphaera e G. cichoracearum publicadas no GenBank (P. xanthii acessos EU327327, EU190973, EF050036, AY450961, AB040326, Podosphaera fusca EU159425, Podosphaera leucotricha EU148597, Podosphaera pannosa AB022348, Podosphaera trydactyla var. trydactyla AB022393, Podosphaera clandestina AB026150, Podosphaera spiraeae AB026153, Podosphaera longiseta AB022422, Podosphaera aphanis var. aphanis AB000938, Podosphaera ferruginea var. ferruginea AB027232, Podosphaera balsaminae AB040344, Golovinomyces cichoracearum AB077688). Relações filogenéticas entre as espécies foram determinadas utilizando 1000 réplicas para cálculo de valor de bootstrap (8) pelo método de máxima parcimônia utilizando o programa MEGA (Molecular Evolutionary Genetics Analysis) versão 4 (18).
RESULTADOS E DISCUSSÃO
Caracterização molecular de isolados de Podosphaera xanthii com marcadores AFLP
Vários fragmentos foram obtidos a partir das três combinações de iniciadores. O número médio de fragmentos obtidos por combinação foi 51 dos quais, em média, 31 foram considerados reproduzíveis e, destes, uma média de 28 foram polimórficos. Para a primeira combinação, 76,92% das bandas reproduzíveis foram polimórficas, para a segunda 91,67% e para a terceira 100%. Ao todo foram avaliados 92 fragmentos, de tamanho variando entre 50 e 800 pb. Não foram encontrados isolados 100% similares, indicando a alta variabilidade entre isolados, como pode ser visto na matriz de distâncias. A similaridade genética máxima observada entre isolados foi de 69% entre os isolados 4840-2 e 4871-2 provenientes de plantas de melão coletadas no Rio Grande do Norte, a primeira em Baraúna e a segunda em Mossoró. Ambos os isolados são da raça 3. A similaridade mínima de 23% verificada entre os isolados 4843-2 e 4841-1. O primeiro foi classificado como raça 3 e o segundo foi classificado como raça 5. Estes isolados foram coletados em melão, mas o primeiro no Ceará, no município de Quixeré, e o segundo no Rio Grande do Norte, em Mossoró. Não foram identificados marcadores que discriminassem raças ou que separassem origens geográficas ou hospedeiros com as combinações de iniciadores utilizadas.
Obteve-se um dendrograma no qual foi possível verificar dois grupos principais com 40% de similaridade genética em média entre eles (Figura 1). No primeiro estão agrupados isolados das raças 1, 3 e 5. No outro, há uma sub-divisão na qual verificou-se similaridade genética média de 42% entre isolados. Neste, um sub-grupo concentrou isolados da raça 0, 1, 3 e 5 e o outro isolados da raça 0, 3 e 5. Não houve preciso agrupamento das raças, já que isolados de raças diferentes foram agrupados em um mesmo grupo. Verificou-se que isolados de melão, melancia e abóbora estavam no primeiro grupo principal. Nos outros dois estavam presentes apenas isolados de melão e melancia. Quanto à origem geográfica, também não houve consistência no agrupamento. Em cada um dos grupos haviam isolados de São Paulo, Rio Grande do Norte, Pernambuco e Ceará.
Bardin et al. (1) relataram baixa diversidade genética entre isolados de P. xanthii coletados de várias cucurbitáceas em várias localidades, a exemplo deste trabalho. Esses autores verificaram níveis de similaridade mínimo e máximo de 64% e 100%, respectivamente. No caso de isolados brasileiros, estes valores variaram de 23% a 69%, evidenciando maior variabilidade do que relatado por Bardin et al. (1). A diversidade de hospedeiros e a quantidade de isolados avaliados por estes autores foram semelhantes aos do presente trabalho. Bardin et al. (1) utilizaram isolados de melão, melancia, pepino e abóbora, totalizando 28 isolados, enquanto que no presente trabalho foram utilizados isolados dos mesmos hospedeiros exceto pepino, num total de 22 isolados. Devido a tais semelhanças, esses fatores possivelmente não foram os responsáveis pelas diferenças entre os dados moleculares de diversidade genética verificados entre os dois trabalhos.
É provável que isolados utilizados por Bardin et al. (1), todos coletados em países do hemisfério Norte, sejam de composição genética mais homogênea do que a existente na população de P. xanthii do Brasil. Essa baixa variabilidade de isolados pode ser devida à introdução mais recente desse patógeno nesses países e ao fato de o Brasil possuir clima tropical favorável ao desenvolvimento do patógeno e variadas culturas de cucurbitáceas presentes na maior parte do ano, disponibilizando tecido hospedeiro. Esses fatores proporcionam maior capacidade de adaptação ou evolução mais rápida de seus novos recombinantes, implicando maior variabilidade genética.
Outro fator que pode explicar a discrepância entre os dois trabalhos reside no fato que neste trabalho foram usados marcadores AFLP, em oposição a marcadores RAPD usados por Bardin et al. (1). A técnica de AFLP é muito mais informativa do ponto de vista genético, visto que permite obter um grande número de fragmentos em um único gel e permite alto poder de detecção de variabilidade genética. Isto por aliar as técnicas de RFLP, a qual explora polimorfismos de ausência e presença de sítios de restrição, e de RAPD, a qual explora polimorfismos em sítios de hibridização de primers (9).
A alta variabilidade genética de isolados de P. xanthii encontrada no Brasil é um entrave para o melhoramento de plantas, já que isso não permite direcionar o melhoramento para produção de novas cultivares, recomendadas para uma dada região, as quais contenham genes de resistência a raças que ocorrem nesta região. Desta forma, o desenvolvimento de estratégias de manejo da resistência do hospedeiro, como o uso de piramidamento de genes, pode ser dificultado.
No presente trabalho não foram verificados grupos que reunissem todos isolados de um mesmo hospedeiro, região de origem ou raça, a exemplo dos resultados relatados por Bardin et al. (1). Essa falta de uniformidade de grupos quanto à origem dos isolados possivelmente indica a não especialização dos mesmos quanto aos seus hospedeiros em nível de espécie. Então, isolados provenientes de melão poderiam infectar plantas de abóbora, por exemplo, e isolados de abóbora poderiam infectar plantas de melão, o que já havia sido relatado por Fazza (7). Por outro lado, devida à intensa dispersão do patógeno tanto no Brasil como nos países amostrados por Bardin et al. (1), é possível que a população de isolados esteja misturada de tal forma, que não há como identificar geneticamente isolados de uma mesma região de origem.
Não foram verificados marcadores raça-específicos. Esses marcadores seriam úteis na identificação prática dessas raças devido à utilização de um método uniforme, reduzindo a variação existente na classificação delas, por meio de série diferencial. A região amostrada pelos iniciadores escolhidos para o AFLP provavelmente não incluiu regiões genômicas que abrigam genes responsáveis pela especificidade com relação ao hospedeiro.
Diversidade genética de isolados de P. xanthii através do seqüenciamento da região ITS-5.8S do rDNA
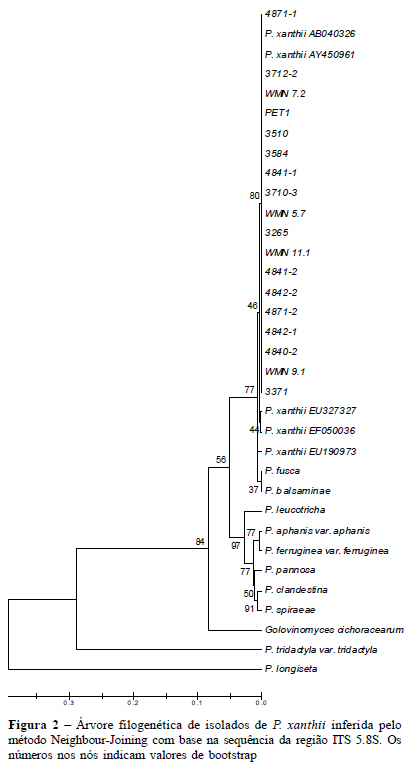
Diferente do que foi observado com o marcador molecular AFLP, nenhuma diversidade foi verificada entre isolados na seqüência de 490 pb da região ITS 5.8S do DNA ribossomal. Os isolados foram agrupados em um único clado com 100% de similaridade junto com duas seqüências de P. xanthii depositadas no GenBank sob números de acesso AB040326 (11) e AY450961 (5) ( Figura 2). O primeiro acesso foi obtido de pepino, no Japão, e o segundo, de Vigna sp., na Austrália.
As outras seqüências de P. xanthii, assim como as de P. fusca e de P. balsaminae, ficaram em clados diferentes ao clado onde foram agrupados os isolados aqui estudados. P. fusca, que também causa oídio em Cucurbitacea, formou grupo junto com P. balsaminae embora o primeiro tenha sido obtido de Calendula officinalis L. e o segundo de Impatiens textori. P. trydactyla var. trydactyla de Prunus japonica e P. longiseta de Prunus grayana, mesmo sendo do mesmo gênero que P. xanthii, foram mais diferentes geneticamente à este que G. cichoracearum sendo este obtido de Lactuca scariola.
O seqüenciamento da região ITS 5.8S do rDNA evidenciou similaridade de 100% entre os isolados brasileiros de P. xanthii, diferindo do resultado obtido com a análise AFLP. Isso ocorreu já que a técnica de AFLP proporciona numerosos marcadores distribuídos randomicamente pelo genoma (2), conforme discutido acima, ao passo que o seqüenciamento aqui realizado foi feito especificamente na região ITS 5.8S do DNA ribossomal. Esta região genômica é utilizada para estudos filogenéticos em nível inter específico, segundo Cunnington et al.; (5), e se aplica particularmente a membros do gênero Podosphaera. Não foi somente neste trabalho que a região ITS evidenciou seqüências iguais de DNA de isolados de oídio; seqüências de nucleotídeos de cinco isolados de oídio obtidos de Cucurbitaceae também foram idênticas conforme Hirata et al.; (11).
Logo, os isolados aqui utilizados são da mesma espécie, já que agruparam com seqüências de P. xanthii da Austrália e do Japão, como confirmado pelo seqüenciamento da região ITS 5.8S do DNA ribossomal e pela árvore filogenética. Entretanto, mais próximos que outras seqüências, uma seqüência de P. xanthii da Hungria e outra de Taiwan formaram um clado e uma seqüência da Itália ficou isolada deste e do clado de P. xanthii, o qual conteve os isolados aqui estudados junto com as seqüências da Austrália e do Japão. Apesar dessa pequena diferença, não há indicativos de que esses isolados da Europa e de Taiwan sejam de outra espécie, visto que não existem diferenças morfológicas entre eles.
É provável que para P. xanthii outra região ou gene seja adequado para estudo filogenético de raças. Conforme Saenz & Taylor (15), genes que codificam proteínas também podem ajudar a entender as relações entre a maioria dos clados identificados no estudo desses autores, os quais demonstraram a utilidade do seqüenciamento da região ITS para identificar a maioria das linhagens de oídio. Com intuito de uma melhor identificação do ancestral do oídio e de ascomicetos próximos a esse grupo de fungos, dados de seqüenciamento de genes mais conservados como a subunidade maior do gene do rRNA ou a subunidade mitocondrial menor do gene do rRNA podem ser úteis, segundo esses mesmos autores. Há necessidade, então, de pesquisas que avaliem novas regiões do genoma ou genes do patógeno para identificação e caracterização dos isolados de oídio de cucurbitáceas, tais como o gene da b-tubulina e histonas (10).
A análise da diversidade genética com base em marcadores AFLP evidenciou alta variabilidade entre isolados. Esta variabilidade não se revelou estruturada segundo hospedeiro ou local de origem geográfica. Em contraposição aos resultados de AFLP, a análise das seqüências da região ITS 5.8S não revelou nenhum polimorfismo entre isolados, indicando que todos são da mesma espécie. A heterogeneidade genética entre isolados de P. xanthii no Brasil representa um entrave ao melhoramento genético do meloeiro. Outras regiões do genoma do fungo devem ser estudadas na tentativa de identificar marcadores que possibilitem distinguir raças de P. xanthii.
AGRADECIMENTOS
Os autores agradecem a Sakata Seeds Sudamerica pelo fornecimento dos isolados e auxílio na classificação dos isolados em raças.
Data de chegada: 26/05/2009.
Aceito para publicação em: 30/06/2010.
- 1. Bardin, M.; Nicot, P.C.; Norman, P.; Lemaire, J.M. Virulence variation and DNA polymorphism in Sphaerotheca fuliginea, causal agent of powdery mildew of cucurbits. European Journal of Plant Pathology, Dordrecht, v. 103, n.6, p. 545554, 1997.
- 2. Bensch, S.; Åkesson, M. Ten years of AFLP in ecology and evolution: why so few animals? Molecular Ecology, Oxford, v. 14, n.10, p. 2899-2914, 2005.
- 3. Cohen, R.; Burger, Y.; Katzir, N. Monitoring physiological races of Podosphaera xanthii (syn. Sphaerotheca fuliginea), the causal agent of powdery mildew in cucurbits: factors affecting race identification and the importance for research and commerce. Phytoparasitica, Bet Dagan, v. 32, n. 2, p. 174-183, 2004.
- 4. Creste, S.; Tulmann Neto, A.; Figueira, A. Detection of single sequence repeat polymorphisms in denaturing polyacrilamide sequencing gels by silver staining. Plant Molecular Biology Reporter, Athens, v. 19, n. 4, p. 299-306, 2001.
- 5. Cunnington, J.H.; Lawrie, A.C.; Pascoe, I.G. Molecular determination of anamorphic powdery mildew fungi on the Fabaceae in Australia. Australasian Plant Pathology, Queensland, v. 33, n.2, p. 281-284, 2004.
- 6. Epinat, C.; Pitrat, M.; Bertrand, F. Genetic analysis of resistance of five melon lines to powdery mildews. Euphytica, Wagening, v. 65, n.2, p. 135 - 144, 1993.
- 7. Fazza, A.C. Caracterização e ocorrência de agentes causais de oídio em cucurbitáceas no Brasil e reação de germoplasma de meloeiro 2006. 60 f. Dissertação (Mestrado em Fitopatologia) Escola Superior de Agricultura "Luiz de Queiroz" - Universidade de São Paulo, Piracicaba.
- 8. Felsenstein, J. Confidence limits on phylogenies: an approach using the bootstrap. Evolution, Lancaster, v. 39, n.4, p. 783-791. 1985
- 9. Ferreira, M.E.; Grattapaglia, D. Introdução ao uso de marcadores moleculares em análise genética 3.ed. Brasília: EMBRAPA, CENARGEN, 1998. 220p.
- 10. Glass, N.L.; Donaldson, G.C. Development of primer sets designed for use with the pcr to amplify conserved genes from filamentous Ascomycetes. Applied and Environmental Microbiology, Washington, v. 61, n. 4, p. 1323-1330, 1995.
- 11. Hirata, T.; Cunnington, J.H.; Paksiri, U.; Limkaisang, S.; Shishkoff, N.; Grigailiunaite, B.; Sato, Y.; Takamatsu, S. Evolutionary analysis of subsection Magnicellulatae of Podosphaera section Sphaerotheca (Erysiphales) based on the rDNA ITS sequences with special reference to host plants. Canadian Journal of Botany, Ottawa, v. 78, n.12, p. 1521-1530, 2000.
- 12. Kurozawa, C.; Pavan, M. A.; Rezende, J. A. M. Doenças das cucurbitáceas (abóbora, abobrinha, chuchu, melancia, melão, moranga, pepino). In: Kimati, H.; Amorim, L.; Rezende, J.A.M.; Bergamin Filho, A.; Camargo, L.E.A. Manual de fitopatologia São Paulo: Agronômica Ceres, 2005. v. 2, cap. 32, p. 293-302.
- 13. Mohamed, Y.F.; Bardin, M.; Nicot, P.C.; Pitrat, M. Causal agents of powdery mildew of cucurbits in Sudan. Plant Disease, St. Paul, v. 79, n.6, p. 634636, 1995.
- 14. Rohlf, F.J. NTSYSpc: numerical taxonomy and multivariate analysis system: version 2.02i (software). New York: Stone Brook, 1998.
- 15. Saenz, G.S.; Taylor, J.W. Phylogeny of the Erysiphales (powdery mildews) inferred from internal transcribed spacer ribosomal DNA sequences. Canadian Journal of Botany, Ottawa, v. 77, n.1, p. 150168, 1999.
- 16. Silva, H.R.; Costa, N.D. Introdução. In: SILVA, H.R.; COSTA, N.D. (Ed.). Melão: produção aspectos técnicos. Brasília: EMBRAPA, 2003. cap. 1, p. 13-14.
- 17. Stadnik, M.J.; Rivera, M.C. Oídios. Jaguariúna : Embrapa Meio Ambiente, 2001. 484 p.
- 18. Tamura, K.; Dudley, J.; Nei, M.; Kumar S. MEGA4: Molecular Evolutionary Genetics Analysis (MEGA) software version 4.0. Molecular Biology and Evolution, Chicago, v. 24, n.8, p. 1596-1599, 2007.
- 19. Thomas, C.E.; Kishaba, N.A.; Mccreight, J.D.; Nugent, P.E. The importance of monitoring races of powdery mildew on muskmelon. Cucurbit Genetics Cooperative Report, Raleigh, n. 7, p. 58 - 59, 1984.
- 20. Thompson, J.D.; Gibson, T.J.; Plewniak, F.; Jeanmougin, F.; Higgins, D.G. The ClustalX windows interface: flexible strategies for multiple sequence alignment aided by quality analysis tools. Nucleic Acids Research, Oxford, v. 25, n.24, p. 48764882, 1997.
- 21. Vos, P.; Hogers, R.; Bleeker, M.; Reijans, M.; Van De Lee, T.; Hornes, M.; Frijters, A.; Pot, J.; Peleman, J.; Kuiper, M.; Zabeau, M. AFLP: a new technique for DNA fingerprinting. Nucleic Acids Research, Oxford, v. 23, n. 21, p. 4407-4414, 1995.
- 22. White, T.J.; Bruns, T.D.; Lee, S.; Taylor, J.W. Amplification and direct sequencing of fungal ribosomal RNA genes for phylogenetics. In: Innis, M.A.; Gelfand, D.H.; Sninsky, J.J.; White, T.J. (ed.). PCR protocols: a guide to methods and applications. San Diego: Academic Press, 1990. p. 315332.
Datas de Publicação
-
Publicação nesta coleção
29 Jun 2011 -
Data do Fascículo
Jun 2011
Histórico
-
Recebido
26 Maio 2009 -
Aceito
30 Jun 2010