Resumos
Grandes quantidades de contaminantes na amostra de DNA dificultam a obtenção de DNA genômico de qualidade durante a extração. A presença de polissacarídeos, fenóis e outros compostos secundários representa o principal problema com o procedimento de isolamento do DNA e sua aplicação subsequente, por inibir a atividade das enzimas Taq DNA polimera-se e enzimas de restrição. Neste estudo, descreveu-se um procedimento modificado baseado no hexadecyltrimethylammonium (CTAB), rendendo DNA genômico satisfatório para técnicas de manipulação subsequente, como reações de PCR e digestão com enzima de restrição. Nesse protocolo foram utilizadas diferentes concentrações de β-mercaptoetanol no tampão de extração (0,0; 0,2; 10; 15; 25; e 50 uL de β-mercaptoetanol/mL do tampão de extração: 100 mM de Tris-HCl, pH 8; 20 mM de EDTA; 1,4 mM de NaCl; 2% de CTAB; 1% de PVP), cujo procedimento foi aplicado no caso de folhas maduras e testado em Annona crassiflora (arati-cum), Eugenia dysenterica (cagaita), Anacardium humilis (caju-do-campo), Hancornia speciosa (mangaba) e Caryocar brasiliense (pequi). O protocolo foi eficiente no isolamento de DNA livre de polissacarídeos e polifenóis, com rendimento do DNA com alto peso molecu-lar, utilizando-se concentrações a partir de 1% de β-mercaptoetanol no tampão de extração. O DNA isolado por esse método mostrou alta pureza, de acordo com as análises de digestão por restrição e amplificação por PCR.
Annona crassiflora (araticum); Eugenia dysenterica (cagaita); Anacardium humilis (caju-do-campo)
Great amounts of contaminants in DNA samples hamper the extraction of high-quality genomic DNA. The main problem is the presence of polysaccharides, phenols and other secondary metabolites that impair the DNA isolation procedure and subsequent application, by inhibiting the Taq DNA polymerase and restriction enzyme activity. This study describes a modified procedure based on hexadecyltrimethylammonium (CTAB) from which genomic DNA, ap-propriate for subsequent manipulation such as PCR reactions and restriction enzyme diges-tion, is derived. This protocol uses different β-mercaptoethanol concentrations in the extrac-tion buffer ( 0.0; 0.2; 10 ; 15; 25 and 50 uL β-mercaptoethanol/ml from the extraction buffer: 100mM Tris-HCl, pH 8 ; 20 mM EDTA, 1.4 M NaCl ; 2% CTAB; 1% PVP ). The procedure was tested on mature leaves of Annona crassiflora (araticum), Eugenia dysenterica (cagaita), Anacardium humilis (caju-do-campo), Hancornia speciosa (mangaba) e Caryocar brasiliensis (pequi ).The protocol was efficient in the isolation of polysaccharide and polyphenol-free DNA of high molecular weight using β-mercaptoethanol concentrations of over 1 % in the extraction buffer. The purity of thereby isolated DNA isolated was high according to the di-gestion analyses by PCR restriction and amplification.
Annona crassiflora (araticum); Eugenia dysenterica (cagaita); Anacardium humilis (caju-do-campo)
Extração de DNA genômico de tecidos foliares maduros de espécies nativas do cerrado
Extraction of genomic DNA from leaf tissues of mature native species of the cerrado
Márcia Nara da Silva
Secretaria de Ciência e Tecnologia do Estado de Goiás, SECTEC, Brasil. E-mail: <marcianara@gmail.com>
RESUMO
Grandes quantidades de contaminantes na amostra de DNA dificultam a obtenção de DNA genômico de qualidade durante a extração. A presença de polissacarídeos, fenóis e outros compostos secundários representa o principal problema com o procedimento de isolamento do DNA e sua aplicação subsequente, por inibir a atividade das enzimas Taq DNA polimera-se e enzimas de restrição. Neste estudo, descreveu-se um procedimento modificado baseado no hexadecyltrimethylammonium (CTAB), rendendo DNA genômico satisfatório para técnicas de manipulação subsequente, como reações de PCR e digestão com enzima de restrição. Nesse protocolo foram utilizadas diferentes concentrações de β-mercaptoetanol no tampão de extração (0,0; 0,2; 10; 15; 25; e 50 uL de β-mercaptoetanol/mL do tampão de extração: 100 mM de Tris-HCl, pH 8; 20 mM de EDTA; 1,4 mM de NaCl; 2% de CTAB; 1% de PVP), cujo procedimento foi aplicado no caso de folhas maduras e testado em Annona crassiflora (arati-cum), Eugenia dysenterica (cagaita), Anacardium humilis (caju-do-campo), Hancornia speciosa (mangaba) e Caryocar brasiliense (pequi). O protocolo foi eficiente no isolamento de DNA livre de polissacarídeos e polifenóis, com rendimento do DNA com alto peso molecu-lar, utilizando-se concentrações a partir de 1% de β-mercaptoetanol no tampão de extração. O DNA isolado por esse método mostrou alta pureza, de acordo com as análises de digestão por restrição e amplificação por PCR.
Palavras-chave:Annona crassiflora (araticum). Eugenia dysenterica (cagaita). Anacardium humilis (caju-do-campo).
ABSTRACT
Great amounts of contaminants in DNA samples hamper the extraction of high-quality genomic DNA. The main problem is the presence of polysaccharides, phenols and other secondary metabolites that impair the DNA isolation procedure and subsequent application, by inhibiting the Taq DNA polymerase and restriction enzyme activity. This study describes a modified procedure based on hexadecyltrimethylammonium (CTAB) from which genomic DNA, ap-propriate for subsequent manipulation such as PCR reactions and restriction enzyme diges-tion, is derived. This protocol uses different β-mercaptoethanol concentrations in the extrac-tion buffer ( 0.0; 0.2; 10 ; 15; 25 and 50 uL β-mercaptoethanol/ml from the extraction buffer: 100mM Tris-HCl, pH 8 ; 20 mM EDTA, 1.4 M NaCl ; 2% CTAB; 1% PVP ). The procedure was tested on mature leaves of Annona crassiflora (araticum), Eugenia dysenterica (cagaita), Anacardium humilis (caju-do-campo), Hancornia speciosa (mangaba) e Caryocar brasiliensis (pequi ).The protocol was efficient in the isolation of polysaccharide and polyphenol-free DNA of high molecular weight using β-mercaptoethanol concentrations of over 1 % in the extraction buffer. The purity of thereby isolated DNA isolated was high according to the di-gestion analyses by PCR restriction and amplification.
Key-words: Annona crassiflora (araticum), Eugenia dysenterica (cagaita) and Anacardium humilis (caju-do-campo).
1. INTRODUÇÃO
Atualmente, diferentes tipos de técnicas moleculares estão disponíveis para detecção do polimorfismo na sequência de DNA como RFLP (Restriction Fragment Length Polymorphism), RAPD (Random Amplified Polymorphic DNA), AFLP (Amplified Fragment Length Polymorphism), VNTR (Variable Number of Tandem Repeats), SSR (Simple Sequence Repeats), SNP (Single Nucleotide Polymorphism) e STS (Sequence Tagged Sites). Essas técnicas moleculares permitem a obtenção de número virtualmente ilimitado de marcadores moleculares que apresentam ampla capacidade de amostragem do genoma. Consequentemente, esses marcadores têm grande potencial em serem utilizados para as mais diversas aplicações, desde estudos genéticos básicos até programas de melhoramento genético de plantas, animais e microrganismos (FERREIRA e GRATTAPAGLIA, 1996). De modo geral, o uso dessas técnicas requerem quantidade suficiente de DNA de boa qualidade, o que é particularmente problemático no caso de estudos com espécies nativas do Cerrado, exigindo o desenvolvimento de protocolos específicos. No entanto, os estudos de identificação e caracterização da diversidade genética por meio de técnicas moleculares envolvem a avaliação de grande número de indivíduos, exigindo a utilização de métodos rápidos e robustos de extração de DNA.
Geralmente, o método de extração de DNA que utiliza detergente CTAB (cetyltrimethylammonium bromide), descrito por Doyle e Doyle (1990), quando aplicado a tecidos maduros de plantas nativas de Cerrado, leva à obtenção de um produto final viscoso e problemático em reações de PCR (Polymerase Chain Reaction), que pode inibir a atividade de várias enzimas usadas em manipulações biológicas de DNA, como Taq DNA polimerase (EDWARD e THOMPSON, 1991; FANG et al., 1992; SHARMA et al., 2002) e enzimas de restrição, HindIII e EcoRI/MseI (DEMEKE e ADAMS, 1992; PERMINGEAT et al., 1998; SCHLINK e RESKI, 2002). Os protocolos tradicionalmente utilizados para extração de DNA vegetal, embora removam as proteínas, nem sempre eliminam efetivamente polissacarídeos (DEMEKE e ADAMS, 1992), pois essas substâncias interferem no rendimento e qualidade do DNA no processo de extração (DESHMUKH et al., 2007). Assim, o processo de extração de ácido nucleico a partir de tecidos foliares maduros de espécies nativas do Cerrado requer adaptações. Os tecidos foliares de diversas espécies vegetais possuem níveis variados de polissacarídeos polifenóis e outros metabólitos secundários que representam o principal problema encontrado no processo de purificação de DNA vegetal. No entanto, esses matabólitos secundários servem para proteger as plantas contra os herbívoros e doenças patogênicas (HARBONE et al., 1991). Dessa forma oferecem resistência natural às degradações física e biológica (QUEIROZ et al., 2002). Compostos fenólicos como os taninos são também utilizados para fabricação de adesivo para madeira, extraídos, por exemplo, da casca de Eucaliptus grandis (VITAL et al., 2004).
Esses contaminantes se ligam firmemente ao ácido nucleico durante o isolamento do DNA e interferem direta ou indiretamente nas reações enzimáticas. Os polissacarídeos podem se coprecipitar com o DNA após a adição do álcool durante a extração, resultando em solução viscosa (SHARMA et al., 2002) de coloração marrom, imprópria para pesquisas que envolvem PCR e digestão por enzima de restrição do DNA. A presença de polifenóis é também problemática, sendo esse tipo de contaminante poderoso agente de oxidação presente em muitas espécies da planta que reduz o rendimento e a qualidade de DNA extraído (LOOMIS, 1974; POREBSKI et al., 1997). Durante a homogeneização dos tecidos no processo de isolamento de DNA, os compostos polifenólicos são oxidados irreversivelmente, ligando-se à proteína e ácidos nucleicos (LOOMIS, 1974; POREBSKI et al., 1997).
Para neutralizar a ação desses contaminantes são usados antioxidantes como β-mercaptoetanol, ácido ascórbico e BSA (Albumina do Soro Bovino) e PVP (Polivinilpirrolidona) (CLARK, 1997; DEHESTANI e TABAR, 2007) no processo de isolamento de DNA. Como exemplo da utilização desses reagentes no processo de extração de DNA, citam-se os experimentos que demonstraram resultados satisfatórios, como a concentração de 5% de β-mercaptoetanol no tampão de extração ao invés de 0,2% (PUCHOOA, 2004), para a espécie Litchi chinensis Sonn, obtendo pellets de DNA de coloração clara, sem limitações no processo de amplificação. Nesse sentido, inclui também o experimento desenvolvido por Li et al. (2002), que utilizaram a concentração final de 2% de β-mercaptoetanol na solução de extração de DNA de Davidia involucrata a partir de seus tecidos foliares maduros, sem nenhum posterior comprometimento na manipulação do DNA.
Embora exista publicação de vários protocolos de isolamento de DNA de planta, foi necessário otimizar um protocolo de extração de DNA específico para remoção de metabólitos secundários em espécies nativas do CCerrado aqui estudadas. Experiências preliminares com essas espécies revelaram elevado índice desses compostos que interferem na execução dos estudos moleculares com marcadores baseados em PCR e enzima de restrição. Portanto, a padronização desse protocolo foi designada especificamente para isolamento do DNA de cinco espécies nativas do Cerrado, utilizando pequenas quantidades de tecidos foliares e obtendo DNA genômico puro com quantidades suficientes para manipulações subsequentes. Esse protocolo foi desenvolvido com a preocupação de obter DNA com maior rendimento e qualidade em relação à pureza das amostras. Assim, este estudo teve como objetivo padronizar o protocolo de extração de DNA empregando folhas de cinco espécies nativas do CCerrado, bem como verificar sua qualidade e quantidade, uma vez que esse bioma deverá ser usado em experimentos envolvendo marcadores moleculares.
2. MATREIAS E MÉTODOS
A coleta do material vegetal para este estudo foi realizada no mês de abril de 2004, na área do arboreto da Escola de Agronomia e Engenharia de Alimento da Universidade Federal de Goiás. Para avaliação do efeito das diferentes concentrações do β-mercaptoetanol no tampão de extração foram utilizadas amostras de folhas maduras das seguintes espécies nativas do Cerrado: araticunzeiro (Annona crassiflora), cagaiteira (Eugenia dysenterica), cajueiro (Anacardium humilis), mangabeira (Hancornia speciosa) e pequizeiro (Caryocar brasiliensis)
A extração do DNA foi realizada utilizando-se o protocolo descrito por Ferreira e Grattapaglia (1996), e diferentes concentrações de β-mercaptoetanol no tampão de extração (0% a 5%), para as cinco espécies nativas do Cerrado estudadas. O protocolo básico de extração consistiu da maceração de aproximadamente 120 mg de tecido foliar fresco em nitrogênio líquido em microtubos de 1,5 mL. As amostras foram incubadas por 30 mi em banho-maria a 60-65 ºC com 700 ìL de tampão de extração pré-aquecido (Tris-HCl 100 mM, pH 8,0; EDTA 20 mM; NaCl 1,4 M; CTAB 2%; e PVP 1%) contendo 0,0; 0,2; 10; 15; 25 e 50 ìL de β-mercaptoetanol.
Durante o procedimento de incubação, os tubos foram agitados a cada 10 min para homogeneização. Após esse período, os tubos foram retirados do banho-maria e resfriados à temperatura ambiente. Em uma capela de exaustão, foram acrescentados 600 ìL do solvente, composto por 24 partes de clorofórmio para 1 de álcool isoamílico (CIA), sendo os tubos agitados durante 10 min. A seguir, procedeu-se à centrifugação à velocidade de 13.000 rpm, durante 5 min. Os tubos foram, então, retirados cuidadosamente e a fase superior (aquosa), transferida para um novo tubo. A este foi adicionado 1/10 do volume de CTAB (10%), que, posteriormente, foi homogeneizado por inversão. O procedimento de extração com 600 ìL de CIA foi repetido duas vezes. Na etapa de precipitação do DNA foram acrescentados 2/3 do volume da solução aquosa de isopropanol frio (-20 ºC). Os tubos foram centrifugados novamente a 7.000 rpm durante 5 min. Após o descarte do sobrenadante, o pellet foi lavado duas vezes em 1,0 mL de etanol 70% e uma vez em 1,0 mL de etanol absoluto, durante 5 min. Posteriormente, os pellets foram secos, ressuspensos com 100 ìL de tampão TE (Tris-HCl 10 mM, pH 8,0; EDTA 0,1 mM) e armazenados em freezer (-20 ºC). O produto das extrações foi visualizado em gel de agarose 1% em tampão TBE (Tris-Borato 90 mM e EDTA 2 mM). Em cada poço do gel foram adicionados 4 ìL da solução de extração, 3 ìL de água ultrapura e 2,0 ìL de LB - tampão de carregamento (azul de bromofenol 0,02%; xileno cianol 0,02%; sacarose, 60%), para um volume total de 8,0 ìL. Após 80 min, os géis foram corados pela adição do brometo de etídio (0,5 ìg/mL, em água destilada) e posterior exposição à luz UV. Os produtos foram quantificados pela comparação com o DNA de fago ë (150, 200, 350 ng).
O DNA genômico extraído conforme protocolo proposto foi digerido utilizando-se 2 ìL da combinação das enzimas EcoRI/MseI [1,25 unidade/ìL cada em 10 mM Tris-HCL, pH 5,0; 50 mM NaCl; 0,1 EDTA; 1 mM DTT; 0,1 mg/ml BSA; 50% (v/v) glycerol; 0,1% triton X-100]; 12,5 ìL de DNA genômico (20 ng/ ìL ); 5 ìL de tampão 5X [50 mM Tris-HCL (pH 7,5), 50 mM Mg acetate, 250 mM K-acetate]; 5,5 ìL de água ultrapura, para um volume final da reação de 25 ìL. As reações foram realizadas a 37 ºC por 2 h, em termociclador. Após a restrição, as enzimas foram inativadas ao se elevar a temperatura para 70 ºC por 15 min. O produto da amplificação do DNA por Reação de Cadeia de Polimerase (PCR) foi obtido utilizando-se seis diferentes primers arbitrários (RAPD) (Random Amplified Polymorphic DNA) com volume final de 25 ìL, contendo a seguinte composição: 3,0 ìL do DNA genômico diluído 1 ng/ ìL, duas unidades de Taq DNA polimerase; 2,5 ìL do tampão 10X buffer plus; 0,2 mM de cada dNTPs, 1,5 mM MgCl2; 2,5 pmol de primers; e 12,85 ìL de água ultrapura. As reações de amplificação foram realizadas em termociclador (PTC-100), cujo programa constou de etapa inicial de desnaturação a 94 ºC, por 5 min, seguido de 48 ciclos, compostos por 92 ºC por 30 seg, 37 ºC por 1 min e 30 seg, 72 ºC por 1 min e 30 seg; a extensão final da reação ocorreu a 72 ºC por 6 min, seguida de incubação a 4 ºC, ao término do programa. Para verificar as digestões e as amplificações do DNA, os produtos dessas reações foram submetidos à eletroforese em gel de agarose 1,0% por 3 e 2 h, respectivamente, em tampão TBE 1X (Tris 0,09 M; ácido bórico 0,09 M; e EDTA 2 mM). Após a corrida, os géis foram corados com brometo de etídeo (0,5 ìg/mL) durante 15 min, para serem posteriormente fotografados.
3. RESULTADOS E DISCUSSÃO
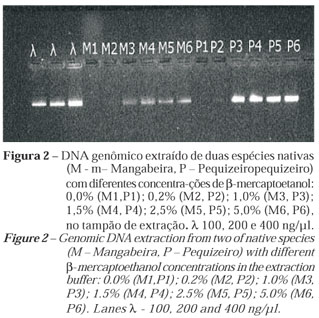
Em função da presença de contaminantes nos tecidos foliares das espécies estudadas, araticunzeiro (Annona crassiflora), cagaiteira (Eugenia dysenterica), cajueiro (Anacardium humilis), mangabeira (Hancornia speciosa) e pequizeiro (Caryocar brasiliense), que interferem no processo de isolamento do DNA, procurou-se otimizar um protocolo de extração para a obtenção de grande quantidade de DNA de boa qualidade. Este estudo partiu do protocolo baseado na utilização do detergente CTAB (FERREIRA e GRATTAPAGLIA, 1996), com diferentes concentrações de β-mercaptoetanol (0,0%, 0,2%, 1,0% , 2,5% e 5%) no tampão de extração. O protocolo descrito aqui é eficiente, barato, com bom rendimento de DNA limpo, boa reprodutibilidade por PCR, como indicado pelos resultados baseados em RAPD e enzimas de restrição (EcoRI/MseI). Os resultados das extrações de DNA das cinco espécies nativas do Cerrado avaliadas em cada concentração de â-mercaptoetanol podem ser visualizados nas Figuras 1 e 2.
Esses resultados sugerem que altas concentrações de β-mercaptoetanol (1,0% a 5%) no tampão de extração permitem a obtenção de maiores quantidades de DNA de boa qualidade, uma vez que não se verificaram nenhuma contaminação e nem sinal de DNA degradado nas amostras que foram extraídas com altas concentrações desses reagentes. E importante salientar que os melhores. resultados foram obtidos com as concentrações de β-mercaptoetanol a partir de 1%, ou seja, 5,0 vezes superior à concentração tradicionalmente recomendada pelo protocolo original. Observou-se ainda que, para todas as espécies estudadas, o padrão de qualidade e quantidade de DNA decai proporcionalmente com a redução da concentração de β-mercaptoetanol no tampão de extração, sugerindo elevado efeito desse componente sobre a qualidade de extração de DNA de tecidos foliares de plantas nativas do Cerrado.
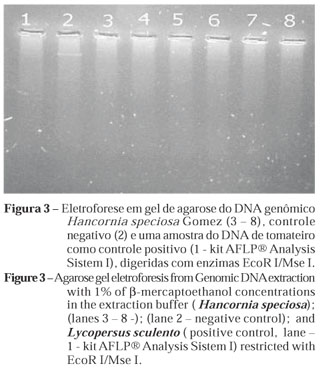
No procedimento referente à digestão por enzima de restrição, cabe ressaltar que foram utilizadas sete amostras de DNA da espécie Hancornia speciosa Gomez obtidas pelo protocolo já descrito, sendo uma das amostras extraídas com a concentração de (0,5%) e as demais com (1,5%) de â-mercaptoetanol, no tampão de extração. O DNA isolado através desse protocolo otimizado foi satisfatório para o processo de digestão do DNA genômico em sítios específicos e amplificação com padrão de migração definido (Figuras 2 e 3). Esse DNA foi utilizado no ensaio de digestão enzimática, e em sete das oito amostras utilizadas apenas uma não foi digerida pela combinação EcoRI/MseI, do kit AFLP® Analysis Sistem I (Figura 2). Assim, constata-se que as amostras de DNA extraídas com 1,5% de β-mercaptoetanol no tampão de extração apresentaram pureza suficiente para os estudos que utilizam diversastécnicas moleculares, incluindo AFLP normalmente empregado em estudos de diversidade genética, técnica essa que requer DNA com alto grau de pureza.
Pela visualização dos produtos da PCR e com a utilização de um marcador de 100 pb no gel de agarose, pode-se observar alta concentração do DNA. Foi verificado um perfil homogêneo e constante de amplificação do DNA genômico resultante das amostras submetidas a concentrações de 1 a 5% de β-mercaptoetanol no tampão de extração. Os fragmentos demonstraram alto peso molecular (Figura 3), um dos aspectos mais importantes para a amplificação bem-sucedida de fragmentos maiores (BORGES, 1997).
O produto da amplificação pelo método de PCR (Polymerase Chain Reation) verificado em gel de agarose, pode-se observar alta concentração do â-mercaptoetanol não comprometem essa técnica; pelo contrário, indica a redução de substâncias que interferem na amplificação do DNA, aumentando a grandeza e a capacidade de detecção das bandas. Dessa forma, não houve nenhum comprometimento nas reações de digestão do DNA com enzima de restrição e nem com amplificação por PCR. Resultados satisfatórios também foram observados por Arbi et al. (2009) no processo de amplificação por PCR (RAPD) e digestão por enzima de restrição (BamH I e EcoR I), com concentração final de 3% do β-mercaptoetanol no tampão de extração.
4. REFERÊNCIAS
Recebido em 28.01.2008 e aceito para publicação em 25.08.2010.
- ARBI, G. et al. A simple, rapid and efficient method for the extraction of genomic DNA from Allium roseum L. (Alliaceae). African Journal of Biotechnology, v.8, n.17, p.4020-4024, 2009.
- DEHESTANI, A.; TABAR S. K. K. A rapid efficient method for DNA isolation from plants with high levels of secondary metabolites. Asian Journal of Plant Sciences, v.6, n.6, p.977-981, 2007.
- DEMEK, T.; ADAMS, R. P. The effects of plant polysaccharides and buffer additives on PCR. Biotecnologia, v.12, n.3, p.333-334, 1992.
- DESHMUKH, V. P. et al. A simple method for isolation of genomic DNA from fresh and dry leaves of Terminalia arjuna (Roxb.) Wight and Argot. Electronic Journal of Biotechnology, v.10, n.3, p.468-472, 2007.
- DOYLE, J. J.; DOYLE, J. L. Isolation of plant DNA from fresh tissue. Focus, v.12, p.13-15, 1990.
- EDWARD, K.; JOHNSTONE, C.; THOMPSON, C. A simple and rapid method for the preparation of plant genomic DNA for PCR analysis. Nucleic Acids Research, v.19, n.6, p.1349, 1991.
- FANG, G.; HAMMER, S.; GROUMET, R. A quick and inexpensive method for removing polysaccharides from plant genomic DNA. Biofeedback, v.13, p.52-54, 1992.
- FERREIRA, M. G.; GRATTAPAGLIA, D. Introdução ao uso de marcadores moleculares em análise genética Brasília: Embrapa-Cenargen, 1996. 220p.
- HARBONE, J. B.; PALO, R. T.; ROBBINS, C. T. Plant defenses against mammalian herbivore Boca Raton: CRC Press LLC, 1991. 192p.
- LI, Y. X.; SU, Z. X.; CHEN, F. Rapid extraction of genomic DNA from leaves and bracts of dove tree (Davidia involucrata). Plant Molecular Biology Reporter, v.20, n.2, p.185a-185e, 2002.
- LOOMIS, M. D. Overcoming problems of phenolics and quinones in the isolation of plant enzymes and organelles. Methods Enzymology, v.31, p.528-544, 1974.
- PERMINGEAT, H. R.; RAMAGNOLI, M. V.; VALLEJOS, R. H. A simple method for isolating high yield and quality DNA from cotton (Gossypium hirsutum L.) leaves. Plant Molecular Biology Reporter, v.16, p.1-6, 1998.
- POREBSKI, S.; BAILEY, L. G. & BAUM, B. R. Modification of a CTAB DNA extraction protocol for plants containing high polysaccharide and polyphenol components. Pant Molecular Biology Reporter, v.15, p.8-15, 1997.
- PUCHOOA, D. A simple, rapid and efficient method for the extraction of genomic DNA from lychee (Litchi chinensis Sonn.). African Journal of Biotechnology, v.3, n.4, p.253-255, 2004.
- QUEIROZ, C. R. A. A.; MORAIS, S. A. L.; NASCIMENTO, E. A. Caracterização dos taninos da aroeira-preta (Myracrodruon urundeuva). Revista Árvore, v.26, n.4, p.485-492, 2002.
- SCHLINK, K.; RESKI, R. Preparing high-quality DNA from moss (Physcomitrella patens). Pant Molecular Biology Reporter, v.20, p.423a-423f, 2002.
- SHARMA, A. D.; GILL, P. K.; SINGH, P. DNA Isolation from dry and fresh samples of polysaccharide-rich plants. Pant Molecular Biology Reporter, v.20, p.415a-415f, 2002.
- VITAL, B. R. et al. Adesivos à base de taninos das cascas de duas espécies de eucalipto para produção de chapas de flocos. Revista Árvore, v.28, n.4, p.571-582, 2004.
Datas de Publicação
-
Publicação nesta coleção
01 Fev 2011 -
Data do Fascículo
Dez 2010
Histórico
-
Recebido
28 Jan 2008 -
Aceito
25 Ago 2010