Resumos
Foram analisadas para a presença de plasmídios 38 amostras de Salmonella Muenster isoladas de suínos e do ambiente de abatedouros localizados no Estado do Rio de Janeiro, no período de março de 1991 a fevereiro de 1992. A seleção das amostras teve como orientação o perfil apresentado frente aos antimicrobianos estreptomicina, tetraciclina, sulfonamida e sulfametoxazol-trimetoprim, sendo analisadas 13 cepas resistentes a um ou mais antimicrobianos, 18 com grau intermediário e sete sensíveis. Plasmidios variando em tamanho de 1,2 Kb a 42 Kb foram detectados em 37 (97,36%) das 38 amostras, correspondendo a 11 perfis distintos (P1 a P11), variando em número de 1 a 6 plasmidios por modelo. O número e diversidade de plasmidios foi maior que os marcos de resistência por cepa. O plasmidio de 2,85 Kb foi o mais freqüente, estando presente em 83,78% das 37 cepas; somente o de 7,95 Kb foi detectado nos dois abatedouros. Não houve paralelismo entre padrão de resistência e perfil plasmidial, onde um mesmo antibiotipo foi encontrado em vários perfis plasmidiais. Os resultados na presente investigação permitiram concluir que a caracterização de plasmidios constituiu-se uma ferramenta útil e simples no rastreamento epidemiológico deste sorovar.
Salmonella Muenster; suíno; resistência antimicrobiana; plasmidios
Thirty-eight strains of Salmonella Muenster, isolated from swine and the abattoir environment, in the State of Rio de Janeiro, Brazil, from March 1991 to February 1992, were analyzed for the presence of plasmids. The strains were selected according to their profile regarding the antimicrobials: streptomycin, tetracycline, sulphonamide and sulfametoxazole-trimethoprim. Thirteen strains were resistant to one or several antimicrobials, 18 with intermediate degree and seven were sensitive. Plasmids varying in size from 1.2 Kb to 42 Kb were detected in 37 (97.36%) of the 38 samples, corresponding to 11 different profiles (P1- P11), varying from 1 to 6 plasmids per model. The number and plasmids diversity was greater than the resistance marks for strains. The plasmid of 2.85 Kb was the most frequent, being present in 83.78% of the 37 strains; only the one of 7.5 Kb was detected at the two slaughterhouses. There was no parallelism between resistance pattern and plasmidial profile, and a same antibiotype was found in several plasmidial profiles. The results of the present investigation, allowed us to conclude that the plasmid characterization is an useful and simple tool for the epidemiological typing of this sorovar.
Salmonella Muenster; swine; antimicrobial resistance; plasmids
Padrão de susceptibilidade a antimicrobianos e perfil plasmidial em Salmonella Muenster isoladas de suínos e do ambiente de abatedouros
Antimicrobial susceptibility pattern and plasmid profile in Salmonella Muenster isolated from swine and abattoir environment, Brazil
Norma S. LázaroI; Anita TibanaII; Eliane M.F. ReisIII; Dália P. RodriguesIII; Bianca R. QuintaesIII; Ernesto HoferIV
IDepto Epidemiologia e Saúde Pública, Instituto de Veterinária, UFRRJ, 23890-000, Seropédica, RJ
IIInst. Microbiologia Prof. Paulo de Góes, UFRJ. E-mail: nslazaro@aol.com
IIILaboratório de. Enterobactérias, Depto Bacteriologia, IOC/FIOCRUZ, Rio de Janeiro
IVLaboratório de Zoonoses Bacterianas, Depto Bacteriologia, IOC/FIOCRUZ
RESUMO
Foram analisadas para a presença de plasmídios 38 amostras de Salmonella Muenster isoladas de suínos e do ambiente de abatedouros localizados no Estado do Rio de Janeiro, no período de março de 1991 a fevereiro de 1992. A seleção das amostras teve como orientação o perfil apresentado frente aos antimicrobianos estreptomicina, tetraciclina, sulfonamida e sulfametoxazol-trimetoprim, sendo analisadas 13 cepas resistentes a um ou mais antimicrobianos, 18 com grau intermediário e sete sensíveis. Plasmidios variando em tamanho de 1,2 Kb a 42 Kb foram detectados em 37 (97,36%) das 38 amostras, correspondendo a 11 perfis distintos (P1 a P11), variando em número de 1 a 6 plasmidios por modelo. O número e diversidade de plasmidios foi maior que os marcos de resistência por cepa. O plasmidio de 2,85 Kb foi o mais freqüente, estando presente em 83,78% das 37 cepas; somente o de 7,95 Kb foi detectado nos dois abatedouros. Não houve paralelismo entre padrão de resistência e perfil plasmidial, onde um mesmo antibiotipo foi encontrado em vários perfis plasmidiais. Os resultados na presente investigação permitiram concluir que a caracterização de plasmidios constituiu-se uma ferramenta útil e simples no rastreamento epidemiológico deste sorovar.
Termos de indexação:Salmonella Muenster, suíno, resistência antimicrobiana, plasmidios.
ABSTRACT
Thirty-eight strains of Salmonella Muenster, isolated from swine and the abattoir environment, in the State of Rio de Janeiro, Brazil, from March 1991 to February 1992, were analyzed for the presence of plasmids. The strains were selected according to their profile regarding the antimicrobials: streptomycin, tetracycline, sulphonamide and sulfametoxazole-trimethoprim. Thirteen strains were resistant to one or several antimicrobials, 18 with intermediate degree and seven were sensitive. Plasmids varying in size from 1.2 Kb to 42 Kb were detected in 37 (97.36%) of the 38 samples, corresponding to 11 different profiles (P1- P11), varying from 1 to 6 plasmids per model. The number and plasmids diversity was greater than the resistance marks for strains. The plasmid of 2.85 Kb was the most frequent, being present in 83.78% of the 37 strains; only the one of 7.5 Kb was detected at the two slaughterhouses. There was no parallelism between resistance pattern and plasmidial profile, and a same antibiotype was found in several plasmidial profiles. The results of the present investigation, allowed us to conclude that the plasmid characterization is an useful and simple tool for the epidemiological typing of this sorovar.
Index terms:Salmonella Muenster, swine, antimicrobial resistance, plasmids.
INTRODUÇÃO
A identificação detalhada de uma cepa é essencial para o monitoramento da disseminação de Salmonella durante surtos em diversas regiões do mundo. O reconhecimento das propriedades fenotípicas através da sorotipagem, biotipagem (Le Minor 1988), tipagem por colicinas (Barker 1980), fagotipagem e testes de susceptibilidade a antimicrobianos (Holmberg et al. 1984) foram, até recentemente, as principais ferramentas para a identificação e rastreamento de uma determinada bactéria em investigações epidemiológicas.
Devido a problemas relacionados à reprodutibilidade ou poder discriminatório associados a esses métodos de tipagem fenotípica, técnicas moleculares foram desenvolvidas propiciando uma boa discriminação entre cepas do mesmo sorotipo ou fagotipo (Threlfall & Frost 1990).
A análise genotípica de Salmonella tem se revelado uma ferramenta útil para tipagem epidemiológica de diferentes sorovares, tais como S. Dublin (Helmuth et al. 1985), S. Heidelberg (Stanley et al. 1992), S. Berta (Olsen et al. 1992), S. Paratyphi B e S. Java (Ezquerra et al. 1993), S. Virchow (Torre et al. 1993), S. Typhi (Threlfall et al. 1994), S. Enteritidis e S. Typhimurium (Millemann et al. 1995).
A análise do perfil plasmidial foi a primeira técnica baseada em DNA para rastreamento epidemiológico (Maslow et al. 1993). Estudos têm demonstrado que este método de tipagem se constitui uma ferramenta discriminatória na caracterização de surtos envolvendo S. Newport (Riley et al. 1983), S. Typhimurium (Holmberg et al. 1984) e S. Enteritidis (Bichler et al. 1994).
Circunstanciando-se em todas estas considerações, procurou-se analisar o perfil plasmidial de Salmonella Muenster isoladas de suínos e do ambiente de abatedouros, tendo como base o padrão de susceptibilidade a antimicrobianos, numa tentativa de arregimentar os elementos necessários que possibilitem uma diferenciação intrínseca e, de tal maneira, utilizar como marcador epidemiológico deste sorovar.
MATERIAL E MÉTODOS
Amostragem. Constituiu-se de 38 cepas de Salmonella Muenster, isoladas de suínos abatidos no Estado do Rio de Janeiro no período de março de 1991 a fevereiro de 1992, a partir de amígdalas (n=16), linfonodos mesentéricos (n=8), pré-escapulares (n=1) e inguinais (n=6), assim como do ambiente de abatedouro (7 amostras).
Determinação do conteúdo plasmidial. A seleção das amostras teve como orientação o perfil apresentado frente aos seguintes antimicrobianos: estreptomicina (Sm), tetraciclina (Tc), Sulfonamida (Su) e sulfametoxazol-trimetoprim (SxTc) através do método de difusão por discos segundo Bauer et al. (1966) e descrito pelo National Commitee for Clinical Laboratory Standards (NCCLS 1998). Desta forma, foram analisadas 38 cepas de S. Muenster das quais, 13 amostras resistentes a um ou mais antimicrobianos, 18 com grau intermediário de sensibilidade e sete sensíveis.
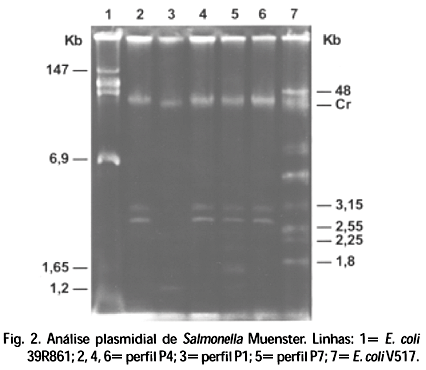
A extração do conteúdo plasmidial foi realizada de acordo com o método de Birnboim & Doly (1979), modificado por Sambrook et al. (1989). Foram utilizados como padrões de peso molecular o DNA plasmidial das cepas de Escherichia coli V517 (Macrina et al. 1978) e 39R861 (Threlfall et al. 1986).
RESULTADOS
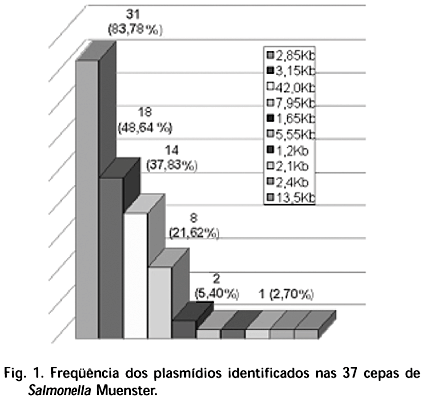
A análise do conteúdo plasmidial de S. Muenster mostrou que plasmídios variando em tamanho de aproximadamente 1,2 Kb a 42 Kb (Fig. 1), foram detectados em 37 (97,36%) das 38 amostras selecionadas, correspondendo A 11 perfis distintos (P1 a P11), variando em número de 1 a 6 plasmídios por modelo (Quadro 1, Fig. 2, 3 e 4). O perfil P8 foi prevalente, sendo detectado em 9 (24,32%) das 37 amostras, seguido de P5 em 8 amostras (21,62%), P4 em 6 amostras (16,21%), P3 em 5 amostras (13,51%), P6 em 3 amostras (8,1%) e os demais em 1 amostra cada (2,7%).
O perfil 3 estava presente em suínos dos dois abatedouros e que P8 e P3 eram os mais disseminados, tendo sido detectados em 3 e 4 visitas aos abatedouros, respectivamente. Ressalta-se que o plasmídio de 2,85 Kb foi comum em 9 dos 11 perfis, estando presente em 31 (83,78%) das 37 cepas que carreavam plasmídios (Quadro 1). De toda a gama de plasmídios identificados nas amostras estudadas, somente o de 7,95 Kb estava presente nos suínos dos dois abatedouros.
Quando discriminou-se a freqüência e distribuição dos plasmiídios de diferentes pesos moleculares em relação aos marcos de resistência Sm e Su, verificou-se que o plasmídio de 2,85 Kb foi o que se apresentou mais estreitamente associado a esses marcos, tanto nas amostras resistentes quanto naquelas com grau intermediário de sensibilidade (Quadro 2).
Não houve paralelismo entre padrão de resistência e perfil plasmidial, tendo sido encontrado até cinco perfis diferentes para o mesmo padrão. Outro aspecto refere-se à presença de plasmídios em 5 de 6 amostras sensíveis aos antimicrobianos testados; dentre estas foram detectados modelos com 1, 2 e 3 plasmídios (perfis 3, 4, 6 e 8 ).
Considerando unicamente as situações de resistência (13 amostras) e sensibilidade (25 amostras), constatou-se que 96% das amostras sensíveis e todas as resistentes carreavam plasmídios.
DISCUSSÃO
Apesar do elevado percentual de isolamento de Salmonella Muenster em 71,73% de 40 suinos portadores e do ambiente de abatedouros (46,1%), no Estado do Rio de Janeiro (Lázaro et al. 1997), as citações na literatura não apontam uma importante intervenção deste sorovar no homem e nos animais, desconhecendo-se inclusive a existência de um sistema de fagotipagem e biotipagem e, até mesmo, um perfil de resistência a antimicrobianos, que possibilitem a sua diferenciação.
No entanto, Benzanson et al. (1983) ressaltaram o valor discriminatório da análise do perfil plasmidial em 10 (25%) de 40 isolados de S. Muenster, dos quais 13 oriundos de fontes animais (bovinos e aves) e 27 do homem. Um plasmídio de 68 Mda (»102 Kb) estava presente em 5 amostras (4 de bovinos e uma de ave) , em 1 amostra (humana) de 26 Mda (39 Kb) e outro de 3 Mda (4,5 Kb) em 4 cepas de origem humana, recuperados em membros de uma mesma família.
De um modo geral, analisando as diferentes configurações, aponta-se a ocorrência de uma população heterogênea de pequenos plasmídios e, com exceção daquele de 42 Kb, os demais se apresentaram com pesos variando de 1,2 a 13,5 Kb, os quais se distribuíram em vários perfis (Quadros 1 e 2).
Os perfis plasmidiais não indicaram uma estreita associação com antibiótico-resistência, até mesmo quando se consideram as amostras que apresentaram grau intermediário de sensibilidade.
Ressalta-se que a presença do plasmídio de 2,85 Kb associado aos marcos Su (em 7 das 10 amostras resistentes) e Sm (5 de 6 amostras), sugere relação com estas características de resistência. Shehabi (1995) detectou um plasmídio de 2,85 Kb em S. Typhimurium, em associação com outros plasmídios em dois modelos de multirresistência envolvendo Sm, Su e Tc, dentre outros antimicrobianos.
A ocorrência do perfil P3 nos dois abatedouros, tendo sido inclusive detectada por ocasião de quatro visitas, sugeriu a disseminação de cepas iguais entre suínos de diferentes origens. Por outro lado, verifica-se uma heterogeneidade de perfis (P4, P8, P9 e P11) nas amostras obtidas dos suínos correspondentes à 7ª visita, demonstrando a provável disseminação de cepas diferentes em animais da mesma procedência.
Devido à carência de estudos em relação ao perfil plasmidial de Salmonella Muenster resistentes a drogas, o confronto dos presentes resultados somente foi possível em relação a outros sorovares.
Plasmídios de baixo peso molecular (2 a 12 Kb), em cepas resistentes a antimicrobianos, particularmente aos marcos Su, Sm e Tc, foram detectados por Holmberg et al. (1984), Simmons et al. (1988), Rivera et al. (1991), Asensi et al. (1995), Millemann et al. (1995), Hampton et al. (1995) e Shehabi (1995). Muito embora, amplamente disseminados entre as amostras resistentes, cumpre salientar que, na maioria das vezes a resistência a estes marcos foi associada a grandes plasmídios (Vinhas & Almeida 1984, Sant'Ana & Chartone-de-Souza 1985, Platt et al. 1987, Rivera et al. 1991, Sant'Ana et al. 1995, Bahrmand & Velayati 1997, Ling et al. 1998).
É importante apontar a destacada ocorrência do plasmídio de 42 Kb em 37,83% das amostras de S. Muenster analisadas (Fig. 1). A presença deste plasmídio de peso molecular relativamente elevado, sugere sua possível correlação com virulência, quando confrontamos com os resultados documentados por vários autores (Helmuth et al. 1985, Platt et al. 1986, Woodward et al. 1989).
Nota-se ainda no Quadro 2 que o número e diversidade de plasmídios foi maior que o número e diversidade de marcos de resistência por cepa. Da mesma forma, o mesmo padrão de resistência (antibiotipo) foi encontrado em vários perfis plasmidiais diferentes, significando que o número de antibiotipos foi menor; neste caso, o perfil plasmidial possibilitou maior discriminação entre os isolados de S. Muenster, sugerindo a presença de diferentes cepas e refletindo a natureza esporádica e não repetitiva dos isolados (Yildirmak et al. 1998).
O conceito de que cepas contendo os mesmos plasmídios pertencem à mesma linha clonal, nem sempre é válido. Tem sido observado, com base em padrões de restrição, idênticos perfis plasmidiais em cepas alocadas em linhas clonais diferentes (Platt et al. 1986). Da mesma forma, isolados de uma mesma geração podem abrigar plasmídios adicionais e apresentar diferentes combinações destes plasmídios (Brown et al. 1992).
Este exemplo foi amplamente observado no presente estudo, com a presença de cepas com fenótipos (antibiotipos) idênticos, carreando plasmídios diferentes (amostras AMI94 e ME7). Chamou a atenção também a presença de cepas que apresentaram antibiotipo e perfil plasmidial distintos em uma mesma fonte de isolamento (amostras ING 66 e MES 66), sugerindo a presença de cepas diferentes em um mesmo suíno.
Outro detalhe interessante refere-se aos isolados com perfis plasmidiais idênticos que variaram em antibiotipo, destacando-se aqueles enquadrados no perfil P8, onde 6 das 9 cepas eram sensíveis a antimicrobianos e, no entanto, carreavam três plasmidios (42 Kb, 3,5 Kb e 2,85 Kb), pressupondo-se que estes podem codificar para outros fatores (Platt et al. 1986).
Salienta-se a presença de plasmídios com o mesmo peso molecular, comuns a vários perfis, particularmente em relação a P2, P4, P6 e P8, onde os plasmídios de 42, 3,15 e 2,85 Kb variam em número e combinações por amostras. Devido à conhecida instabilidade dos plasmídios, os quais podem ser perdidos ou adquiridos durante o processo evolucionário (Threlfall & Frost 1990), admite-se precariamente, a provável presença do perfil plasmidial P8 nas amostras pertencentes aos perfis P2, P4 e P6.
Com base nestas observações, não é possível afirmar que os isolados de S. Muenster, objeto do presente estudo, correspondem a um grande número de cepas de diferentes linhas clonais, até mesmo pela facilidade com que os plasmídios são transferidos entre cepas circulantes. Por outro lado, cepas diferentes podem carrear plasmídios que, apesar de se apresentarem com o mesmo tamanho, não são idênticos, sendo portanto diferenciados mediante o emprego de enzimas de restrição (Platt et al. 1986).
No entanto, os resultados permitiram concluir sobre a importância da caracterização plasmidial dos isolados de Salmonella Muenster, constituindo-se na presente investigação uma ferramenta útil e simples no rastreamento epidemiológico.
Recebido em 21 de outubro de 2002.
Aceito para publicação em 4 de setembro de 2003.
- Asensi M.D., Costa A.P., Reis E.M.F. & Hofer E. 1995. Lysotypes and plasmidial profile of Salmonella serovar Typhimurium isolated from children with enteric processes in the cities of Rio de Janeiro, RJ, and Salvador, BA - Brazil. Revta Inst. Med. Trop. 37:297-302.
- Bahrmand A.R. & Velayati A.A. 1997. Antimicrobial resistance pattern and plasmid profile of Salmonella typhi isolated from an outbreak in Teheran province. Scand. J. Infect. Dis. 29:265-269.
- Barker R.M. 1980. Colicinogeny in Salmonella typhimurium J. Gen. Microbiol. 120:21-26.
- Bauer A.W., Kirby W.M.M., Sherris J.C. & Turey M. 1966. Antibiotic susceptibility testing by a standardized single disc method. Am. J. Clin. Path. 45:493-496.
- Bezanson G.S., Khakhria R. & Pagnutti D. 1983. Plasmid profiles of value in differentiating Salmonella muenster isolates. J. Clin. Microbiol. 17:1159-1160.
- Bichler L.A., Nagaraja K.V. & Pomeroy B.S. 1994. Plasmid diversity in Salmonella enteritidis of animal, poultry and human origin. J. Food Prot. 57:4-11.
- Birnboim H.C. & Doly J. 1979. A rapid alkaline extraction procedure for screening recombinant plasmid DNA. Nucleic Acids Res. 7:1513-1523.
- Brown D.J., Olsen J.E. & Bisgaard M. 1992. Salmonella enterica infection, cross infection and persistance within the environment of a broiler patent stock unit in Denmark. Zentralbl. Bakteriol. 277:129-138.
- Ezquerra E., Burnens A., Jones C. & Stanley J. 1993. Genotypic typing and phylogenetic analysis of Salmonella paratiphi B and S. java with IS200. J. Gen. Microbiol. 139:2409-2414.
- Hampton M.D., Threlfall E.J., Frost J.A., Ward L.R. & Rowe B. 1995. Salmonella typhimurium DT 193: differentiation of an epidemic phage type by antibiogram, plasmid profile, plasmid fingerprint and Salmonella plasmid virulence (spv) gene probe. J. Appl. Bacteriol. 78:402-408.
- Helmuth R., Stephan R., Bunge C., Hoog B., Steinebeck A. & Bulling E. 1985. Epidemiology of virulence-associated plasmid and outer membrane protein patterns within seven common Salmonella serotypes. Infect. Immun. 48:175-182.
- Holmberg S.D., Wachsmyth I.K., Hickman-Brenner F.W. & Cohen M.L. 1984. Comparison of plasmid profile analysis, phage typing, and antimicrobial susceptibility testing in characterizing Salmonella typhimurium isolates from outbreaks. J. Clin. Microbiol. 19:100-104.
- Lázaro N.S., Tibana A. & Hofer E. 1997. Salmonella spp. in healthy swine and in abattoir environments in Brazil. J. Food Prot. 60:1029-1033.
- Le Minor H. 1988. Typing of Salmonella species. Europ. J. Clin. Microbiol. Infect. Dis. 7:214-218.
- Ling J.M., Koo I.C., Kam K.M. & Cheng A.F. 1998. Antimicrobial susceptibilities and molecular epidemiology of Salmonella enterica serotype Enteritidis strains isolated in Hong Kong from 1986 to 1996. J. Clin. Microbiol. 36:1693-1699.
- Macrina F.L., Kopcko D.J., Jones K.R., Mayers D.J. & McCowan S.M. 1978. A multiple plasmid-containing Escherichia coli strain: convenient source of size reference plasmid molecules. Pasmid. 1:417-420.
- Maslow J.N., Mulligan M.E. & Arbit R.D. 1993. Molecular epidemiology: Application of contemporary techniques to the typing of microrganisms. Clin. Inf. Dis. 17:153-164.
- Millemann Y., Lesage M.C., Chaslus-Danda E. & Lafont J.P. 1995. Value of plasmid profiling, ribotyping, and detection of IS200 for tracing avian isolates of Salmonella typhimurium and S. enteritidis J. Clin. Microbiol. 33:173-179.
- National Commitee for Clinical Laboratory Standards. 1998. Performance Standards for Antimicrobial Disk Susceptibility Tests. 18:1-82.
- Olsen J.E., Brown D.J., Baggesen D.L. & Bisgaard M. 1992. Biochemical and molecular characterization of Salmonella enterica serotype berta and comparison of methods for typing. Epidemiol. Infect. 108:243-260.
- Platt D.J., Brown D.J. & Munro D.S. 1986. The distribution of plasmids among a representative colletion of Scottish strains of salmonellae. J. Hyg. Camb. 97:199-204.
- Platt D.J., Brown D.J., Old D.C., Barker R.M., Munro D.S. & Taylor J. 1987. Old and new techniques together resolve a problem of infection by Salmonella typhimurium Epidem. Infect. 99:137-142.
- Platt D.J., Chesham J.S., Brown D.J., Kraft C.A. & Taggart J. 1986. Restriction enzyme fingerprinting of enterobacterial plasmids: a simple strategy with wide application. J. Hyg. Cam. 97:205-210.
- Riley L.W., Di Fernando G.T., De Melfi T.M. & Cohen M.L. 1983. Evaluation of isolated cases of salmonellosis by plasmid profile analysis: introduction and transmission of a bacterial clone by provooked roast beef. J. Infect. Dis. 148:12-17.
- Rivera M.J., Rivera N., Castillo M., Rubio M.C. & Gómes-lus R. 1991. Molecular and epidemiological study of Salmonella clinical isolates. J.Clin. Microbiol. 29:927-932.
- Sambrook J.; Fritsch E.F. & Maniatis T. 1989. Molecular cloning: a laboratory manual. 2nd Ed. Cold Spring Harbor Laboratory, Cold Spring Harbor, N.Y., p.125-128.
- Sant'Ana Y.X. & Chartone-Souza E. 1985. Plasmids that simultaneously carry markers for colicinogeny and resistance to tratracycline in Salmonella typhimurium. Revta Brasil. Gen. 8:231-239.
- Sant'Ana Y.X., Cassali G.D., Barbosa A.J.A., Zucchi T.M.A.D. & Chartone-Souza E. 1995. Salmonella typhimurium plasmids simultaneously coding for tetracycline resistance, colicin production and pathogenicity. Revta Microbiol. 26:239-245.
- Shehabi A.A. 1995 Extra-intestinal infections with multiply drug-resistant Salmonella typhimurium in hospitalized patients in Jordan. Europ. J. Clin. Microbiol. Infect. Dis. 14:448-451.
- Simmons K.W., Wooley R.E. & Brown J. 1988. Comparison of virulence factors and R plasmids of Salmonella spp. isolated from healthy and ill swine. Appl. Environ. Microbiol. 54:760-767.
- Stanley J., Burness A., Powell N., Chowdry N. & Jones C. 1992. The insertion sequence IS200 fingerprints chromosomal genotypes and epidemiological relationships in Salmonella heidelberg J. Gen. Microbiol. 138:2329-2336.
- Threlfall E.J. & Frost J.A. 1990. A Review - The identification, typing and fingerprinting of Salmonella: laboratory aspects and epidemiological applications. J. Appl. Bacteriol. 68:5-16.
- Threlfall E.J., Rowe B., Fergunson J.L. & Ward L. 1986. Characterization of plasmids conferring resistance to gentamicin and apramycin in strains of Salmonella typhimurium phage type 204C isolated in Britain. J. Hyg. 97:419-426.
- Threlfall E.J., Torre W., Ward L.R., Dávalos-Pérez A., Rowe B. & Gilbert I. 1994. Insertion sequence IS200 fingerprinting of Salmonella typhi: an assessment of epidemiological applicability. Epidemiol. Infect. 112:253-261.
- Torre E., Threlfall E.J., Hampton M.D., Ward L.R., Gilbert I. & Rowe B. 1993. Characterization of Salmonella virchow phage types by plasmid profile and IS200 distribution. J. Appl. Bacteriol. 75:435-440.
- Vinhas S.A. & Almeida D.F. 1984. Plasmid mediated antibiotic resistance and colicinoneny among Salmonella in Rio de Janeiro, Brazil. Anais. Acad. Bras. Ciênc. 56:319-322.
- Woodward M.J., Mclaren I. & Wray B. 1989. Distribution of virulence plasmids within Salmonellae. J. Gen. Microbiol. 135:503-511.
- Yildirmak T., Yazgan A. & Ozcengiz G. 1998. Multiple drug resistance patterns and plasmid profiles of non-typhi salmonellae in Turkey. Epidemiol. Infect. 121: 303-307.
Datas de Publicação
-
Publicação nesta coleção
27 Jul 2004 -
Data do Fascículo
Jun 2004
Histórico
-
Aceito
04 Set 2003 -
Recebido
21 Out 2002