Resumos
A produção recombinante de agonistas dos receptores do reconhecimento de padrão do sistema imune inato tem fornecido uma nova ferramenta para a produção de imunoestimulantes para animais. O padrão molecular associado ao patógeno (PAMP), flagelina, codificado pelo gene fljB de Salmonella Typhimurium e o padrão molecular associado ao dano (DAMP) HSP60, codificado pelo gene groEL da S. Typhimurium e S. Enteritidis, são reconhecidos por receptores de reconhecimento de padrões (RRPs) do sistema imune inato das aves. No presente estudo, foi feita a clonagem de fragmentos genéticos dos genes fljB de S. Typhimurium e groEL de S. Typhimurium e S. Enteritidis inseridos no vetor de expressão pET100/D-TOPO e transformados em células de E. coli TOP10. Os clones foram avaliados pela PCR de colônia, PCR de DNA plasmidial e sequenciamento genômico para a confirmação da presença desses genes. Na PCR de colônia, foram identificadas em 80%, 60% e 80% das colônias transformadas, a presença dos genes groEL (S. Enteritidis), groEL (S. Typhimurium) e fljB (S. Typhimurium) respectivamente. O sistema de clonagem adotado possibilitou a produção de clones dos fragmentos genéticos da HSP60 e flagelina das cepas de Salmonella, permitindo a utilização posterior desses clones em ensaios de expressão gênica, com potencial futuro de serem utilizados como imunoestimulante inespecífico das aves.
Proteína recombinante; PAMP; flagelina; DAMP; HSP60; aves
The recombinant production of innate immune system pattern recognition receptor agonists has provided a new tool for the production of immunostimulants for animals. The molecular pattern associated with the pathogen (PAMP), flagellin, coded by the fljB gene from Salmonella Typhimirium, and the molecular pattern associated to the damage (DAMP), HSP60, coded by the groEL gene from S. Typhimurium and S. Enteritidis, are recognized by pattern recognition receptors (PRRs) of the innate immune system of birds. In the present study, we performed the cloning of genetic fragments of the genes fljB, from S. Typhimurium, and groEL from S. Typhimurium and S. Enteritidis inserted in expression vector pET100/D-TOPO and transformed in E. coli TO10 cells. The clones were evaluated by colony PCR, plasmidial DNA PCR and genome sequencing in order to confirm the presence of these genes. In the colony PCR, we identified the presence of genes groEL (S. Enteritidis), groEL (S. Typhimurium) and fljB (S. Typhimurium) in 80%, 60% and 80% of the transformed colonies, respectively. The cloning system adopted allowed the production of HSP60 genetic fragment clones and flagellin of Salmonella strains, allowing the posterior use of these clones in gene expression trials, with the future potential of being used as non-specific immunostimulants for birds.
Recombinant protein; PAMP; flagellin; DAMP; HSP60; birds
ANIMAIS DE PRODUÇÃO/LIVESTOCK DISEASES
Seleção, caracterização e clonagem dos genes fljB e groEL agonistas dos receptores de reconhecimento de padrão do sistema imune inato das aves
Selection, characterization and cloning of the fljB and groEL genes agonists of the innate immune system pattern recognition receptors of birds
Bruno A. SoaresI* * Corresponding author: b runoantunes.soares@yahoo.com.br ; Solange A. SágioII; Ana P. PeconickIII; Priscilla R. BarriosIII; Antônio Chalfun-JúniorII; Geraldo M. CostaIII; Joziana M.P. BarçanteIII; Nelson R.S. MartinsI
ILaboratório de Doenças das Aves, Escola de Veterinária (EV), Universidade Federal de Minas Gerais (UFMG), Av. Presidente Antônio Carlos 6627, Campus Pampulha, Belo Horizonte, MG 31270-901, Brasil. E-mails: nrsmartins@gmail.com;
IILaboratório de Fisiologia Molecular Vegetal (LFV), Universidade Federal de Lavras (UFLA), Campus UFLA, Lavras, MG 37200-000, Brasil. E-mails: solsagio@gmail.com, chalfunjunior@dbi.ufla.br
IIIDepartamento de Medicina Veterinária Preventiva (DMV), UFLA, Campus UFLA, Lavras, MG 37200-000. E-mails: anappeconick@dmv.ufla.br, prbarrios@dmv.ufla.br, gmcosta@dmv.ufla.br
ABSTRACT
The recombinant production of innate immune system pattern recognition receptor agonists has provided a new tool for the production of immunostimulants for animals. The molecular pattern associated with the pathogen (PAMP), flagellin, coded by the fljB gene from Salmonella Typhimirium, and the molecular pattern associated to the damage (DAMP), HSP60, coded by the groEL gene from S. Typhimurium and S. Enteritidis, are recognized by pattern recognition receptors (PRRs) of the innate immune system of birds. In the present study, we performed the cloning of genetic fragments of the genes fljB, from S. Typhimurium, and groEL from S. Typhimurium and S. Enteritidis inserted in expression vector pET100/D-TOPO and transformed in E. coli TO10 cells. The clones were evaluated by colony PCR, plasmidial DNA PCR and genome sequencing in order to confirm the presence of these genes. In the colony PCR, we identified the presence of genes groEL (S. Enteritidis), groEL (S. Typhimurium) and fljB (S. Typhimurium) in 80%, 60% and 80% of the transformed colonies, respectively. The cloning system adopted allowed the production of HSP60 genetic fragment clones and flagellin of Salmonella strains, allowing the posterior use of these clones in gene expression trials, with the future potential of being used as non-specific immunostimulants for birds.
Index terms: Recombinant protein, PAMP, flagellin, DAMP, HSP60, birds.
RESUMO
A produção recombinante de agonistas dos receptores do reconhecimento de padrão do sistema imune inato tem fornecido uma nova ferramenta para a produção de imunoestimulantes para animais. O padrão molecular associado ao patógeno (PAMP), flagelina, codificado pelo gene fljB de Salmonella Typhimurium e o padrão molecular associado ao dano (DAMP) HSP60, codificado pelo gene groEL da S. Typhimurium e S. Enteritidis, são reconhecidos por receptores de reconhecimento de padrões (RRPs) do sistema imune inato das aves. No presente estudo, foi feita a clonagem de fragmentos genéticos dos genes fljB de S. Typhimurium e groEL de S. Typhimurium e S. Enteritidis inseridos no vetor de expressão pET100/D-TOPO e transformados em células de E. coli TOP10. Os clones foram avaliados pela PCR de colônia, PCR de DNA plasmidial e sequenciamento genômico para a confirmação da presença desses genes. Na PCR de colônia, foram identificadas em 80%, 60% e 80% das colônias transformadas, a presença dos genes groEL (S. Enteritidis), groEL (S. Typhimurium) e fljB (S. Typhimurium) respectivamente. O sistema de clonagem adotado possibilitou a produção de clones dos fragmentos genéticos da HSP60 e flagelina das cepas de Salmonella, permitindo a utilização posterior desses clones em ensaios de expressão gênica, com potencial futuro de serem utilizados como imunoestimulante inespecífico das aves.
Termos de indexação: Proteína recombinante, PAMP, flagelina, DAMP, HSP60, aves.
INTRODUÇÃO
A avicultura industrial representa importante setor da produção de alimentos com impacto social e econômico no mundo. A sanidade dos plantéis avícolas é essencial para o abastecimento e a segurança alimentar dos mercados consumidores. Além disso, alguns patógenos aviários como o vírus da influenza aviária, Salmonella e Campylobacter causam infecções zoonóticas (Kaiser 2010). Um dos métodos empregados na prevenção de enfermidades nas aves, a vacinação, é considerada uma das melhores estratégias com custo-benefício, para aumentar a saúde dos animais (Aucouturier et al. 2001). Entretanto, alguns relatos da utilização de determinados tipos de vacinas aviárias evidenciaram a seleção de patógenos com maiores virulências, como o vírus da doença de Marek (Gimeno 2008, Nair 2005). Desta forma, torna-se necessário o desenvolvimento de novos e melhores produtos imunoestimulantes que visam modular respostas protetoras eficazes nas aves.
O sistema imunológico das aves pode ser dividido em sistema inato e adaptativo (Kaiser 2010). O sistema imune inato é considerado a primeira linha de defesa do hospedeiro dado a capacidade de induzir uma resposta rápida e menos específica quando em contato com agentes invasores (Miyaji et al. 2011). O reconhecimento dos patógenos na imunidade inata é mediado pelos Receptores de Reconhecimento de Padrões (RRPs) (Juul-Madsen et al. 2008). Esses receptores reconhecem padrões moleculares associados aos patógenos (PAMPs) compartilhados entre diferentes microrganismos infecciosos, e padrões moleculares associados aos danos (DAMPs) que são ligantes endógenos, muitas vezes referidos como "sinais de perigo" liberados durante o dano tecidual ocorrido em sítios infecciosos e de necrose celular (Tapping 2009, Drexler & Foxwell 2010, Moser & Leo 2010, Takeuchi & Akira 2010). Uma das principais famílias dos RRPs é a dos receptores Toll-like (TLRs) que exercem a função de discriminar os diferentes patógenos, principalmente pelo reconhecimento de PAMPs, e estão envolvidos em um sistema de sinalização que é conservado em mamíferos, aves, insetos e plantas (Means et al. 2000). Os DAMPs são detectados por diversos RRPs que ainda não são bem especificados (Pockley 2001), embora alguns estudos referenciem o reconhecimento dessas moléculas por alguns TLRs (Stewart & Young 2004, Bianchi 2007).
Bactérias patogênicas flagelares expressam a flagelina para promover motilidade, colonização e invasão da mucosa (Bonifield & Hughes 2003). A flagelina exibe algumas características de PAMPs, incluindo sequência conservada, componente importante para o microrganismo, larga distribuição entre bactérias e capacidade de induzir o TLR5 (Hayashi et al. 2001, Berin et al. 2002, Zeng et al. 2003, Ramos et al. 2004).
As proteínas de choque térmico (HSPs) são expressas constitutivamente em todas as células de mamíferos, aves, plantas e em microrganismo, e são classificadas como chaperoninas moleculares (Wallin et al. 2002). HSPs microbianas possuem perfil imunoestimulante baseado no reconhecimento por receptores endocíticos, na internalização das células apresentadoras de antígenos, e na apresentação por via de moléculas do complexo principal de histocompatibilidade (MHC) (Wallin et al. 2002, Tobian et al. 2004). Outro fator apontado por alguns trabalhos é a capacidade de HSPs induzir a maturação fenotípica e funcional das células apresentadoras de antígenos (APCs) (Somersan et al. 2001, Noessner et al. 2002, Wallian et al. 2002).
Diversos experimentos confirmam o potencial de flagelina (Pino et al. 2005, Huleatt et al. 2007, Skountzov et al. 2010) e HSPs (Harmala et al. 2006, Paliwal et al. 2008, Paliwal et al. 2011) produzidas de forma recombinante na capacidade de atuar como imunoestimulantes do sistema imune dos mamíferos. Com isso, objetivou-se, com este trabalho selecionar, caracterizar e clonar transgenes codificadores de agonistas dos receptores de reconhecimento de padrões do sistema imune inato em aves, visando posterior utilização como imunoestimulante na produção avícola.
MATERIAL E MÉTODOS
Análise in silico. As análises in silico, ou estudos de bioinformática, constituíram na pesquisa de genes ou fragmentos genéticos de agonistas para receptores de reconhecimento do sistema imune inato de aves e mamíferos depositados em bancos de dados da National Center for Biotechnology Information - NCBI (2012). Trabalhou-se com as sequências nucleotídicas dos agonistas flagelina e da HSP60 presentes em bactérias. Foram feitas análises de similaridade, utilizando-se o BLAST ou Basic Local Alinhament Sequence Tool (Altschul et al. 1990)e o site de alinhamento JustBIO (http://www.justbio.com) entre diferentes espécies de bactérias para determinação de porções mais conservadas entre as cepas e seleção do fragmento para produção de forma recombinante.
Cultivo bacteriano. Os agonistas de escolha foram relatados em diversos patógenos microbianos, sendo que para esse experimento foram utilizadas cepas de Salmonella Typhimurium (ST) e Salmonella Enteritidis (SE). Foi utilizado como meio de crescimento o meio brain heart infusion (Merck), BHI, preparado conforme as recomendações do fabricante.
Desenho, síntese e utilização de iniciadores. Os iniciadores (forward e reverse) para flanquear as regiões do gene responsável por codificar a flagelina, gene fljB, foram retirados da literatura (Quadro 1). Em seguida, foi realizada a verificação dos iniciadores quanto à capacidade de reconhecer a sequência do gene alvo utilizando-se BLAST (Basic Local Alignment Sequence Tool) (Altschul et al. 1990). Os pares de iniciadores para o gene codificador da HSP60, gene groEL, sequência de número de acesso no NCBI JQ433685.1, foram desenhados no software PrimerQuest (Quadro 1).
O desenho dos iniciadores forward para a inserção dos fragmentos genéticos no vetor de clonagem pET100-D/TOPO® (Invitrogen) foi realizado conforme as especificações do fabricante (Quadro 2). Foi realizada a modificação dos iniciadores forward estabelecidos anteriormente, com adição upstream da sequência CACC na extremidade 5´ do iniciador. Além dessa modificação, foi adicionada após a sequência CACC uma sequência ATG responsável na ligação do ribossomo no processo de tradução. Os iniciadores reverse não foram modificados devido a não complementariedade da sequência GTGG livre do vetor.
Extração do DNA total. Num primeiro momento foi extraído o DNA total das bactérias S. Typhimurium e S. Enteritidis para posterior amplificação dos fragmentos de genes selecionados. Para a extração do DNA total das amostras foi utilizado o kit comercial Wizard® Genomic DNA Purification (Promega), seguindo as recomendações do fabricante.
A integridade do DNA foi avaliada em eletroforese de gel de agarose 1,2% corado com Gel Red™ 1x . As concentrações (ng/µl) também foram determinadas no espectrofotômetro NanoVue™ plus.
Reação em Cadeia pela Polimerase (PCR). Os fragmentos de DNA desejados foram amplificados por PCR utilizando-se o DNA purificado anteriormente. Para cada gene de cada cepa bacteriana, foi padronizada uma reação de PCR.
Amplificação do gene fljB de Salmonella Typhimurium. A reação foi constituída de 2,5µl de tampão de PCR 10X (100 Mm Tris-HCl ph 8,4; 500mM KCl); 3,0mM de MgCl2; 0,2mM de dNTPs (dATP, dTTP, dCTP e dGTP); 50ng do DNA; 1,6 U da Taq DNA Polimerase; 0,5µM dos iniciadores forward e reverse; 14,7µl de água deionizada autoclavada para completar um volume final de 25µl.
As condições para a reação foram: um passo de desnaturação a 95°C por 5 min, seguido de 35 ciclos de 95°C por 1 minuto, anelamento de 65°C por 1 min, extensão a 72°C por 30 segundos e extensão final a 72°C por 7 min.
Amplificação do gene groEL de S. Typhimurium e S. Enteritidis. A reação foi constituída de 2,5 µl de tampão de PCR 10X (100 Mm Tris-HCl ph 8,4; 500mM KCl); 2,5mM de MgCl2; 0,2mM de dNTPs (dATP, dTTP, dCTP e dGTP); 120ng do DNA; 1,6 U da Taq DNA Polimerase; 0,5µM dos iniciadores forward e reverse; 13,7µl de Água MiliQ autoclavada para completar um volume final de 25µl.
As condições para a reação foram: um passo de desnaturação a 95°C por 5 min, seguido de 35 ciclos de 95°C por 1 min, anelamento de 62°C por 1 min, extensão a 72°C por 30 segundos e extensão final a 72°C por 7 min.
Eletroforese em gel de agarose. As amostras foram aplicadas em gel de agarose 1,5%, corado com Gel Red™ 1X, conforme recomendações do fabricante, em cuba de eletroforese horizontal, tendo TBE (Tris-borato 0,09 M e EDTA 0,002 M) como tampão de corrida. Com o auxílio de padrão de peso molecular com os tamanhos de 50, 150, 300, 500, 750 e 1000 pb ( PCR Marker®, Promega) foram identificadas as bandas de interesse.
Clonagem de fragmentos com extremidades coesivas. A clonagem do DNA foi realizada por meio do vetor pET100/D-TOPO® (Invitrogen), que possui o promotor T7 que permite alto nível de expressão para proteínas recombinantes em E. coli competentes. No sistema TOPO® os produtos de PCR são direcionalmente clonados pela adição de quatro bases no primer forward (CACC) que hibridiza com as bases da saliência livre do vetor de clonagem (GTGG) estabilizando o produto de PCR para o direcionamento da clonagem na orientação correta.
O primeiro passo da clonagem foi o da produção de produtos de PCR com extremidades coesivas pela reação de PCR com uma DNA polimerase com atividade revisora, a Pfx50™ (Invitrogen). Posteriormente foi realizada a ligação do produto de PCR com extremidade coesiva ao vetor de clonagem pET100/D-TOPO® (Invitrogen) seguindo as recomendações do fabricante.
Em seguida, realizou-se a reação de transformação da clonagem em células de E. coli competentes TOP10 (Invitrogen) pelo método de transformação química seguindo as especificações do fabricante. Alíquotas de 100 e 200 µl de cada reação de transformação da clonagem foram semeadas em placas pré-aquecidas de LB (Merck) com ampicilina (50µg/ml), e foram incubadas durante a noite a 37°C.
Para a análise dos transformantes, foram escolhidas dez colônias do crescimento anterior. As colônias individuais foram retiradas da placa, depositadas em um tubo de (0,2 ml) para PCR e em seguida cultivadas em 3 ml de LB líquido com ampicilina (50µg/ml) em estufa a 37°C a 200rpm por 24 h.
As reações de PCR de colônia foram padronizadas conforme as reações de PCR estabelecidas inicialmente na identificação dos fragmentos genéticos nas cepas bacterianas.
A próxima etapa consistiu na extração do DNA plasmidial do cultivo bacteriano. Foi utilizado o kit PureLink ™ HQ Mini Kit Plasmid Purification (Invitrogen) seguindo as recomendações do fabricante. Após a obtenção do DNA plasmidial, foram realizadas a determinação das concentrações (ng/µl) do DNA plasmidial no espectrofotômetro NanoVue™ plus, e outra PCR com esse DNA para confirmar as amostras positivas para o vetor com o inserto dos fragmentos genéticos desejados.
Sequenciamento. Os fragmentos dos genes amplificados na PCR e visualizadas na eletroforese em gel de agarose foram purificados utilizando o kit Wizard® SV Gel and PCR Clean-Up System (Promega®) de acordo com as especificações do fabricante. Posteriormente, esses fragmentos foram submetidos ao sequenciamento genético no sequenciador automático capilar ABI310 (Applied Biosystems) pelo método enzimático descrito por Sanger et al. (1977) no Laboratório de Genética da Escola de Veterinária da UFMG. As amostras de DNA plasmidial foram sequenciadas com o mesmo método.
As reações de sequenciamento foram estudadas e analisadas por programas computacionais. O programa Seqman do pacote DNASTAR (DNASTAR Inc.) foi utilizado para comparar os esferogramas com as sequências nucleotídicas e exclusão dos vetores e incorporações incorretas. O software BioEdit® versão 7. 2. 3 (Hall 2013) e o site JustBio (http://www.justbio.com) forneceram as ferramentas para realizar o alinhamento das sequências nucleotídicas com dedução dos aminoácidos correspondentes. Foram feitas buscas por similaridade, utilizando-se BLAST (Altschul et al., 1990), com sequências armazenadas no GenBank™.
As sequências geradas foram comparadas com as sequências dos fragmentos dos genes disponíveis no banco de dados do NCBI (2012) e com a sequência do vetor de clonagem disponível pelo fornecedor (Invitrogen). Os algoritmos BLASTn e BLASTx (Basic Alignment Search Tool - blast 2012) do pacote BLAST 2.0 (Basic Alignment Search Tool) disponibilizado pelo NCBI foram utilizados para a busca de similaridade entre os nucleotídeos.
RESULTADOS
Análise in silico das sequências dos agonistas
Inicialmente, a pesquisa realizada no NCBI para o agonista flagelina e HSP60 gerou no total 34218 e 19375 sequências genéticas, respectivamente. Com o objetivo de cultivar espécies do gênero Salmonella para as etapas subsequentes do ensaio experimental, foram selecionadas das 23 sequências do gene fljB da Salmonella Typhimurium e das 860 sequências da HSP60 do gênero Salmonella, as sequências de número de acesso NC_016860.1, AM933172.1 e JQ433685.1, respectivamente para os genes fljB (526pb), groEL (S. Enteritidis, 1461 pb) e groEL (S. Typhimurium, 1647 pb). Utilizou-se a sequência consenso, com 100% de identidade, dos gene fljB e dos genes groEL para avaliação e construção de iniciadores. A similaridade da sequência do gene groEL (209 pb) entre as duas cepas bacterianas utilizadas foi de 99,03%, o que permitiu a utilização dos mesmos iniciadores para amplificar fragmentos desse gene com as duas diferentes amostras de DNA genômico na PCR.
PCR para genes agonistas de Salmonella Enteritidis e S . Typhimurium
O tamanho dos fragmentos amplificados do gene groEL de S. Enteritidis, gene groEL e fljB de S. Typhimurium foram 209pb, 209pb e 526 pb respectivamente (Fig.1). Esses resultados da PCR corroboram com a alta especificidade dos iniciadores para a amplificação desses genes. O alinhamento das sequências obtidas no presente estudo com sequências depositadas em bancos de dados públicos confirmaram a identidade dos amplicons com os genes groEL e fljB.
Produção de produtos de PCR com extremidades coesivas para inserção no vetor de clonagem pET100/D-TOPO
Posteriormente, na reação de produção de produtos de PCR com extremidades coesivas para inserção no vetor de clonagem pET100/D-TOPO, o resultado da eletroforese em gel de agarose mostrou as bandas esperadas com os tamanhos próximos aos dos genes, gene fljB de S. Typhimurium com 533pb e genes groEL das duas cepas com 216pb (Fig.2).
Confirmação da presença do inserto em colônias de Escherichia coli competentes pela PCR de colônia e sequenciamento genético
Foi detectado o crescimento de colônias de E. coli TOP10 no meio sólido, após a reação de transformação da clonagem pelo método de transformação química. O total de 10 colônias desse crescimento foi escolhido aleatoriamente para cada fragmento genético, e analisadas quanto à presença do inserto pela técnica da PCR de colônia. Foram identificadas em 80%, 60% e 80% das colônias transformadas, a presença dos genes groEL (S. Enteritidis), groEL (S. Typhimurium) e fljB (S. Typhimurium) respectivamente (Fig,3). As amostras que amplificaram bandas inespecíficas, com tamanhos diferentes do esperado, provavelmente por uma inserção incorreta ou duplicada do transgene, foram descartadas dos procedimentos científicos seguintes.
As colônias positivas na técnica de PCR de colônia foram submetidas ao crescimento em meio líquido, e posteriormente o DNA plasmidial extraído dessas colônias foram avaliadas quanto à presença do inserto clonado pela PCR.
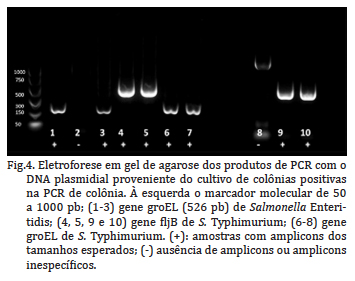
Apenas as amostras de DNA plasmidial que apresentaram a visualização das bandas dos amplicons com os tamanhos esperados na eletroforese em gel de agarose, foram submetidas ao sequenciamento genômico (Fig.4). O sequenciamento confirmou a presença dos fragmentos genéticos na direção correta, das amostras de DNA plasmidial.
DISCUSSÃO E CONCLUSÃO
No presente trabalho, o PAMP flagelina de S. Typhimurium, codificada pelo gene fljB, foi escolhida devido seu potencial de ativação de um membro do grupo dos TLRs, o TLR5 (Ramos et al. 2004, Metcalfe et al. 2010, Putten et al. 2010). Com o objetivo de trabalhar com agonistas com atividades DAMP que possuem capacidade de ativar receptores ainda não especificados na literatura, foi escolhida a HSP60 das cepas S. Typhimurium e S. Enteritidis, ambas codificadas pelo gene groEL (Ensgraber & Loos 1992, Nair et al. 2002, Rainczak et al. 2011).
A proteína de choque térmico, codificadas pelo gene groEL apresenta em sua sequência regiões que são altamente conservadas quando analisadas numa mesma espécie (Karuna et al. 2003, Rebecca et al. 2002). No presente experimento, a avaliação in silico confirmou uma região de alta similariedade entre esse DAMP da S. Enteritidis e S. Typhimurium, o que justificou a utilização de um mesmo iniciador para amplificação dessas sequências genéticas na PCR. Esses achados corroboram com o estudo realizado por Hu et al. (2010) que avaliaram a presença de regiões conservadas nas sequências do gene groEL de diferentes sorovares de Salmonella.
Diversos trabalhos que têm por ferramenta a clonagem de genes imunoestimulantes fizeram a produção de produtos de PCR para inserção nos plasmídeos de clonagem, adequando às especificidades dos vetores utilizados (Skountzou et al. 2010, Paliwal et al. 2011, Rainczak et al. 2011). Esse é o primeiro trabalho com a adoção do pET100/D-TOPO para clonagem de genes bacterianos agonistas do sistema imune inato das aves. A utilização deste vetor deve-se a capacidade de atuar em ensaios de clonagem e de expressão heteróloga, o que viabiliza a utilização dos produtos clonados na geração de proteínas recombinantes.
Os iniciadores utilizados por Dilmaghani et al. (2011) para o gene fljB foram os mesmos adotados no presente estudo. Desta forma, a visualização dos produtos de PCR do gene fljB na eletroforese em gel de agarose com os mesmos tamanhos encontrados por esses autores, confirmaram a presença desse gene nas amostras provenientes de S. Typhimurium. Rainczak et al. (2011) adotaram iniciadores para o gene groEL da S. Enteritidis que produziram amplicons de 1600 pb para posterior inserção em vetores de clonagem. A produção de amplicons de 209 pares de bases para esses genes de ambas as cepas foi devido à possibilidade de utilizar esses clones para procedimentos de expressão heteróloga dos fragmentos genéticos de tamanhos reduzidos e futuramente avaliar o potencial de pequenos peptídeos recombinantes estimularem o sistema imune inato das aves.
Mazur et al. (2010) realizaram a clonagem do antígeno p24 do capsídeo do vírus da imunodeficiência felina com adoção do pET100/D-TOPO. Esses pesquisadores adotaram as mesmas especificações utilizadas nesse presente trabalho para a geração de produtos de PCR com extremidades coesivas, como a adição CACC nos iniciadores forward e utilização da DNA polimerase de alta fidelidade, que posteriormente permitiu a inserção desses fragmentos na orientação correta nos plasmídeos. Como referido por Mazur et al. (2010), a adoção desse sistema de clonagem é viável para a produção de proteínas recombinantes uma vez que os plasmídeos confirmados com as sequências podem ser inseridas em E. coli de expressão por metodologia simples e custo viável. Portanto, no presente trabalho, foi utilizado esse sistema visando objetivos futuros de gerar produtos imunobiológicos que possam ser produzidos de forma recombinante em escala industrial.
Na PCR de colônia, o número de colônias com a presença dos fragmentos genéticos foi considerado suficiente para as etapas subsequentes de cultivo e extração de DNA plasmidial. Esses resultados demonstraram a eficiência das células de E. coli competentes TOP10 nos processos de transformação e replicação dos plasmídeos. Resultados similares foram encontrados por Goudarzi et al. (2009) que clonaram o gene fliC codificador da flagelina e utilizaram células de E. coli competentes TOP10 no sistema de clonagem.
A confirmação das colônias com os fragmentos genéticos foi feita pela técnica de PCR de colônia, conforme realizado por Zhi-Hua et al. (2006) que clonaram um gene codificante de um tipo de HSP. Seron et al. (2004) utilizaram o mesmo sistema de clonagem do presente estudo para o gene da anidrase carbônica, entretanto a confirmação da presença do gene nos clones foi realizada pelo sequenciamento direto das colônias. No presente trabalho, com o objetivo de selecionar com maior segurança as amostras de DNA plasmidial com o vetor e o inserto, foram realizadas a PCR com o DNA plasmidial e o sequenciamento genético dessas.
Diante dos resultados obtidos, o processo de clonagem dos fragmentos genéticos agonistas dos RRPs provenientes de ambas as cepas resultou em amostras de DNA plasmidial com os vetores e insertos na direção correta para a introdução em células de Escherichia coli de expressão. A possibilidade de utilizar esse ensaio experimental para promover a expressão desses genes e avaliação da resposta imune aviária, mediada pela inoculação desses peptídeos são baseadas nos testes in vitro e in vivo para genes imunoestimulantes em mamíferos.
O gene groEL de S. Typhi foi clonado e expresso em hospedeiro heterólogo por Paliwal et al. (2008). Tais pesquisadores adotaram metodologia de cultivo bacteriano, obtenção de DNA genômico e clonagem similares ao do presente trabalho. Posteriormente à expressão da HSP60, foi realizada a inoculação dessa proteína isolada, e em conjunto com um desafio bacteriano em camundongos. A administração individual desse DAMP recombinante levou a produção de maiores níveis de interleucinas e respostas humorais, o que demonstrou a possibilidade dessas proteínas atuarem tanto como moléculas de reconhecimento das respostas imunes inatas e como antígenos bacterianos capazes de gerar respostas imunes adaptativas. Posteriormente, a co-administração da HSP60 com S. Typhimurium detectou o aumento da produção dos títulos de anticorpos da classe IgG, além dos níveis mais elevados de IL-4 e IFN-γ. Esses achados endossam os estudos da utilização de HSPs recombinantes como candidatos imunoestimulantes inespecíficos e vacinais.
Diversas pesquisas reforçam o potencial imunoestimulante da flagelina. A inoculação in vivo desse PAMP, endereçada a sítios como o trato respiratório, resultou no recrutamento de citocinas e moléculas inflamatórias para esses locais (Honko & Mizel 2004). O trabalho desenvolvido por Skountzou et al. (2010) mostrou a eficiência da flagelina recombinante codificada pelo gene fliC de S. Typhimurium no estímulo das respostas imunológicas em camundongos. Nesse estudo, a clonagem do gene fliC em vetores apropriados, permitiu a expressão em células bacterianas de E. coli e purificação dessas proteínas para inoculação intranasal associadas com uma vacina inativada para gripe aviária. Resultados superiores foram encontrados em comparação ao grupo de animais que não receberam a administração de flagelina, como aumento da porcentagem de células inatas recrutadas, maior expressão de TLR5, aumento de citocinas (IFN-γ e TNF-α) e das respostas imunes humorais, além da indução da resposta imune celular com perfil Th2.
A inoculação in vivo do gene fliC em aves foi testada recentemente por Chaung et al. (2012) que administraram a flagelina recombinante concomitante com a vacina inativada da gripe aviária, e obtiveram respostas semelhantes aos resultados obtidos por Skountzou et al. (2010) em camundongos, além da otimização das respostas imunes humorais de mucosa e a detecção inesperada da expressão dos TLR3 em células mononucleares periféricas.
Existem poucos trabalhos que esclarecem as atividades imunoestimulantes do outro gene responsável por condificar a flagelina, o fljB. Pino et al. (2005) evidenciaram que o gene fljB recombinante, co-administrado com antígenos provenientes de Streptococcus mutans em camundongos, levaram as células apresentadoras de antígenos a apresentarem maior expressão de moléculas coestimulatórias (B7-1 e B7-2), que contribuíram no aumento das respostas do tipo Th1 e Th2. Também foi demonstrado que esse gene flagelar tem a capacidade de aumentar tanto as respostas imunitárias humorais sistêmicas como de mucosa, favorecendo sua aplicabilidade como adjuvante.
Em consonância com a tecnologia do DNA recombinante, as etapas procedidas nesse experimento, como a análise in silico dos genes e a clonagem são cruciais para a expressão dessas proteínas e avaliação da resposta imune no hospedeiro. O foco desse ensaio foi iniciar a produção recombinante do PAMP fljB e do DAMP HSP60, uma vez que não existem trabalhos visando esse objetivo com genes provenientes do gênero Salmonella e como alvo o sistema imune inato das aves. Desta forma, concluímos que o sistema de clonagem utilizado neste experimento foi capaz de gerar clones aos fragmentos genéticos das HSP60 e flagelina, o que permite utilizar esse sistema de clonagem na expressão heteróloga desses genes agonistas.
Agradecimentos
À Fundação de Apoio e Pesquisa de Minas Gerais (Fapemig) pelo financiamento do projeto de pesquisa. À Coordenação de Aperfeiçoamento de Pessoal de Nível Superior (CAPES), pela concessão de bolsa de mestrado, e ao Laboratório de Fisiologia Molecular Vegetal pela estrutura disponibilizada para execução do experimento.
Recebido em 27 de setembro de 2013.
Aceito em 7 de janeiro de 2014.
- Altschul S.F., Gish W., Miller W., Myers E.W. & Lipman D.J. 1990.
- Aucouturier J.,
- BLAST 2012. Basic alignment search tool. BLAST assembled refseq genomes. Disponível em <http://www.ncbi.nlm.nih.gov./BLAST> Acesso em 10 dez. 2012.
- Berin M.C.,
- Bianchi M.E. 2007.
- Bonifield H.R. &
- Chaung H.C., Cheng L.T., Hung L.H., Tsai P.C.,
- Dilmaghani M., Ahmadi
- Drexler S.K. &
- Ensgraber M. &
- Gimeno I.M. 2008.
- Goudarzi
- Hall
- Harmala
- Hayashi F., Smith
- Honko A.N. &
- Hu
- Huleatt J.W., Jacobs
- Juul-Madsen H.R.,
- Kaiser P. 2010.
- Karuna P., Yasuyuki
- Lim Y.H., Hirose K.,
- Mazur
- Means T.K., Golenbock
- Metcalfe H.J., Best
- Miyaji
- Moser M. & Leo O.
- Nair S., Lin T.K.,
- Nair V. 2005.
- NCBI 2012. National Center for Biotechnology Information. Disponível em <http://www.ncbi.nlm.nih.gov/> Acesso em 10 dez. 2012.
- Noessner
- Paliwal P.K., Bansal A.,
- Paliwal
- Pino O., Martin M.
- Pockley A.G. 2001.
- Putten J.P.M. van, Bouwman
- Rainczak K., Bajzert
- Ramos H.C., Rumbo M.
- Rebecca S., Wong Y.
- Sanger F., Nicklen S.
- Seron T.J., Hill J.
- Skountzou I., Martin
- Somersan
- Stewart G.R. &
- Takeuchi O. &
- Tapping R.I. 2009. Innate
- Tobian A.A., Canaday
- Wallin R.P.A.,
- Zeng H., Carlson
- Zhi-Hua L., Yang Q.
Datas de Publicação
-
Publicação nesta coleção
16 Maio 2014 -
Data do Fascículo
Mar 2014
Histórico
-
Aceito
07 Jan 2014 -
Recebido
27 Set 2013