RESUMO:
Objetivou-se padronizar uma reação do tipo multiplex PCR (mPCR) para detectar Microsporum canis, Microsporum gypseum e o complexo Trichophyton mentagrophytes em amostras de pelos e/ou crostas de cães e gatos. 250 amostras de pelos e/ou crostas de cães e gatos foram analisadas por meio de exame direto e cultura, o DNA das mesmas foi extraído para mPCR. Primers foram desenhados e como controle positivo da reação utilizou-se o DNA extraído de colônias de M. canis (URM 6273), M. gypseum (URM 6921) e T. mentagrophytes (URM 6211), provenientes da Coleção de Culturas (Micoteca URM), Departamento de Micologia, Centro de Ciências Biológicas da Universidade Federal de Pernambuco (CCB/UFPE). Como controles negativos de reação, utilizou-se água destilada esterilizada e DNA extraído de Alternaria sp. para verificar a especificidade dos primers. Do total de amostras analisadas, 15 (6%) foram identificadas, em cultura, como dermatófitos, e destas, 10 foram M. canis, três M. gypseum e dois T. mentagrophytes (complexo). Destas 15 amostras positivas, 11 (73,3%) foram detectadas por meio da mPCR. Além destas, seis outras, negativas em cultura, foram identificadas como M. gypseum. Verificou-se uma boa concordância entre os resultados da cultura e mPCR (Kappa: 0,66). O protocolo padronizado neste estudo pode ser utilizado como um método de triagem, por apresentar uma sensibilidade maior que a da cultura, usado paralelamente aos exames de rotina, permitindo um diagnóstico em menor tempo.
TERMOS DE INDEXAÇÃO:
Multiplex PCR; dermatófitos; pelos; crostas; cães; gatos; espaçadores transcritos internos; queratina; loci; micoses
ABSTRACT:
The aim of this study was to standardize a multiplex PCR (mPCR) reaction to detect Microsporum canis, Microsporum gypseum and the Trichophyton mentagrophytes complex in dog and cat fur and/or crusts. 250 fur and/or crusts samples from dogs and cats were analyzed by direct examination and culture, DNA from them was extracted for mPCR. Primers were designed and the DNA extracted from colonies of M. canis (URM 6273), M. gypseum (URM 6921) and T. mentagrophytes (URM 6211) from the Collection of Cultures - URM Micoteca - Department of Mycology, Biological Sciences Center of the Federal University of Pernambuco (CCB / UFPE). As negative controls, sterile distilled water and DNA extracted from Alternaria sp., were used to verify the specificity of the primers. Of the total samples analyzed, 15 (6%) were identified in culture as dermatophytes, and of these, 10 were M. canis, three M. gypseum and two T. mentagrophytes (complex). Of these 15 positive samples, 11 (73.3%) were detected by mPCR. Besides these, six others, negative in culture, were identified as M. gypseum. There was good agreement between culture results and mPCR (Kappa: 0.66). The protocol standardized in this study can be used as a screening method, because it has a sensitivity greater than that of the culture, used in parallel to the routine exams, allowing a diagnosis in a shorter time.
INDEX TERMS:
PCR multiplex; dermatophytes; dogs; cats; fur; crusts; internal transcribed spacers; keratin; loci; mycoses
Introdução
A dermatofitose é uma das dermatopatias mais comuns na clínica veterinária e humana, causada por um grupo de fungos que possuem a capacidade de invadir e se desenvolver em estruturas queratinizadas e semiqueratinizadas, os dermatófitos (Liu et al. 2000Liu D., Coloe S., Baird R., & Pedersen J. 2000. Application of PCR to the identification of dermatophyte fungi. J. Med. Microbiol. 49(6):493-497. <http://dx.doi.org/10.1099/0022-1317-49-6-493> <PMid:10847201>
https://doi.org/10.1099/0022-1317-49-6-4...
, Cavalcanti et al. 2002Cavalcanti J.N., Guerra J.L., Gambale W., Corrêa B. & Paula C.R. 2002. Histopathologic and mycologic aspects of experimental infection of guinea pigs with Microsporum canis. Braz. J. Vet. Res. Anim. Sci. 39(5):238-243. <http://dx.doi.org/10.1590/S1413-95962002000500004>
https://doi.org/10.1590/S1413-9596200200...
, Liu et al. 2002Liu D., Pearce L., Lilley G., Coloe S., Baird R. & Pedersen J. 2002. PCR identification of dermatophyte fungi Trichophyton rubrum, T. soudanense and T. gourvilii. J. Med. Microbiol. 51(2):117-122. <http://dx.doi.org/10.1099/0022-1317-51-2-117> <PMid:11863262>
https://doi.org/10.1099/0022-1317-51-2-1...
).
Os gêneros de dermatófitos mais envolvidos em infecções em animais são Microsporum e Trichophyton (Scott et al. 1996Scott D.W., Miller Junior W.H. & Griffin C.E. 1996. Muller & Kirk Dermatologia de Pequenos Animais. Tradução da 5ª ed. Cid Figueiredo, Interlivros, Rio de Janeiro. 1130p., Quinn et al. 2005Quinn P.J., Markey B.K., Carter M.E., Donnelly W.J. & Leonard F.C. 2005. Dermatófitos, p.224-228. In: Ibid. (Eds), Microbiologia Veterinária e Doenças Infecciosas. Artmed, Porto Alegre.). Ainda, de acordo com Balda et al. (2004)Balda A.C., Larsson C.E., Otsuka M. & Gambale W. 2004. Estudo retrospectivo de casuística das dermatofitoses em cães e gatos atendidos no serviço de dermatologia da Faculdade de Medicina Veterinária e Zootecnia da Universidade de São Paulo. Acta Scient. Vet. 32(2):133-140., Microsporum canis, Microsporum gypseum e o complexo Trichophyton mentagrophytes são os principais agentes etiológicos das dermatofitoses em cães e gatos, pois são dotados de alta infectividade e baixa patogenicidade e virulência.
A identificação laboratorial dos dermatófitos depende do seu crescimento em meio de cultura apropriado, devidamente suplementado, para só após, serem avaliados seus aspectos morfológicos macro e microscópicos, no entanto, essa identificação pode ser dificultada devido a não produção de macroconídeos, estruturas chave neste processo (Shehata et al. 2008Shehata A.S., Mukherjee P.K., Aboulatta H.N., El Akhras A.I., Abbadi S.H. & Ghannoum M.A. 2008. Single-Step PCR Using (GACA)4 Primer: utility for rapid identification of dermatophyte species and strains. J. Clin. Microbiol. 46(8):2641-2645. <http://dx.doi.org/10.1128/JCM.00697-08> <PMid:18579714>
https://doi.org/10.1128/JCM.00697-08...
).
Além disso, espécies fúngicas nunca antes isoladas, podem vir a parasitar animais de companhia que passaram a dividir espaço com animais exóticos introduzidos nos lares, o que vai exigir técnicas mais específicas e profissionais mais qualificados (Kim et al. 2011Kim J.Y., Choe Y.B., Ahn K.J. & Lee Y.W. 2011. Identification of dermatophytes using multiplex polymerase chain reaction. Ann. Dermatol. 23(3):304-312. <http://dx.doi.org/10.5021/ad.2011.23.3.304> <PMid:21909200>
https://doi.org/10.5021/ad.2011.23.3.304...
).
Alguns métodos moleculares foram desenvolvidos e vêm contribuindo de maneira significativa na identificação desses agentes e suas espécies, sobretudo a técnica da Reação em Cadeia da Polimerase (PCR), por ser sensível, específica e rápida (Faggi et al. 2001Faggi E., Pini G., Campisi E., Bertellini C., Difonzo E. & Mancianti F. 2001. Application of PCR to distinguish common species of Dermatophytes. J. Clin. Microbiol. 39(9):3382-3385. <http://dx.doi.org/10.1128/JCM.39.9.3382-3385.2001> <PMid:11526185>
https://doi.org/10.1128/JCM.39.9.3382-33...
).
Variações desta técnica têm sido usadas no auxílio da identificação e diferenciação de isolados que não são facilmente identificáveis por meio das técnicas laboratoriais e moleculares convencionais (Gräser et al. 1998Gräser Y., El Fari M., Presber W., Sterry W. & Tietz H.-J. 1998. Identification of common dermatophytes (Trichophyton, Microsporum, Epidermophyton) using polymerase chain reactions. Brit. J. Dermatol. 138(4):576-582. <http://dx.doi.org/10.1046/j.1365-2133.1998.02165.x> <PMid:9640360>
https://doi.org/10.1046/j.1365-2133.1998...
, Jackson et al. 1999Jackson C.J., Barton R.C. & Evans E.G.V. 1999. Species identification and strain differentiation of dermatophyte fungi by analysis of ribosomal-DNA intergenic spacer regions. J. Clin. Microbiol. 37(4):931-936. <PMid:10074504>, Liu et al. 2001Liu D., Pearce L., Lilley G., Coloe S., Baird R. & Pedersen J. 2001. A specific PCR assay for the dermatophyte fungus Microsporum canis. Med. Mycol. 39(2):215-219. <http://dx.doi.org/10.1080/mmy.39.2.215.219> <PMid:11346271>
https://doi.org/10.1080/mmy.39.2.215.219...
, Shehata et al. 2008Shehata A.S., Mukherjee P.K., Aboulatta H.N., El Akhras A.I., Abbadi S.H. & Ghannoum M.A. 2008. Single-Step PCR Using (GACA)4 Primer: utility for rapid identification of dermatophyte species and strains. J. Clin. Microbiol. 46(8):2641-2645. <http://dx.doi.org/10.1128/JCM.00697-08> <PMid:18579714>
https://doi.org/10.1128/JCM.00697-08...
, Mirzahoseini et al. 2009Mirzahoseini H., Omidinia E., Shams-Ghahfarokhi M., Sadeghi G. & Razzaghi-Abyaneh M. 2009. Application of PCR-RFLP to rapid identification of the main pathogenic Dermatophytes from clinical specimens. Iran. J. Public. Health 38(1):18-24., Brillowska-Dabrowska et al. 2013Brillowska-Dabrowska A., MichaŁek E., Saunte D.M.L., SØgaard Nielsen S. & Arendrup M.C. 2013. PCR test for Microsporum canis identification. Med. Mycol. 51(6):576-579. <http://dx.doi.org/10.3109/13693786.2012.755741> <PMid:23294424>
https://doi.org/10.3109/13693786.2012.75...
, Leal et al. 2017Leal C.A.S., Kim P.C.P., Mota A.R., Lima D.C.V., Gomes A.L.V., Pinheiro Júnior J.W., Silva L.B.G. & Mota R.A. 2017. PCR-RFLP: uma alternativa a cultura (padrão Ouro) no diagnóstico de dermatófitos em cães e gatos. Pubvet 11(2):181-186. <http://dx.doi.org/10.22256/pubvet.v11n2.181-186>
https://doi.org/10.22256/pubvet.v11n2.18...
).
A Multiplex PCR (mPCR) é outra variação da PCR convencional utilizada no diagnóstico de microrganismos, que se baseia no uso de dois ou mais pares de primers na mesma reação, permitindo a amplificação simultânea de mais de uma sequência de DNA-alvo presente na amostra analisada, de maneira específica, barateando e deixando a análise mais ampla e com economia de tempo (Silva 2008Silva M.A. 2008. Utilização de PCR multiplex para o diagnóstico etiológico da mastite bovina, Brasil. Dissertação de Mestrado, Escola de Veterinária, Universidade Federal de Minas Gerais, Belo Horizonte. 32p.).
Multiplex PCRs foram desenvolvidas e utilizadas para identificação de espécies de dermatófitos utilizando DNA extraído de culturas e diretamente de amostras de raspados de pele em humanos (Brillowska-Dabrowska et al. 2007Brillowska-Dabrowska A., Saunte D.M. & Arendrup M.C. 2007. Five-hour diagnosis of dermatophyte nail infections with specific detection of Trichophyton rubrum. J. Clin. Microbiol. 45(4):1200-1204. <http://dx.doi.org/10.1128/JCM.02072-06> <PMid:17267633>
https://doi.org/10.1128/JCM.02072-06...
, Kim et al. 2011Kim J.Y., Choe Y.B., Ahn K.J. & Lee Y.W. 2011. Identification of dermatophytes using multiplex polymerase chain reaction. Ann. Dermatol. 23(3):304-312. <http://dx.doi.org/10.5021/ad.2011.23.3.304> <PMid:21909200>
https://doi.org/10.5021/ad.2011.23.3.304...
, Dhib et al. 2014Dhib I., Fathallah A., Yaacoub A., Hadj Slama F., Said M.B. & Zemni R. 2014. Multiplex PCR assay for the detection of common dermatophyte nail infections. Mycoses 57(1):19-26. <http://dx.doi.org/10.1111/myc.12096> <PMid:23734576>
https://doi.org/10.1111/myc.12096...
, Mehlig et al. 2014Mehlig L., Garve C., Ritschel A., Zeiler A., Brabetz W., Weber C. & Bauer A. 2014. Clinical evaluation of a novel commercial multiplex-based PCR diagnostic test for differential diagnosis of dermatomycoses. Mycoses 57(1):27-34. <http://dx.doi.org/10.1111/myc.12097> <PMid:23734586>
https://doi.org/10.1111/myc.12097...
, Spiliopoulou et al. 2015Spiliopoulou A., Bartzavali C., Jelastopulu E., Anastassiou E.D. & Christofidou M. 2015. Evaluation of a commercial PCR test for the diagnosis of dermatophyte nail infections. J. Med. Microbiol. 64(Pt 1):25-31. <http://dx.doi.org/10.1099/jmm.0.079962-0> <PMid:25418736>
https://doi.org/10.1099/jmm.0.079962-0...
), demonstrando o potencial da técnica, no entanto, pesquisas que tenham por objetivo o desenvolvimento deste tipo de técnica para a detecção de dermatófitos em animais, são escassas(Kano et al. 2003Kano R., Hirai A., Muramatsu M., Watari T. & Hasegawa A. 2003. Direct detection of dermatophytes in skin samples based on sequences of the chitin synthase 1 (CHS1) gene. J. Vet. Med. Sci. 65(2):267-270. <http://dx.doi.org/10.1292/jvms.65.267> <PMid:12655126>
https://doi.org/10.1292/jvms.65.267...
, Cafarchia et al. 2013Cafarchia C., Gasser R.B., Figueredo L.A., Weigl S., Danesi P., Capelli G. & Otranto D. 2013. An improved molecular diagnostic assay for canine and feline dermatophytosis. Med. Mycol. 51(2):136-143. <http://dx.doi.org/10.3109/13693786.2012.691995> <PMid:22686247>
https://doi.org/10.3109/13693786.2012.69...
, Dabrowska et al. 2014Dabrowska I., Dworecka-Kaszak B. & Brillowska-Dąbrowska A. 2014. The use of a one-step PCR method for the identification of Microsporum canis and Trichophyton mentagrophytes infection of pets. Acta Biochim. Pol. 61(2):375-378. <PMid:24945136>).
Objetivou-se neste estudo padronizar uma reação do tipo multiplex PCR (mPCR) para detecção de Microsporum canis, Microsporum gypseum e Trichophyton mentagrophytes em amostras de pelos e/ou crostas de cães e gatos.
Material e Métodos
Local da pesquisa e amostragem. A pesquisa foi desenvolvida no Laboratório de Doenças Infecciosas dos Animais Domésticos, Universidade Federal Rural de Pernambuco (DMV/UFRPE). O projeto foi submetido e aprovado pela Comissão de Ética no Uso de Animais da Universidade Federal Rural de Pernambuco, Recife, Brasil (Licença nº 003/2014).
Neste estudo foram analisadas 250 amostras de pelos e/ou crostas de cães e gatos, com dermatopatias, atendidos no Hospital Veterinário da UFRPE e encaminhados ao laboratório para diagnóstico.
Exame direto. Utilizou-se o hidróxido de potássio (KOH) (30%) para clarificação dos pelos e escamas, objetivando a identificação de estruturas de parasitismo e artroconídios (Scott et al. 1996Scott D.W., Miller Junior W.H. & Griffin C.E. 1996. Muller & Kirk Dermatologia de Pequenos Animais. Tradução da 5ª ed. Cid Figueiredo, Interlivros, Rio de Janeiro. 1130p.).
Cultura. Todas as 250 amostras foram cultivadas, independente do resultado do exame direto, em placas de Petri contendo ágar dextrose Sabouraud, adicionado de extrato de levedura, cloranfenicol e ciclo-heximida. As placas foram incubadas em ambiente de aerobiose em temperatura ambiente (25-27°C) e examinadas diariamente por até cinco semanas (Cruz 2010Cruz L.C.H. 2010. Dermatófitos, p.111-142. In: Ibid. (Ed.), Micologia Veterinária. 2ª ed. Revinter, Rio de Janeiro.).
Extração do DNA. O DNA das 250 amostras foi extraído utilizando-se o kit DNeasy Blood & Tissue (QIAGEN®, Hilden, Germany) de acordo com o protocolo estabelecido pelo fabricante para tecidos em geral, com algumas modificações: o protocolo recomenda um tempo máximo de incubação, em banho seco com agitação (1500rpm) à 56°C, de 40 minutos, por se tratar de um grupo de fungos que possui quitina na parede celular; este tempo foi aumentado para 1h; outro acréscimo feito ao protocolo foi a adição, após este período, de 4μL de RNase à suspensão, visando a purificação do DNA; outra modificação realizada foi a redução do volume da solução tampão AE (eluente) de 200μL para 100μL, já que a quantidade de DNA fúngico presente na amostra do pelo ou crosta é pequena.
Como controle positivo da reação utilizou-se o DNA extraído das colônias de M. canis (URM 6273), M. gypseum (URM 6921) e T. mentagrophytes (URM 6211), provenientes da Coleção de Culturas (Micoteca URM), Departamento de Micologia, Centro de Ciências Biológicas, Universidade Federal de Pernambuco (CCB/UFPE).
Como controles negativos de reação, utilizou-se água destilada esterilizada e DNA extraído de Alternaria sp. para verificar a especificidade dos primers.
Desenho dos primers. Foram selecionadas sequências de referência para as espécies alvo a partir de um estudo no banco virtual (GenBank/NCBI) da região ITS, na sua porção mais variável, utilizando-se o programa Primer 3 do NCBI que sugeriu sequências compostas de 18 a 25 bases para serem empregadas como iniciadores, respeitando-se as condições para que uma reação de Multiplex PCR possa ser realizada. Estes fragmentos foram testados quanto à especificidade pelo alinhamento local (BLAST/NCBI) frente às sequências nucleotídicas de inúmeros organismos depositados no “GenBank”. O software OligoAnalyzer1.0.3 também foi utilizado para verificação de possíveis alinhamentos inespecíficos.
Após todas as análises, os primers foram selecionados e estão demonstrados no Quadro 1.
Sequências nucleotídicas dos primers desenhados a partir das regiões gênicas ITS 1 e 2 e do gene 5.8S rRNA, utilizados neste estudo
Amplificação do DNA. As reações de amplificação do DNA foram otimizadas para um volume final de 15μL, contendo: 3μL de DNA genômico; 0,25μL de cada primer (três pares) a 10μM (Quadro 1); 3μL de Água ultrapura e 7,5 μL de Top Taq™ Master Mix (QIAGEN®, Hilden, Germany).
O protocolo térmico utilizado para a amplificação foi o seguinte: desnaturação inicial a 94°C por 7 minutos seguida por 30 ciclos de 95°C por 30 segundos, 60°C por 1 minuto e 72°C por 30 segundos, finalizando com uma extensão final a 72°C por 6 minutos. Após a amplificação, o produto de PCR foi plotado em gel de agarose 3%, corado com Blue Green LoadingDye I (LGC Biotecnologia) e colocado em cuba de eletroforese com tampão Tris-acetato-EDTA (1X) (Amresco®) para posterior visualização sob a luz ultravioleta e fotodocumentação.
Análise estatística. Para o estudo de concordância entre os testes foi utilizado o coeficiente Kappa (K) e a interpretação convencional dos valores K adotada foi: 0,00-0,20 = concordância fraca; 0,21-0,40 = regular; 0,41-0,60 = moderada; 0,61-0,80 = boa; 0,81-1,00 = muito boa. Valores negativos foram interpretados como equivalentes a 0,00 (Landis & Koch 1977Landis J.R. & Koch G.G. 1977. The measurement of observer agreement for categorical data. Biometrics 33(1):159-174. <http://dx.doi.org/10.2307/2529310> <PMid:843571>
https://doi.org/10.2307/2529310...
). Também foram realizados cálculos de sensibilidade e especificidade conforme Pereira (2003)Pereira M.G. 2003. Epidemiologia Teoria e Prática. 7ª reimpr. Guanabara Koogan, Rio de Janeiro. 596p..
Para o cálculo de concordância foi utilizado o programa computacional Bioestat versão 5.0 (Ayres et al. 2007Ayres M., Ayres Júnior M., Ayres D.L. & Santos A.S. 2007. BioEstat 5.0: aplicações estatísticas nas áreas das ciências biológicas e médicas. MCT, IDSM, CNPq, Belém. 364p.).
Resultados
Das 250 amostras analisadas, 15 (6%) foram identificadas, em cultura, como dermatófitos, e destas, 10 foram Microsporum canis, três M. gypseum e duas como pertencentes ao complexo Trichophyton mentagrophytes.
Destas 15 amostras positivas, 11 (73,3%) foram detectadas por meio da mPCR, e além destas, seis outras, negativas em cultura, foram identificadas como M. gypseum.
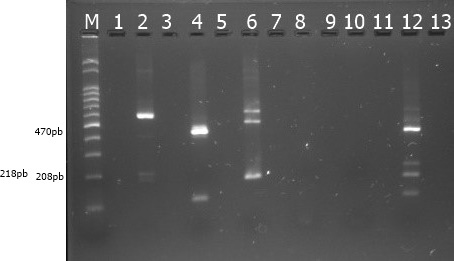
O Quadro 2 apresenta as análises estatísticas dos resultados observados na cultura e na mPCR. A Figura 1 demonstra os diferentes fragmentos amplificados no protocolo padronizado.
Multiplex PCR em gel de agarose 3%. M = Marcador molecular 100pb (LGC biotecnologia); 1, 3, 5, 7, 9, 11 e 13 = poços vazios; 2 =Microsporum canis (URM 6273); 4 = M. gypseum (URM 6921); 6 = Trichophyton mentagrophytes (URM 6211); 8 = Alternaria sp.; 10 = controle negativo (H2O destilada esterilizada); 12 = reação multiplex (mix DNA).
Discussão
Na Figura 1 observam-se as reações de amplificação do tipo Multiplex PCR para Microsporum canis, M. gypseum e Trichophyton mentagrophytes (complexo), que segundo Rodrigues et al. (2006)Rodrigues J.J.S., Silva R.C. & Siqueira M.M. 2006. Técnicas de biologia molecular aplicadas ao diagnóstico, p.16-40. In: Rosseti M.L., Silva C.M.D. & Rodrigues J.J.S. (Eds), Doenças Infecciosas: diagnóstico molecular. Guanabara Koogan, Rio de Janeiro., é aquela na qual vários loci são amplificados ao mesmo tempo e no mesmo frasco, proporcionando economia de tempo, reagentes e dando mais rapidez ao resultado.
As regiões ITS além de serem relativamente curtas, aparecem em grande número de cópias no genoma e isto pode ser verificado nos resultados obtidos. Isto confirma que os amplicons observados (Fig.1) tratam-se realmente de sequências contidas na região-alvo (ITS 1 e 2) utilizada em vários estudos moleculares para diferenciação entre espécies de fungos (Fungaro 2001Fungaro M.H.P. 2001. PCR na micologia: diagnóstico e análise de variabilidade. Biotecnol. Ciênc. Desenvolv. 14:12-16.).
No poço 12 foram incluídos, no mesmo tubo, os três pares de primers desenhados e o DNA das três espécies pesquisadas (mix de DNAs), acrescidos do DNA de Alternaria sp., que serviu como um segundo controle negativo. Após eletroforese foi possível verificar a amplificação de todos os fragmentos que correspondiam a detecção dos fungos presentes na amostra.
Kim et al. (2011)Kim J.Y., Choe Y.B., Ahn K.J. & Lee Y.W. 2011. Identification of dermatophytes using multiplex polymerase chain reaction. Ann. Dermatol. 23(3):304-312. <http://dx.doi.org/10.5021/ad.2011.23.3.304> <PMid:21909200>
https://doi.org/10.5021/ad.2011.23.3.304...
desenharam três pares de primers visando desenvolver uma mPCR para detectar onze espécies de dermatófitos em DNA extraído de raspados de pele de pacientes humanos com diagnóstico clínico de dermatofitose.
No entanto, foram necessárias três reações, em separado, com protocolos térmicos individualizados, para em conjunto, identificar o fungo presente na amostra analisada. Fato que não ocorreu no nosso estudo, onde foi possível diferenciar as três espécies pesquisadas numa única reação e inclusive detectar as mesmas, presentes simultaneamente.
Outro ponto que difere este protocolo daquele desenvolvido por Kim et al. (2011)Kim J.Y., Choe Y.B., Ahn K.J. & Lee Y.W. 2011. Identification of dermatophytes using multiplex polymerase chain reaction. Ann. Dermatol. 23(3):304-312. <http://dx.doi.org/10.5021/ad.2011.23.3.304> <PMid:21909200>
https://doi.org/10.5021/ad.2011.23.3.304...
é o volume final de cada reação, onde padronizaram 50μL para cada reação, logo, em três reações diferentes, necessárias para a identificação do fungo, totaliza-se 150μL, sendo dez vezes maior que o volume final padronizado neste trabalho.
Dhib et al. (2014)Dhib I., Fathallah A., Yaacoub A., Hadj Slama F., Said M.B. & Zemni R. 2014. Multiplex PCR assay for the detection of common dermatophyte nail infections. Mycoses 57(1):19-26. <http://dx.doi.org/10.1111/myc.12096> <PMid:23734576>
https://doi.org/10.1111/myc.12096...
também desenvolveram uma mPCR com o objetivo de identificar e diferenciar espécies do complexo T. mentagrophytes e T. rubrum em tineas e onicomicoses humanas, a partir dos iniciadores desenhados. Da mesma forma que a anterior, a reação ocorre em três etapas e o volume total da reação também foi de 50μL por etapa, totalizando 150μL.
Outras pesquisas foram desenvolvidas utilizando a mPCR em comparação com técnicas de rotina para detecção de espécies de dermatófitos diretamente de amostras clínicas humanas (Brillowska-Dabrowska et al. 2007Brillowska-Dabrowska A., Saunte D.M. & Arendrup M.C. 2007. Five-hour diagnosis of dermatophyte nail infections with specific detection of Trichophyton rubrum. J. Clin. Microbiol. 45(4):1200-1204. <http://dx.doi.org/10.1128/JCM.02072-06> <PMid:17267633>
https://doi.org/10.1128/JCM.02072-06...
, Mehlig et al. 2014Mehlig L., Garve C., Ritschel A., Zeiler A., Brabetz W., Weber C. & Bauer A. 2014. Clinical evaluation of a novel commercial multiplex-based PCR diagnostic test for differential diagnosis of dermatomycoses. Mycoses 57(1):27-34. <http://dx.doi.org/10.1111/myc.12097> <PMid:23734586>
https://doi.org/10.1111/myc.12097...
, Spiliopoulou et al. 2015Spiliopoulou A., Bartzavali C., Jelastopulu E., Anastassiou E.D. & Christofidou M. 2015. Evaluation of a commercial PCR test for the diagnosis of dermatophyte nail infections. J. Med. Microbiol. 64(Pt 1):25-31. <http://dx.doi.org/10.1099/jmm.0.079962-0> <PMid:25418736>
https://doi.org/10.1099/jmm.0.079962-0...
), e em todas observou-se um aumento da sensibilidade nos resultados, fato também verificado neste estudo, onde seis amostras de DNA extraído de pelos, que em cultura tiveram resultado negativo, apresentaram um fragmento compatível com M. gypseum.
Este protocolo padronizado, atendeu a todos os pré-requisitos citados por Rodrigues et al. (2006)Rodrigues J.J.S., Silva R.C. & Siqueira M.M. 2006. Técnicas de biologia molecular aplicadas ao diagnóstico, p.16-40. In: Rosseti M.L., Silva C.M.D. & Rodrigues J.J.S. (Eds), Doenças Infecciosas: diagnóstico molecular. Guanabara Koogan, Rio de Janeiro., onde além de economizar tempo e reagentes, conseguiu diferenciar as três espécies pesquisadas numa reação única e ainda elevou a sensibilidade no diagnóstico, comprovando ser uma ferramenta de grande utilidade na rotina laboratorial.
Dhib et al. (2014)Dhib I., Fathallah A., Yaacoub A., Hadj Slama F., Said M.B. & Zemni R. 2014. Multiplex PCR assay for the detection of common dermatophyte nail infections. Mycoses 57(1):19-26. <http://dx.doi.org/10.1111/myc.12096> <PMid:23734576>
https://doi.org/10.1111/myc.12096...
, recomendaram a utilização da mPCR, devido a sua alta sensibilidade, também em casos onde a cultura é negativa ou ocorre contaminação da mesma, dificultando ou impossibilitando a identificação do agente patogênico.
Dessa forma, o desenvolvimento de protocolos moleculares capazes de detectar a presença de agentes causadores de enfermidades, principalmente zoonóticas, de forma mais rápida numa amostra encaminhada para análise, pode contribuir bastante para o avanço da área estudada, assim como para o tratamento e controle das mesmas.
E em se tratando do estreito contato dos cães e gatos com os humanos e da escassez de pesquisas na área da micologia veterinária, pesquisas como estas, são de grande importância para a saúde animal e, porque não dizer, para saúde humana também.
Conclusão
O protocolo padronizado neste estudo pode ser utilizado como um método de triagem, por apresentar uma sensibilidade maior que a da cultura, usado paralelamente aos exames de rotina, permitindo um diagnóstico em menor tempo.
Agradecimentos
À Fundação de Amparo à Ciência e Tecnologia de Pernambuco (FACEPE) pela bolsa concedida.
Referências
- Ayres M., Ayres Júnior M., Ayres D.L. & Santos A.S. 2007. BioEstat 5.0: aplicações estatísticas nas áreas das ciências biológicas e médicas. MCT, IDSM, CNPq, Belém. 364p.
- Balda A.C., Larsson C.E., Otsuka M. & Gambale W. 2004. Estudo retrospectivo de casuística das dermatofitoses em cães e gatos atendidos no serviço de dermatologia da Faculdade de Medicina Veterinária e Zootecnia da Universidade de São Paulo. Acta Scient. Vet. 32(2):133-140.
- Brillowska-Dabrowska A., Saunte D.M. & Arendrup M.C. 2007. Five-hour diagnosis of dermatophyte nail infections with specific detection of Trichophyton rubrum J. Clin. Microbiol. 45(4):1200-1204. <http://dx.doi.org/10.1128/JCM.02072-06> <PMid:17267633>
» https://doi.org/10.1128/JCM.02072-06 - Brillowska-Dabrowska A., MichaŁek E., Saunte D.M.L., SØgaard Nielsen S. & Arendrup M.C. 2013. PCR test for Microsporum canis identification. Med. Mycol. 51(6):576-579. <http://dx.doi.org/10.3109/13693786.2012.755741> <PMid:23294424>
» https://doi.org/10.3109/13693786.2012.755741 - Cafarchia C., Gasser R.B., Figueredo L.A., Weigl S., Danesi P., Capelli G. & Otranto D. 2013. An improved molecular diagnostic assay for canine and feline dermatophytosis. Med. Mycol. 51(2):136-143. <http://dx.doi.org/10.3109/13693786.2012.691995> <PMid:22686247>
» https://doi.org/10.3109/13693786.2012.691995 - Cavalcanti J.N., Guerra J.L., Gambale W., Corrêa B. & Paula C.R. 2002. Histopathologic and mycologic aspects of experimental infection of guinea pigs with Microsporum canis Braz. J. Vet. Res. Anim. Sci. 39(5):238-243. <http://dx.doi.org/10.1590/S1413-95962002000500004>
» https://doi.org/10.1590/S1413-95962002000500004 - Cruz L.C.H. 2010. Dermatófitos, p.111-142. In: Ibid. (Ed.), Micologia Veterinária. 2ª ed. Revinter, Rio de Janeiro.
- Dabrowska I., Dworecka-Kaszak B. & Brillowska-Dąbrowska A. 2014. The use of a one-step PCR method for the identification of Microsporum canis and Trichophyton mentagrophytes infection of pets. Acta Biochim. Pol. 61(2):375-378. <PMid:24945136>
- Dhib I., Fathallah A., Yaacoub A., Hadj Slama F., Said M.B. & Zemni R. 2014. Multiplex PCR assay for the detection of common dermatophyte nail infections. Mycoses 57(1):19-26. <http://dx.doi.org/10.1111/myc.12096> <PMid:23734576>
» https://doi.org/10.1111/myc.12096 - Faggi E., Pini G., Campisi E., Bertellini C., Difonzo E. & Mancianti F. 2001. Application of PCR to distinguish common species of Dermatophytes. J. Clin. Microbiol. 39(9):3382-3385. <http://dx.doi.org/10.1128/JCM.39.9.3382-3385.2001> <PMid:11526185>
» https://doi.org/10.1128/JCM.39.9.3382-3385.2001 - Fungaro M.H.P. 2001. PCR na micologia: diagnóstico e análise de variabilidade. Biotecnol. Ciênc. Desenvolv. 14:12-16.
- Gräser Y., El Fari M., Presber W., Sterry W. & Tietz H.-J. 1998. Identification of common dermatophytes (Trichophyton, Microsporum, Epidermophyton) using polymerase chain reactions. Brit. J. Dermatol. 138(4):576-582. <http://dx.doi.org/10.1046/j.1365-2133.1998.02165.x> <PMid:9640360>
» https://doi.org/10.1046/j.1365-2133.1998.02165.x - Jackson C.J., Barton R.C. & Evans E.G.V. 1999. Species identification and strain differentiation of dermatophyte fungi by analysis of ribosomal-DNA intergenic spacer regions. J. Clin. Microbiol. 37(4):931-936. <PMid:10074504>
- Kano R., Hirai A., Muramatsu M., Watari T. & Hasegawa A. 2003. Direct detection of dermatophytes in skin samples based on sequences of the chitin synthase 1 (CHS1) gene. J. Vet. Med. Sci. 65(2):267-270. <http://dx.doi.org/10.1292/jvms.65.267> <PMid:12655126>
» https://doi.org/10.1292/jvms.65.267 - Kim J.Y., Choe Y.B., Ahn K.J. & Lee Y.W. 2011. Identification of dermatophytes using multiplex polymerase chain reaction. Ann. Dermatol. 23(3):304-312. <http://dx.doi.org/10.5021/ad.2011.23.3.304> <PMid:21909200>
» https://doi.org/10.5021/ad.2011.23.3.304 - Landis J.R. & Koch G.G. 1977. The measurement of observer agreement for categorical data. Biometrics 33(1):159-174. <http://dx.doi.org/10.2307/2529310> <PMid:843571>
» https://doi.org/10.2307/2529310 - Leal C.A.S., Kim P.C.P., Mota A.R., Lima D.C.V., Gomes A.L.V., Pinheiro Júnior J.W., Silva L.B.G. & Mota R.A. 2017. PCR-RFLP: uma alternativa a cultura (padrão Ouro) no diagnóstico de dermatófitos em cães e gatos. Pubvet 11(2):181-186. <http://dx.doi.org/10.22256/pubvet.v11n2.181-186>
» https://doi.org/10.22256/pubvet.v11n2.181-186 - Liu D., Coloe S., Baird R., & Pedersen J. 2000. Application of PCR to the identification of dermatophyte fungi. J. Med. Microbiol. 49(6):493-497. <http://dx.doi.org/10.1099/0022-1317-49-6-493> <PMid:10847201>
» https://doi.org/10.1099/0022-1317-49-6-493 - Liu D., Pearce L., Lilley G., Coloe S., Baird R. & Pedersen J. 2001. A specific PCR assay for the dermatophyte fungus Microsporum canis Med. Mycol. 39(2):215-219. <http://dx.doi.org/10.1080/mmy.39.2.215.219> <PMid:11346271>
» https://doi.org/10.1080/mmy.39.2.215.219 - Liu D., Pearce L., Lilley G., Coloe S., Baird R. & Pedersen J. 2002. PCR identification of dermatophyte fungi Trichophyton rubrum, T. soudanense and T. gourvilii J. Med. Microbiol. 51(2):117-122. <http://dx.doi.org/10.1099/0022-1317-51-2-117> <PMid:11863262>
» https://doi.org/10.1099/0022-1317-51-2-117 - Mehlig L., Garve C., Ritschel A., Zeiler A., Brabetz W., Weber C. & Bauer A. 2014. Clinical evaluation of a novel commercial multiplex-based PCR diagnostic test for differential diagnosis of dermatomycoses. Mycoses 57(1):27-34. <http://dx.doi.org/10.1111/myc.12097> <PMid:23734586>
» https://doi.org/10.1111/myc.12097 - Mirzahoseini H., Omidinia E., Shams-Ghahfarokhi M., Sadeghi G. & Razzaghi-Abyaneh M. 2009. Application of PCR-RFLP to rapid identification of the main pathogenic Dermatophytes from clinical specimens. Iran. J. Public. Health 38(1):18-24.
- Pereira M.G. 2003. Epidemiologia Teoria e Prática. 7ª reimpr. Guanabara Koogan, Rio de Janeiro. 596p.
- Quinn P.J., Markey B.K., Carter M.E., Donnelly W.J. & Leonard F.C. 2005. Dermatófitos, p.224-228. In: Ibid. (Eds), Microbiologia Veterinária e Doenças Infecciosas. Artmed, Porto Alegre.
- Rodrigues J.J.S., Silva R.C. & Siqueira M.M. 2006. Técnicas de biologia molecular aplicadas ao diagnóstico, p.16-40. In: Rosseti M.L., Silva C.M.D. & Rodrigues J.J.S. (Eds), Doenças Infecciosas: diagnóstico molecular. Guanabara Koogan, Rio de Janeiro.
- Scott D.W., Miller Junior W.H. & Griffin C.E. 1996. Muller & Kirk Dermatologia de Pequenos Animais. Tradução da 5ª ed. Cid Figueiredo, Interlivros, Rio de Janeiro. 1130p.
- Shehata A.S., Mukherjee P.K., Aboulatta H.N., El Akhras A.I., Abbadi S.H. & Ghannoum M.A. 2008. Single-Step PCR Using (GACA)4 Primer: utility for rapid identification of dermatophyte species and strains. J. Clin. Microbiol. 46(8):2641-2645. <http://dx.doi.org/10.1128/JCM.00697-08> <PMid:18579714>
» https://doi.org/10.1128/JCM.00697-08 - Silva M.A. 2008. Utilização de PCR multiplex para o diagnóstico etiológico da mastite bovina, Brasil. Dissertação de Mestrado, Escola de Veterinária, Universidade Federal de Minas Gerais, Belo Horizonte. 32p.
- Spiliopoulou A., Bartzavali C., Jelastopulu E., Anastassiou E.D. & Christofidou M. 2015. Evaluation of a commercial PCR test for the diagnosis of dermatophyte nail infections. J. Med. Microbiol. 64(Pt 1):25-31. <http://dx.doi.org/10.1099/jmm.0.079962-0> <PMid:25418736>
» https://doi.org/10.1099/jmm.0.079962-0
Datas de Publicação
-
Publicação nesta coleção
Set 2018
Histórico
-
Recebido
22 Jul 2017 -
Aceito
01 Nov 2017