Resumos
No intuito de avaliar a magnitude e a distribuição da variabilidade genética existente em populações naturais de araticunzeiro, seis populações oriundas de duas regiões do Estado de Goiás foram amostradas (30 indivíduos cada) e analisadas para quatro sistemas enzimáticos: 6-Fosfogluconato Desidrogenase (6PGD), Fosfoglucomutase (PGM), Malato Desidrogenase (MDH) e Leucina Aminopeptidase (LAP). Dois locos diméricos foram encontrados para a enzima 6PGD, e um loco monomérico para os demais sistemas. Somente a enzima MDH apresentou monomorfismo em todas as populações, sendo que o número médio de alelos por loco polimórfico foi igual a dois. A heterozigosidade total média foi de 0,357, denotando a existência de elevada variabilidade genética na região para esta espécie. Cerca de 19% da variação genética total foram devidos a diferenças interpopulacionais, indicando uma elevada divergência genética entre as populações. A análise de variância das freqüências alélicas para os locos polimórficos no conjunto de populações evidenciou um elevado grau de estruturação genética em nível populacional, tendo-se observado coeficientes significativos para o parentesco entre indivíduos dentro de populações e para a endogamia total em nível individual. A avaliação da divergência genética entre as populações sugeriu a existência de um efeito da distribuição espacial sobre a magnitude da similaridade entre as mesmas. Os resultados sugerem que a espécie se reproduz preferencialmente por alogamia, em conformidade com achados de outros autores.
Annona; caracterização genética; cerrado; isoenzima
The level and distribution of the genetic variability in natural populations of "araticunzeiro" were estimated using a sample of six natural populations, collected in two different regions of the State of Goiás, Brazil. Samples comprising 30 individuals from each population were genetically evaluated for four allozyme loci: 6-Phosphogluconate Dehydrogenase (6PGD), Phosphoglucomutase (PGM), Malate Dehydrogenase (MDH) and Leucine Aminopeptidase (LAP). Two dimeric loci were detected for the 6PGD isozyme system while all the other systems evaluated showed typical monomeric loci pattern. No polymorphism was detected for the MDH enzymatic system. The estimated average number of alleles per polymorphic loci was two. The estimated total heterozygosity (averaged across loci) was 0.357, suggesting that a high level of genetic variability is present in the species along the sampled geographic region. About 19% of the total genetic variation was found to be due to differences at the population level, suggesting the existence of a high genetic divergence among populations. Accordingly, the analysis of variance of allelic frequencies for each polymorphic loci showed a strong genetic structure at the population level. Significant values for the average coefficients of coancestry among individuals within populations and for total inbreeding were found. The genetic divergences between pairs of populations showed to be partially correlated to the geographic distances between pair members. Results obtained here suggested that A. crassiflora is preferentially alogamous, in conformity with other authors findings.
Annona; cerrado; genetic characterization; isozyme
Caracterização genética de populações naturais de araticunzeiro (Annona crassiflora Mart. - Annonaceae) no Estado de Goiás
Genetic characterization of natural populations of "araticunzeiro" (Annona crassiflora Mart. - Annonaceae) collected in the State of Goiás, Brazil
Mariana P.C. TellesI; Fabrizio D. ValvaII; Ludmila F. BandeiraII; Alexandre S.G. CoelhoII,1 1 Autor para correspondência: acoelho@icb1.ufg.br
IUniversidade Católica de Goiás, Departamento de Zootecnia, Caixa Postal 86, 74605-010 Goiânia, GO, Brasil
IIUniversidade Federal de Goiás, Instituto de Ciências Biológicas, Departamento de Biologia Geral, Caixa Postal 131, 74001970 Goiânia, GO, Brasil
RESUMO
No intuito de avaliar a magnitude e a distribuição da variabilidade genética existente em populações naturais de araticunzeiro, seis populações oriundas de duas regiões do Estado de Goiás foram amostradas (30 indivíduos cada) e analisadas para quatro sistemas enzimáticos: 6-Fosfogluconato Desidrogenase (6PGD), Fosfoglucomutase (PGM), Malato Desidrogenase (MDH) e Leucina Aminopeptidase (LAP). Dois locos diméricos foram encontrados para a enzima 6PGD, e um loco monomérico para os demais sistemas. Somente a enzima MDH apresentou monomorfismo em todas as populações, sendo que o número médio de alelos por loco polimórfico foi igual a dois. A heterozigosidade total média foi de 0,357, denotando a existência de elevada variabilidade genética na região para esta espécie. Cerca de 19% da variação genética total foram devidos a diferenças interpopulacionais, indicando uma elevada divergência genética entre as populações. A análise de variância das freqüências alélicas para os locos polimórficos no conjunto de populações evidenciou um elevado grau de estruturação genética em nível populacional, tendo-se observado coeficientes significativos para o parentesco entre indivíduos dentro de populações e para a endogamia total em nível individual. A avaliação da divergência genética entre as populações sugeriu a existência de um efeito da distribuição espacial sobre a magnitude da similaridade entre as mesmas. Os resultados sugerem que a espécie se reproduz preferencialmente por alogamia, em conformidade com achados de outros autores.
Palavras-chave:Annona, caracterização genética, cerrado, isoenzima
ABSTRACT
The level and distribution of the genetic variability in natural populations of "araticunzeiro" were estimated using a sample of six natural populations, collected in two different regions of the State of Goiás, Brazil. Samples comprising 30 individuals from each population were genetically evaluated for four allozyme loci: 6-Phosphogluconate Dehydrogenase (6PGD), Phosphoglucomutase (PGM), Malate Dehydrogenase (MDH) and Leucine Aminopeptidase (LAP). Two dimeric loci were detected for the 6PGD isozyme system while all the other systems evaluated showed typical monomeric loci pattern. No polymorphism was detected for the MDH enzymatic system. The estimated average number of alleles per polymorphic loci was two. The estimated total heterozygosity (averaged across loci) was 0.357, suggesting that a high level of genetic variability is present in the species along the sampled geographic region. About 19% of the total genetic variation was found to be due to differences at the population level, suggesting the existence of a high genetic divergence among populations. Accordingly, the analysis of variance of allelic frequencies for each polymorphic loci showed a strong genetic structure at the population level. Significant values for the average coefficients of coancestry among individuals within populations and for total inbreeding were found. The genetic divergences between pairs of populations showed to be partially correlated to the geographic distances between pair members. Results obtained here suggested that A. crassiflora is preferentially alogamous, in conformity with other authors findings.
Key words:Annona, "cerrado", genetic characterization, isozyme
Introdução
Os marcadores moleculares (isoenzimáticos, protéicos e de DNA) têm sido amplamente utilizados na caracterização genética de populações naturais de plantas e animais, por permitirem a obtenção de um grande número de informações com um esforço relativamente pequeno (Hillis et al. 1996, Ferreira & Grattapaglia 1998, Alfenas et al. 1998). Uma característica importante destes estudos é o fato de que estes marcadores são considerados neutros do ponto de vista evolutivo (Sunnucks 2000), sendo este um pressuposto básico em algumas análises de divergência entre populações, apesar da controvérsia existente sobre a neutralidade de alguns sistemas enzimáticos (Gray 1996).
Os parâmetros genéticos populacionais estimados com base em marcadores podem ser utilizados para diversos fins. Quando o objetivo é a conservação de espécies importantes, ou de espécies que estão inseridas em biomas que devem ser preservados, estes parâmetros podem ser úteis na detecção de populações que apresentem diferentes magnitudes de variabilidade genética e que, portanto, requerem diferentes estratégias para sua conservação in situ ou ex situ (Avise & Hamrick 1996, Newton et al. 1999). Quando, por outro lado, o interesse for a domesticação da espécie e sua utilização econômica, estes parâmetros podem auxiliar na definição de programas de coleta visando a seleção de apenas parte da variabilidade que seja de interesse para o melhorista (Borém 1998).
Finalmente, os dados moleculares podem ser utilizados para melhor compreender a dinâmica dos alelos nas populações de uma determinada espécie, fornecendo subsídios para o maior entendimento dos processos microevolutivos que estão atuando na diferenciação destas populações (Reis 1996, Avise 2000). Estas informações, aliadas aos conhecimentos da história de vida e de características ecológicas da espécie, permitem conhecer parte da sua biologia, bem como da sua interação com outras espécies do bioma no qual ela está inserida.
As espécies nativas dos cerrados merecem especial atenção, pois este bioma foi considerado recentemente como um dos "hotspots" mundiais de diversidade (Myers et al. 2000). Dentre as inúmeras frutíferas nativas que apresentam potencial de utilização em sistemas tradicionais de produção agrícola, o araticunzeiro (Annona crassiflora Mart.), da família Annonaceae, destaca-se pelo sabor de seus frutos, que são muito apreciados e, por isso, facilmente comercializáveis. Os frutos do araticunzeiro podem ser consumidos in natura, como, também, ser utilizados na fabricação de compotas, doces, geléias, sorvetes, sucos, licores e vinagres. Tratase de uma planta que apresenta valor ornamental e medicinal, sendo empregada popularmente como antidiarréica (sementes) e como antimicrobiana, por suas propriedades antifúngica e antibacteriana (Almeida 1998).
O presente trabalho teve como objetivo geral a caracterização genética de populações naturais de araticunzeiro, coletadas no Estado de Goiás, com base em marcadores isoenzimáticos. Para tanto, procedeuse a avaliação da estrutura, magnitude e distribuição da variabilidade genética existente entre e dentro das populações, bem como foi estimada a taxa de fecundação cruzada aparente.
Material e métodos
Foram consideradas neste estudo seis populações naturais de araticunzeiro, distantes entre si pelo menos 30km (tabela 1), com amostras constituídas de 30 indivíduos por população. De cada indivíduo, foram coletadas, ao acaso, duas folhas maduras, que foram acondicionadas em sacos plásticos, contendo pequena quantidade de água, e mantidas sob baixa temperatura até o processamento final no laboratório. Esse tamanho amostral pode ser considerado suficiente para a estimativa das freqüências alélicas dentro das populações, a partir de marcadores que apresentam locos com padrão de herança codominante (Araújo 1996, Hillis et al. 1996).
A extração e a análise isoenzimática foram realizadas no dia seguinte à coleta, em tampão de Namkoong, descrito em Alfenas et al. (1991). A separação por eletroforese foi realizada em gel de penetrose a 13,5% usando-se o sistema N(3-Aminopropil)-morfolina + Ácido cítrico (0,04M), definido por Clayton & Tretiak (Alfenas et al.1991). Foram analisados: 6-Fosfogluconato Desidrogenase (6PGD), Fosfoglucomutase (PGM), Malato Desidrogenase (MDH) e Leucina Aminopeptidase (LAP), segundo protocolo descrito em Lebrun & Chevalier (1988). Com base na interpretação dos zimogramas, os indivíduos foram genotipados para cada um dos locos enzimáticos encontrados.
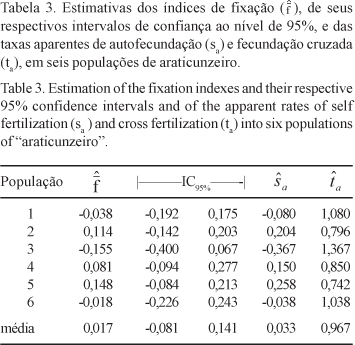
Os desvios das freqüências genotípicas obtidas em relação às freqüências esperadas pelas proporções de equilíbrio de Hardy-Weinberg foram estimados e testados utilizando-se o teste exato de Haldane (1954). Para cada população, foram estimados os valores do índice de fixação médio ( ) e das taxas aparentes de autofecundação (sa) e de fecundação cruzada (ta). Para as estimativas obtidas dos índices de fixação médios considerando cada população, foram estabelecidos intervalos de confiança a 95% de probabilidade utilizando-se o procedimento de "bootstrap" com 10.000 repetições (Weir 1996). A magnitude da variabilidade genética foi avaliada pela porcentagem de locos polimórficos, do número médio de alelos por loco e do índice de diversidade genética de Nei (1973).
) e das taxas aparentes de autofecundação (sa) e de fecundação cruzada (ta). Para as estimativas obtidas dos índices de fixação médios considerando cada população, foram estabelecidos intervalos de confiança a 95% de probabilidade utilizando-se o procedimento de "bootstrap" com 10.000 repetições (Weir 1996). A magnitude da variabilidade genética foi avaliada pela porcentagem de locos polimórficos, do número médio de alelos por loco e do índice de diversidade genética de Nei (1973).
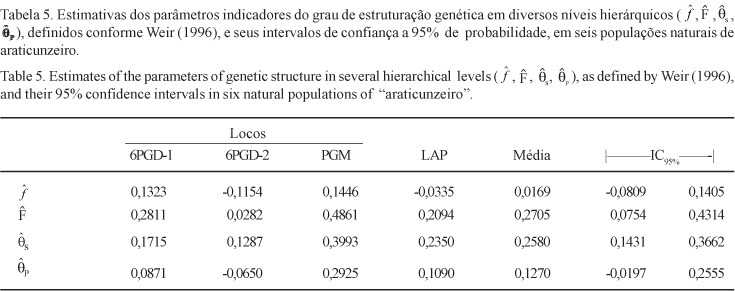
A estrutura genética das populações foi avaliada em termos da decomposição da diversidade genética total em seus componentes inter e intrapopulacionais, conforme preconizado por Nei (1973) e com base no procedimento de análise de variância de freqüências alélicas descrito em Weir (1996). Esta metodologia permitiu a obtenção de estimativas do coeficiente de endogamia intrapopulacional (f), equivalente ao coeficiente FIS de Wright; de endogamia total (F), equivalente ao coeficiente FIT de Wright, e dos coeficientes de coancestria para indivíduos dentro de populações (qs) e populações dentro de regiões (qp), equivalentes, respectivamente, aos coeficientes FST e FRT de Wright,. Foram obtidos intervalos de confiança a 95% de probabilidade, para as estimativas desses coeficientes de endogamia através do procedimento de "bootstrap" sobre locos, com 10.000 repetições.
Como medida de distância genética entre os pares de populações, foram utilizadas a distância genética de Reynolds et al. (1983) baseada no coeficiente de coancestria qs (Weir 1996) e a distância genética de Nei (1978). A partir das estimativas de distâncias genéticas foram, então, construídos dendrogramas, utilizando-se o método UPGMA ("Unweighted Pair Group Method Arithmetic Average"- Agrupamento não-ponderado aos pares utilizando médias aritméticas), conforme metodologia descrita em Sneath e Sokal (1973). A representatividade dos agrupamentos foi avaliada pelo coeficiente de correlação cofenética.
As matrizes de distância também foram representadas graficamente pelo método de ordenação denominado Escalonamento Multidimensional Não-Métrico (NMDS), proposto por Kruskal (1964a, b). Esta técnica parte de uma configuração inicial de pontos (populações) alocados ao acaso em um número reduzido de dimensões, normalmente 2-D ou 3-D. Com base nessa distribuição inicial são calculadas novas distâncias, as quais são comparadas às originais e, através de um procedimento iterativo, as diferenças entre essas matrizes são minimizadas utilizando uma estatística denominada stress (S). Quanto mais próximo de zero for o valor de S, menor a distorção e, portanto, melhor a representação das distâncias.
As matrizes de distâncias genéticas foram, em seguida, comparadas com a matriz de distância geográfica através de testes de Mantel (Manly 1997). O valor Z de Mantel é dado por:
onde Xij e Yij são elementos das matrizes X e Ya serem comparadas (no caso, as matrizes de distância geográfica e genética, respectivamente). A significância desse valor de Z pode ser obtida comparando-se o valor observado com valores de uma distribuição nula, construída recalculandose os valores de Z diversas vezes, aleatorizando, em cada uma delas, a ordem dos elementos de uma das matrizes. A estatística Z possui uma relação monotônica com o r de Pearson entre as matrizes (correlação matricial), de modo que ela é, de fato, utilizada para testar a significância do r (Manly 1997). Neste trabalho, 500 permutações aleatórias foram utilizadas para testar a significância das correlações matriciais.
Para o cálculo das estatísticas descritivas e para a análise de variância de freqüências alélicas foram utilizados os pacotes GDA (Genetic Data Analysis II) (Weir 1996) e TFPGA 1.3 (Tools For Population Genetic Analyses) (Miller 1997). As análises de divergência genética multivariadas (UPGMA e escalonamento multidimensional não-métrico) foram realizadas utilizando o pacote NTSYS 1.5 (Numerical Taxonomy and Multivariate Analysis System) (Rohlf 1989).
Resultados e Discussão
Foram identificados cinco locos isoenzimáticos: um loco para cada uma das enzimas PGM, MDH e LAP, e dois locos para a enzima 6PGD (denominados 6PGD-1 e 6PGD-2). O loco para a enzima MDH demonstrou ser monomórfico, enquanto os demais apresentaram dois alelos, com freqüências variáveis. A porcentagem de locos polimórficos foi estimada em 80%, devendo-se, no entanto, considerar esse valor com reserva, dado o pequeno número de locos amostrados. Entre as enzimas polimórficas, todas apresentaram um mecanismo de expressão compatível com aquele de enzimas monoméricas, tendo sido identificados os genótipos com base nesta hipótese.
Os resultados da análise de aderência das freqüências genotípicas observadas em relação às proporções esperadas segundo as condições de equilíbrio de Hardy-Weinberg foram, de maneira geral, concordantes em indicar que as populações de araticunzeiro se comportam como populações panmíticas (tabela 2), com taxas aparentes de fecundação cruzada próximas de 1,0 (tabela 3).
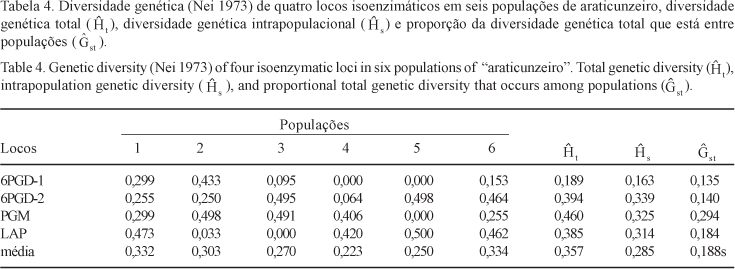
A análise da estrutura genética das populações (tabela 4) revelou, em termos dos locos polimórficos, a existência de elevada variabilidade genética total para a espécie na região. A diversidade genética (heterozigosidade) variou entre 0,189 e 0,460, com valor médio igual a 0,357, o que mostra a existência de heterogeneidade nas freqüências alélicas dos diferentes locos. Pode-se dizer ainda que, em média, 18,8% da variabilidade total foram devidos a diferenças interpopulacionais, denotando um elevado nível de diferenciação geográfica entre as populações, quando comparadas às de outros estudos com populações naturais de plantas (Hamrick & Loveless 1989, Dias & Kageyama 1991, Carthew 1993, Hamrick & Nason 1996). Estes resultados foram confirmados pela análise de variância de freqüências alélicas, que forneceu um valor significativo para a estimativa do coeficiente de parentesco de indivíduos dentro de populações (tabela 5), denotando uma maior similaridade genética entre indivíduos de uma mesma população se comparada àquela entre indivíduos tomados ao acaso do grupo formado por todas as populações. O nível de parentesco médio encontrado entre indivíduos de uma mesma população (qs) foi igual a 0,2580 (tabela 5), equivalente ao parentesco entre indivíduos de famílias de irmãos-germanos. Isto sugere que as populações provavelmente originaram-se de muito poucos indivíduos aparentados. Cumpre salientar que a dispersão dos frutos de araticum, que contêm muitas sementes, se dá por animais. Assim, a hipótese de que frutos únicos, contendo múltiplos indivíduos aparentados, possam dar origem a diferentes populações deve ser averiguada.
Embora a estimativa para o coeficiente de endogamia total (F) tenha sido significativa, não se encontrou uma estruturação significativa em nível intrapopulacional ou de regiões. Assim, pode-se afirmar que a endogamia existente se deve a um efeito que ocorre em nível populacional, provavelmente decorrente do pequeno tamanho dessas unidades (deriva genética).
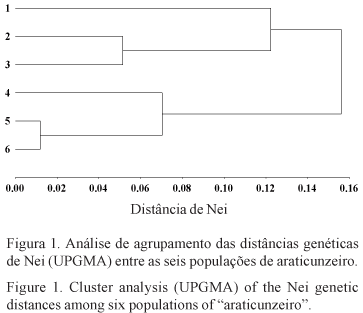
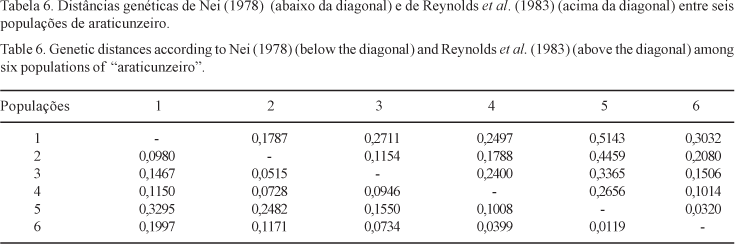
As matrizes de distâncias genéticas de Nei (1978) e de Reynolds et al. (1983) entre os pares de populações locais (tabela 6) revelaram os mesmos padrões de similaridade genética entre as populações locais, apresentando uma correlação de 0,9643. Assim, as análises de divergência foram baseadas nas distâncias genéticas de Nei. Essa matriz foi utilizada como base para a construção de um dendrograma através do método UPGMA, que sugere a existência de dois grupos de populações locais, formando um padrão hierárquico de semelhança genética da forma [(4,5,6)],[(2,3),1)] (figura 1). A correlação cofenética deste agrupamento, no entanto foi baixa (rC = 0,5818). A análise de ordenação por NMDS elaborada para representar, graficamente, a posição relativa das populações no espaço genético de duas dimensões está apresentada na figura 2. O valor de stress (S) do NMDS em 2-D foi igual a 0,0366, indicando que a representação das distâncias genéticas está acurada. O padrão encontrado de divergência entre as populações é semelhante ao do UPGMA, estando possivelmente associado à distribuição espacial dessas populações na região.
Os testes de Mantel mostraram que existe uma relação entre as distâncias geográficas e as distâncias genéticas estimadas, tanto para as distâncias de Nei (1978) (r = 0,4206; p = 0,070), quanto para as de Reynolds et al. (1983) (r = 0,4617; p = 0,074). No entanto, o número de populações é reduzido e, portanto, é interessante que se realizem coletas em mais localidades a fim de proceder a investigações mais refinadas de análise espacial da variabilidade genética. Essas informações podem ser úteis na elaboração de um programa efetivo de coleta, conservação e exploração do germoplasma.
As análises sugerem que a reprodução ocorre, preferencialmente, por fecundação cruzada, o que está de acordo com os dados sobre a biologia floral dessa espécie apresentados por Gottsberger (1994). Conforme relata esse autor, as plantas da família Annonaceae têm flores que apresentam dicogamia (protoginia), sendo as fases feminina (período de receptividade dos estigmas) e masculina (período de liberação do pólen) nitidamente distintas. Outra característica marcante das flores de Annonaceae dos Cerrados é o fechamento das pétalas sobre os órgãos reprodutivos durante a antese. A cavidade assim formada tem funções na polinização das flores, sendo chamada "câmara de polinização" ou "câmara floral". A polinização é feita exclusivamente por coleópteros.
Observou-se um valor significativo para o coeficiente de endogamia total, decorrente de uma estruturação genética em nível populacional, sendo as populações constituídas por indivíduos com grau de coancestria acima da média. Como conseqüência, uma significativa parcela da variabilidade genética total encontra-se entre as populações locais, estando as diferenças interpopulacionais associadas, possivelmente, à posição geográfica das populações. Deste modo, seguindo raciocínio análogo ao desenvolvido por Araújo (1996), se o interesse do pesquisador é preservar o máximo de variabilidade genética, deve-se, durante a coleta, priorizar o maior número possível de populações e não o maior número de indivíduos dentro de poucas populações, já que há uma diferenciação significativa entre as populações.
No estabelecimento de programas de melhoramento genético e de conservação deve-se levar em consideração também informações sobre a divergência adaptativa presente em outros caracteres fenotípicos, especialmente aqueles de interesse comercial, uma vez que o padrão de evolução desses caracteres ocorre de maneira diferente daqueles caracteres neutros, como pressuposto no caso das isoenzimas (Lynch 1996, Crandall et al. 2000).
Agradecimentos - Este trabalho foi desenvolvido com auxílio financeiro da Funape (Fundação de Apoio à Pesquisa da Universidade Federal de Goiás) e da Finep (Financiadora e Estudos e Projetos/MCT). Ao Dr. José Alexandre Felizola Diniz Filho pelo auxílio e concessão do pacote NTSYS.
(recebido: 26 de dezembro de 2001; aceito: 8 de janeiro de 2003)
- ALFENAS, A.C., DUSI, A., ZERBINI JÚNIOR, F.M., ROBINSON, I.P., MICALES, J.A., OLIVEIRA, J.R., DIAS, L.A.S., SCORTICHINI, M., BONDE, M.R., ALONSO, S.K., JJUNGHANS, T.G. & BRUNE, W.1998. Eletroforese de isoenzimas e proteínas afins - fundamentos e aplicações em plantas e microrganismos. Universidade Federal de Viçosa, Viçosa.
- ALFENAS, A.C., PETERS, I., BRUNE,W. & PASSADOR, G.C.1991. Eletroforese de proteínas e isoenzimas de fungos e essências florestais. Universidade Federal de Viçosa, Viçosa.
- ALMEIDA, S.P. 1998.Frutas nativas do cerrado: caracterização físico-química e fonte potencial de nutrientes. In Cerrado: ambiente e flora. Embrapa-CPAC, Planaltina, p.247-281.
- ARAÚJO, A.M. 1996. Tamanho populacional e tamanho amostral: a minimização dos riscos. Brazilian Journal of Genetics 19:1-5.
- AVISE, J.C. 2000. Phylogeography. Harvard University Press, Cambridge.
- AVISE, J.C. & HAMRICK, j.L. 1996. Conservation genetics, case histories from nature. Chapman & Hall, New York.
- BORÉM, A. 1998. Melhoramento de Plantas. 2a ed. Universidade Federal de Viçosa, Viçosa.
- CARTHEW, S.M.1993.Population genetic structure of Banksia spinulosa Heredity 70:566-573.
- CRANDALL, K.A., BININDA-EMONDS, O.R.P., MACE, G.M. & WAYNE, R.K. 2000. Considering evolutionary processes in conservation biology. Trends in Ecology and Evolution 15:290-295.
- DIAS, L.A.S. & KAGEYAMA, P.Y. 1991. Variação genética em espécies arbóreas e conseqüências para o melhoramento florestal. Agrotrópica 3:119-127.
- FERREIRA, M.E. & GRATTAPAGLIA, D. 1998. Introdução ao uso de marcadores moleculares em análise genética. Embrapa-Cenargen, Brasília.
- GOTTSBERGER, G. 1994. Annonáceas do cerrado e sua polinização. Revista Brasileira de Biologia 54:391-402.
- GRAY, A.1996. Genetic diversity and its conservation in natural populations of plants. Biodiversity Letters 3:71-80.
- HALDANE, J.B.S. 1954. An exact test for randomness of mating. Journal of Genetics 52:631-635.
- HAMRICK, J.L. & LOVELESS, M.D. 1989.The genetic structure of tropical tree populations: associations with reproductive biology. In The Evolutionary Ecology of Plants (J.H. Bock & Y.B. Linhart, eds.). Westview Press, Boulder, p.129-146.
- HAMRICK, J.L. & NASON, J.D.1996. Consequences of dispersal in plants. In Population dynamics in ecological space and time (O.E. Rhodes Jr., R.K. Chesser & M.H. Smith, eds.). The University of Chicago Press, Chicago, p. 203-236.
- HILLIS D.M., MORITZ, C. & MABLE, B.K. 1996. Molecular Systematics. Sinauer Associates, Sunderland.
- KRUSKAL, J.B. 1964a. Multidimensional scaling by optimizing goodness of fit to a nonmetric hypothesis. Psychometrika 29:1-27.
- KRUSKAL, J.B. 1964b. Non-metric multidimensional scaling: a numerical method. Psychometrika 29:28-42.
- LEBRUN, P. & CHEVALIER, M.H. 1988. Starch and polyacrylamid gel eletrophoresis of Hevea brasiliensis: a laboratory manual. IRCA-CIRAD Publishers, Montpellier.
- LYNCH, M. 1996. A quantitative-genetic perspective on conservation issues. In Conservation genetics, case histories from nature (J.C. Avise & J.L. Hamrick, eds.). Chapman & Hall, New York, p.471-501.
- MANLY, B.F.J. 1997. Randomization, bootstrap and Monte Carlo methods in Biology. Chapman & Hall, London.
- MILLER, M. 1997. Tools For Populations Genetic Analyses (TFPGA) 1.3: A windows program for analyses of allozyme and molecular population genetic data. Distribuído pelo autor.
- MYERS, N., MITTERMEIER, R.A., MITTERMEIER, C.G., FONSECA, G.A.B. & KENT, J. 2000. Biodiversity hotspots for conservation priorities. Nature 403:853-858.
- NEI, M. 1973. Analysis of genetic diversity in subdivided populations. Proceedings of National Academy of Sciences 70:3321-3323.
- NEI, M. 1978. Estimation of average heterozygosity and genetic distance from a small number of individuals. Genetics 89:583-590.
- NEWTON, A.C., ALLNUTT, T.R., GILLIES, A.C.M., LOWE, A.J. & ENNOS, R.A. 1999. Molecular phylogeography, intraspecific variation and the conservation of tree species. Trends in Ecology and Evolution 14:140-145.
- REIS, M.S.1996. Dinâmica da movimentação dos alelos: subsídios para conservação e manejo de populações naturais de plantas. Brazilian Journal of Genetics 19:37-47.
- REYNOLDS, J., WEIR, B.S., COCKERHAM, C.C. 1983. Estimation of the coancestry coefficient: Basis for a short-term genetic distance. Genetics 105:767-779.
- RITLAND, K.1990. A series of FORTRAN computer programs for estimating plant mating systems. Journal of Heredity 81:235-237.
- ROHLF, F.J. 1989. NTSYS-Pc:Numerical Taxonomy and Multivariate Analysis System. Exeter Publishers, New York.
- SNEATH, P.H. & SOKAL, R.R. 1973. Numerical Taxonomy: the principles and practice of numerical classification. W.H. Freeman, San Francisco.
- SUNNUCKS, P. 2000. Efficient genetic markers for population biology. Trends in Ecology and Evolution 15:199-1203.
- WEIR, B.S. 1996. Genetic Data Analysis II - Methods for Discrete Population Genetic Data. Sinauer Associates, Sunderland.
Datas de Publicação
-
Publicação nesta coleção
17 Nov 2003 -
Data do Fascículo
Mar 2003
Histórico
-
Recebido
26 Dez 2001 -
Aceito
08 Jan 2003