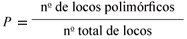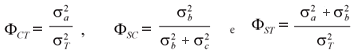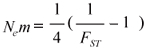Resumos
O presente trabalho teve como objetivo utilizar marcadores RAPD para conhecer a variabilidade genética de populações de Tibouchina papyrus (Pohl) Toledo, provenientes da região de Serra Dourada e Serra dos Pirineus, no Estado de Goiás. Os seis iniciadores RAPD produziram um total de 147 locos, variando entre 23 e 26 por iniciador. A avaliação hierárquica da estruturação da variabilidade genética, realizada pela a AMOVA, considerando a existência de duas regiões (Serra dos Pirineus e Serra Dourada) apresentou uma estimativa de F ST = 0,3439. O valor do componente entre regiões (F CT) foi igual a 18,96% e a variação entre populações dentro de regiões igual a 15,43%. As estimativas de fluxo gênico sugerem a existência de uma baixa proporção de migrantes entre populações. As análises multivariadas (UPGMA e NMDS) indicam que existe uma relação entre distância genética e espaço geográfico, hipótese esta que foi confirmada por uma análise de padrão espacial utilizando o teste de Mantel (r = 0,71; P = 0,015 com 1000 permutações aleatórias). Os resultados indicam assim que esta estrutura tenha se originado seguindo um modelo de diferenciação estocástica (neutro), ou seja, por um balanço entre fluxo gênico a curtas distâncias e deriva genética nas populações. Os valores de diversidade genética obtidos apóiam a hipótese de que a espécie T. papyrus é uma espécie xenógama facultativa e o fato de não serem encontrados altos níveis de homozigose, indica que devem existir mecanismos relacionados à biologia reprodutiva da espécie, que previnem, de alguma maneira, a ocorrência de elevadas taxas de endogamia, que poderia ter um efeito deletério em médio e longo prazo.
cerrado; RAPD; Tibouchina papyrus; variabilidade genética
This paper has the aim of using RAPD markers to describe the genetic of variability of Tibouchina papyrus (Pohl) Toledo local populations, from the regions of Serra Dourada and Serra de Pirineus, in Goiás State, Central Brazil. The six RAPD primers generated a total of 147 loci, varying from 23 to 26 per primer. The hierarchical evaluation of genetic variability, performed using an Analysis of Molecular Variance (AMOVA) considering the local populations within the two regions (i.e., Serra Dourada and Serra de Pirineus), showed an estimate of overall diversity among populations equal to F ST = 0.3439. The value of divergence between regions (F CT) was equal to 18.96% so that the variation among local populations within regions was equal to 15.43%. Estimates of gene flow suggest a small number of migrants among local populations per generation. Multivariate analyses (UPGMA and NMDS) indicated that a relationship between genetic and geographical distances exists, which was confirmed by a spatial pattern analysis using Mantel test (r = 0.71; P < 0.015 with 1000 random permutations). Thus, this structure was originated from a stochastic neutral model of population differentiation, in which drift within populations is counteracted by short distance gene flow. Despite strong levels of population structure (i.e., high divergence among populations within regions), the diversity observed support the hypothesis that T. papyrus is a facultative xenogamous species. The lack of high levels of homozigosity indicates that ecological mechanisms related to species' reproductive biology prevent, someway, a high level of endogamy within local structured populations, which could cause deleterious effects in a long run.
cerrado; genetic variability; RAPD; Tibouchina papyrus
ARTIGO DE REVISÃO
Estrutura genética em populações naturais de Tibouchina papyrus (pau-papel) em áreas de campo rupestre no cerrado
Genetic structure of natural populations of Tibouchina papyrus (pau-papel) in areas of cerrado rupestrian fields
Mariana Pires de Campos TellesI,1 1 . Autor para correspondência: mpctelles@pq.cnpq.br ; Sandra Pereira da SilvaI; Juliana Rosa RamosI; Thannya Nascimento SoaresI; Dayane Borges MeloI; Lucileide Vilela ResendeI; Eliane cotrim BatistaI; Breno de Faria VasconcellosII
IUniversidade Federal de Goiás, Departamento de Biologia Geral, Laboratório de Genética & Biodiversidade, Caixa Postal 131, 74605-010 Goiânia, GO, Brasil
IIUniversidade Católica de Goiás, Departamento de Zootecnia, Caixa Postal 86, 74605-010 Goiânia, GO, Brasil
ABSTRACT
This paper has the aim of using RAPD markers to describe the genetic of variability of Tibouchina papyrus (Pohl) Toledo local populations, from the regions of Serra Dourada and Serra de Pirineus, in Goiás State, Central Brazil. The six RAPD primers generated a total of 147 loci, varying from 23 to 26 per primer. The hierarchical evaluation of genetic variability, performed using an Analysis of Molecular Variance (AMOVA) considering the local populations within the two regions (i.e., Serra Dourada and Serra de Pirineus), showed an estimate of overall diversity among populations equal to FST = 0.3439. The value of divergence between regions (FCT) was equal to 18.96% so that the variation among local populations within regions was equal to 15.43%. Estimates of gene flow suggest a small number of migrants among local populations per generation. Multivariate analyses (UPGMA and NMDS) indicated that a relationship between genetic and geographical distances exists, which was confirmed by a spatial pattern analysis using Mantel test (r = 0.71; P < 0.015 with 1000 random permutations). Thus, this structure was originated from a stochastic neutral model of population differentiation, in which drift within populations is counteracted by short distance gene flow. Despite strong levels of population structure (i.e., high divergence among populations within regions), the diversity observed support the hypothesis that T. papyrus is a facultative xenogamous species. The lack of high levels of homozigosity indicates that ecological mechanisms related to species' reproductive biology prevent, someway, a high level of endogamy within local structured populations, which could cause deleterious effects in a long run.
Key words:cerrado, genetic variability, RAPD, Tibouchina papyrus
RESUMO
O presente trabalho teve como objetivo utilizar marcadores RAPD para conhecer a variabilidade genética de populações de Tibouchina papyrus (Pohl) Toledo, provenientes da região de Serra Dourada e Serra dos Pirineus, no Estado de Goiás. Os seis iniciadores RAPD produziram um total de 147 locos, variando entre 23 e 26 por iniciador. A avaliação hierárquica da estruturação da variabilidade genética, realizada pela a AMOVA, considerando a existência de duas regiões (Serra dos Pirineus e Serra Dourada) apresentou uma estimativa de FST = 0,3439. O valor do componente entre regiões (FCT) foi igual a 18,96% e a variação entre populações dentro de regiões igual a 15,43%. As estimativas de fluxo gênico sugerem a existência de uma baixa proporção de migrantes entre populações. As análises multivariadas (UPGMA e NMDS) indicam que existe uma relação entre distância genética e espaço geográfico, hipótese esta que foi confirmada por uma análise de padrão espacial utilizando o teste de Mantel (r = 0,71; P = 0,015 com 1000 permutações aleatórias). Os resultados indicam assim que esta estrutura tenha se originado seguindo um modelo de diferenciação estocástica (neutro), ou seja, por um balanço entre fluxo gênico a curtas distâncias e deriva genética nas populações. Os valores de diversidade genética obtidos apóiam a hipótese de que a espécie T. papyrus é uma espécie xenógama facultativa e o fato de não serem encontrados altos níveis de homozigose, indica que devem existir mecanismos relacionados à biologia reprodutiva da espécie, que previnem, de alguma maneira, a ocorrência de elevadas taxas de endogamia, que poderia ter um efeito deletério em médio e longo prazo.
Palavras-chave: cerrado, RAPD, Tibouchina papyrus, variabilidade genética
Introdução
As transformações ocorridas nos últimos anos no cerrado trouxeram grandes danos ambientais fragmentação de habitats, extinção da biodiversidade, invasão de espécies exóticas, erosão dos solos, poluição de aquíferos, degradação de ecossistemas, alterações nos regimes de queimadas, desequilíbrios no ciclo do carbono e possivelmente modificações climáticas regionais (Klink & Machado 2005). Muitas espécies da flora do cerrado estão sendo ameaçadas de extinção, principalmente as de interesse madeireiro e medicinal (Vieira & Martins 2000), outras, porém, são endêmicas e apresentam áreas de distribuição restrita a determinadas regiões no cerrado, e, portanto estão mais vulneráveis às modificações nos seus habitats. Devido ao grande endemismo de espécies e à atual velocidade de devastação, o cerrado é considerado, desde o ano 2000, um dos 25 hot spots de diversidade biológica do mundo, prioritários para a conservação (Myers et al. 2000).
Tibouchina papyrus (Pohl) Toledo é uma espécie arbustiva, pertencente à família Melastomataceae, endêmica do bioma Cerrado. Apresenta uma floração abundante com flores alvas, casca do tronco escamado em lâminas finíssimas e suas características peculiares fazem com que apresente potencial de utilização como ornamental. Sua área de ocorrência natural é restrita e se limita aos campos rupestres do cerrado, nas regiões da Serra dos Pireneus em Pirenópolis, GO, Serra Dourada em Goiás, GO e em Natividade, TO (Almeida et al. 1998, Montoro & Santos 2007). Alguns trabalhos sobre a fenologia e a reprodução de T. papyrus (Montoro & Santos 2007, J. T. Chaves Filho, dados não publicados), sugerem que a espécie é xenógama facultativa, o que explica a baixa formação de frutos a partir de autopolinização (12%), tendo uma maior formação de frutos por polinização cruzada (45%).
É sabido que a perda de variabilidade genética pode ser decorrente de alguns fatores ecológicos e microevolutivos tais como flutuações demográficas, endogamia, deriva genética, além de modificações e/ou flutuações nas características do ambiente (Primack & Rodrigues 2001). Estudos que se propõe avaliar a variabilidade genética de espécies endêmicas do Cerrado permitem a obtenção de informações que auxiliam a avaliação da probabilidade de persistência das populações locais dessas espécies, contribuindo em última análise para a conservação da espécie e do bioma no qual ela é originária.
A caracterização genética de populações utilizando marcadores moleculares é fundamentada na análise das diferenças nos perfis genéticos e, ocasionalmente, na identificação de alelos específicos em populações. Atualmente existe uma grande variedade de marcadores moleculares disponíveis na literatura, que apresentam considerável polimorfismo e, portanto, permitem analisar a variabilidade genética dentro e entre populações (Alfenas et al. 2006). O RAPD (Random Amplified Polymorfic DNA) é um marcador molecular que pode ser indicado para uma avaliação prévia da diversidade genética em populações de espécies que ainda são pouco conhecidas do ponto de vista científico, como é o caso do pau-papel, pois não são marcadores espécie-específico. A técnica RAPD é relativamente simples de ser executada no laboratório, rápida na obtenção dos dados, com custo relativamente baixo quando comprado a outras técnicas moleculares (Borém & Caixeta 2006). Outro ponto importante diz respeito à repetibilidade dos locos RAPD, que pode ser assegurada seguindo critérios rígidos de controle das reações de PCR e de escolha dos iniciadores e do número de locos necessários para a estimativa robusta de parâmetros genético populacionais (Telles et al. 2001, Ramos et al. 2008).
A conservação da diversidade genética é uma condição para a manutenção de todos os níveis de biodiversidade e é um componente essencial da sustentabilidade das populações (Namkoong et al. 2002). Nesta perspectiva, espécies endêmicas como a T. papyrus, merecem uma atenção especial mesmo estando com parte de suas populações dentro de áreas legalmente protegidas, como em parques ou reservas ecológicas. Neste contexto, o objetivo geral do trabalho foi utilizar marcadores RAPD para conhecer a variabilidade genética existente em populações de T. papyrus, provenientes das regiões de Serra Dourada e da Serra de Pirineus, no Estado de Goiás. Mais especificamente, procurou-se: 1) Avaliar a diversidade genética dentro das populações; 2) Estimar a magnitude e a distribuição da variabilidade genética entre e dentro das populações e entre regiões; 3) Avaliar, de maneira indireta, o fluxo gênico entre as populações; 4) Estimar a divergência genética entre as populações; 5) Verificar se a variabilidade genética está estruturada espacialmente. Considerando o endemismo e a distribuição disjunta das populações de T. papyrus, espera-se que a variabilidade genética esteja altamente estruturada entre as regiões, uma vez que essas populações estão isoladas e distantes geograficamente a muitas gerações, o que provavelmente elimina a possibilidade de ocorrência fluxo gênico atual.
Material e métodos
Área de coleta e obtenção do DNA Foram amostradas folhas de árvores localizadas em duas regiões de ocorrência natural de T. papyrus. A primeira na Serra dos Pirineus, localizada nos Municípios de Pirenópolis, GO e a segunda, na região da Serra Dourada, localizada no Município de Goiás, GO (tabela 1). As folhas foram coletadas e acondicionadas em sacos de papel (identificados) e transportadas em caixa de isopor com gelo, para melhor conservação. No momento da coleta todas as áreas foram georeferenciadas para as análises espaciais.
O DNA foi extraído das folhas de 327 indivíduos, distribuídos em seis subpopulações, sendo duas oriundas da Serra Dourada e quatro de Pirenopólis, denominadas SD1, SD2, P1, P2, P3 e P4, contendo respectivamente 71, 69, 67, 36, 42 e 42 indivíduos por subpopulação (tabela 1). A extração do DNA foi realizada conforme o protocolo descrito por Ferreira & Grattapaglia (1998). Após a extração, o DNA total de cada uma das plantas foi quantificado por eletroforese com o auxílio do marcador de peso molecular Low DNA Mass, fornecido pela Invitrogen. O gel, depois de corado, foi visualizado em um transiluminador de luz ultravioleta e fotografado para a estimativa da quantidade de DNA em cada amostra de DNA. Em seguida, a solução estoque de DNA foi diluída para uma concentração final de trabalho de aproximadamente 3 ng µL-1.
Amplificação e obtenção dos dados moleculares Para a amplificação dos locos foram utilizados seis iniciadores RAPD (OPB-4, OPB-5, OPB-10, OPM-3, OPM-5, OPM-13) que haviam sido previamente selecionados. A amplificação, via reação em cadeia da polimerase (PCR), para cada iniciador RAPD, nas seis populações, foi conduzida em um mesmo termociclador, vizando minimizar os possíveis riscos com relação à mudança nas condições de amplificação, que podem gerar problemas de repetibilidade (Ramos et al. 2008). O programa de amplificação utilizado apresenta a temperatura de anelamento do iniciador RAPD igual 37 ºC, que é um pouco mais alta do que o comumente utilizado, aumentando assim a especificidade do pareamento do iniciador à região alvo, o que consequentemente melhora o produto amplificado pela enzima Taq DNA polimerase.
As reações de PCR para um volume final de 20 µl foram montadas da seguinte maneira: 0,45 ng µL-1 de DNA (3 ng µL-1); 2,6X do tampão da enzima (10X); 1,95 mol mL-1 de MgCl2 (50 mM); 0,26 mol mL-1 de d'NTP (2,5 mM); 0,05 U da enzima Taq-Polimerase (Amersham Pharmacia Biotech™), completando o volume com 9,34 µL de H2O Milli-Q.
Para a amplificação dos fragmentos de DNA foi utilizado o programa de termociclagem descrito a seguir: (1º) desnaturação do DNA a 96 ºC por 3 minutos e (2º) a 92 ºC por 1 minuto; (3º) anelamento do iniciador RAPD a 37 ºC por 1 minuto; (4º) extensão a 72 ºC por 1 minuto; (5º) 40 ciclos seguindo do 2º ao 4º passo; (6º) extensão de 3 minutos a 72 ºC para finalizar os produtos amplificados. Os fragmentos amplificados foram analisados por eletroforese em gel de agarose 1,4% com TBE 1X.
A codificação dos géis de RAPD foi realizada segundo a metodologia usual, atribuindo presença (1) ou ausência (0) do fragmento (banda), para cada indivíduo. O marcador de peso molecular utiliza-se após o de 100 bp (Invitrogen). Foram codificados somente os fragmentos mais nítidos no gel, para facilitar a distinção entre eles e minimizar erros de obtenção dos genótipos entre os géis. Além disso, foi respeitado um intervalo de tamanho dos fragmentos no gel, que não ultrapassou os limites, em pares de bases, entre 2600 (superior) e 200 (inferior). Desta codificação foi obtida uma matriz de dados binários que foi utilizada para as análises genético populacionais.
Variabilidade, estrutura genética e fluxo gênico A variabilidade genética dos locos RAPD foi estimada, a partir da matriz de dados binários resultante da análise dos géis conforme descrito em Weir (1996), Freeland (2005) e Alfenas et al. (2006). Inicialmente, a variabilidade genética foi avaliada, para cada iniciador RAPD, por meio da estimativa do polimorfismo presente nos locos, considerando-se todos os indivíduos simultaneamente e, em seguida, cada população separadamente. Portanto, foi estimado o número e proporção de locos (ou bandas) polimórficos:
A diversidade genética (h), para cada população, foi avaliada conforme descrito por Nei (1973), sendo calculada seguinte maneira:
em que,  é a frequência do alelo i e m é o número de alelos encontrado em cada loco (Freeland 2005).
é a frequência do alelo i e m é o número de alelos encontrado em cada loco (Freeland 2005).
A avaliação da proporção da variabilidade genética que está entre e dentro das populações foi obtida pelo método mais comumente utilizado para dados de marcadores "dominantes", a Análise de Variância Molecular (AMOVA) proposta por Excoffier et al. (1992). Esse procedimento foi realizado no programa Arlequin versão 2.000 (Schneider et al. 2007). Essa metodologia permite desdobrar a distância entre e dentro de populações locais ou regiões, utilizando uma estratégia semelhante à realizada na ANOVA convencional, mas parte de uma matriz de distância euclidiana calculada entre indivíduos com base na presença (1) e ausência (0) das bandas RAPD. O modelo matemático que descreve a AMOVA é um modelo hierárquico que considera os níveis de regiões, de populações e de indivíduos dentro de população, conforme a seguinte equação:
em que x é a média geral, a é o efeito de regiões, b de populações e c de haplótipos dentro de população, dentro de regiões, sendo que esses efeitos são aditivos, aleatórios e independentes, associados aos componentes de variância,  ,
,  ,
,  e, respectivamente, e a variância total
e, respectivamente, e a variância total  é obtida pela soma destes.
é obtida pela soma destes.
O componente de variância inter-regional e inter-populacional pode ser extraído por equações das esperanças de quadrado médio (QMD), análogo à análise de variância convencional das frequências alélicas, utilizadas para estimar o ΦST (Cockerham 1969, 1973). O mesmo procedimento pode ser empregado com base no desdobramento da soma de quadrado entre as distâncias, utilizando as chamadas estatísticas-F. Como na ANOVA convencional, pode-se reescrever essas equações em termos das estatísticas F entre as regiões (ΦCT), entre as populações locais dentro de regiões (ΦSC) e entre todas as populações locais (ΦST), de modo que:
A significância associada a cada uma destas estimativas foi obtida utilizando-se 5.000 permutações da seguinte maneira: para  e ΦST permutando-se indivíduos entre populações entre regiões; para
e ΦST permutando-se indivíduos entre populações entre regiões; para  e ΦSC permutando-se indivíduos entre populações dentro regiões; para
e ΦSC permutando-se indivíduos entre populações dentro regiões; para  e ΦCT permutando-se populações entre regiões.
e ΦCT permutando-se populações entre regiões.
Para obter uma distribuição nula dessas estatísticas, foram utilizados procedimentos de aleatorização, por permutações aleatórias das fileiras (e colunas correspondentes) da matriz de distâncias quadráticas (Mantel 1967). Os componentes de variância foram estimados para cada uma das matrizes permutadas (cerca de 5.000 permutações).
Com o intuito de obter uma estimativa indireta do fluxo gênico entre essas populações, foi utilizada a abordagem proposta em Freeland (2005), que se baseia na teoria das estatísticas-F proposta por Wright (1951). Esta proposta parte da premissa de que existe uma relação simples entre divergência genética, medida pelo ΦST (ou análogos), e a intensidade de fluxo gênico entre essas populações é dada por:
em que, Ne é o tamanho efetivo de cada população e m é a taxa de migração entre as populações. Portanto, Nem é a proporção de adultos reprodutivos migrantes. A partir desta equação anterior, pode-se, portanto, calcular o valor de Nem, como sendo:
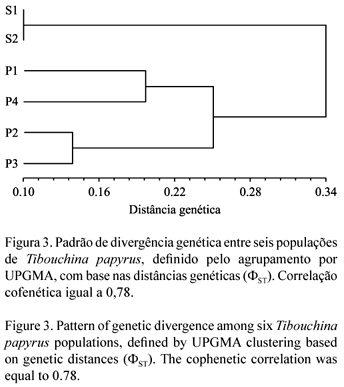
Divergência genética e padrão espacial A divergência genética entre as populações foi avaliada com base na matriz de distância genética obtida pela AMOVA (ΦST par a par), que é a que apresenta um menor número de pressupostos em relação ao equilíbrio de Hardy-Weinberg, tamanho amostral e padrão de herança do marcador, pois foi desenvolvida para este tipo de dado. Após a estimativa da divergência genética, vários métodos multivariados podem ser aplicados. Neste estudo, a matriz de distâncias genéticas foi analisada, inicialmente, a partir de uma análise de agrupamento tipo UPGMA (Unweighted Pair-group Method by Arithmetic Averages). A representatividade deste dendrograma foi testada por meio da correlação entre as distâncias genéticas originais e as distâncias entre as populações no dendrograma (correlação cofenética) (Legendre & Legendre 1998).
Apesar de o agrupamento pelo método do UPGMA ser uma das técnicas mais utilizadas neste tipo de estudo, ele pode não representar adequadamente as distâncias genéticas, indicando um falso arranjo hierárquico entre as populações, quando existe entre elas, de fato, um padrão contínuo ou reticulado. Em função desses problemas, as distâncias genéticas também foram analisadas por uma técnica de ordenação que visa representar graficamente a dissimilaridade entre as populações, o escalonamento multidimensional não-métrico (non-metric multidimensional scaling) (Lessa 1990). Esta técnica parte de uma configuração inicial de pontos (populações) alocados ao acaso em um número reduzido de dimensões, normalmente 2-D ou 3-D. Com base na distribuição ao acaso das populações são calculadas novas distâncias e estas são comparadas com as distâncias genéticas originais, através de um procedimento iterativo, com o objetivo de minimizar as diferenças entre as matrizes. Essa minimização é mensurada, ao longo do processo iterativo, por uma estatística denominada estresse (S). Quanto mais próximo de zero for o valor de S, menor a distorção e, portanto, melhor a representatividade das distâncias genéticas (Manly 1997).
Com intuito de analisar os padrões de variação espacial, em um contexto multivariado, foi realizado o teste de Mantel. Para tanto, foi feita a estimativa do coeficiente de correlação de Pearson (r) entre as matrizes de distâncias genéticas e as distâncias geográficas entre as populações (Epperson 2003). Para se testar a significância das correlações matriciais foram utilizadas 1.000 permutações aleatórias (Manly 1997).
Resultados e discussão
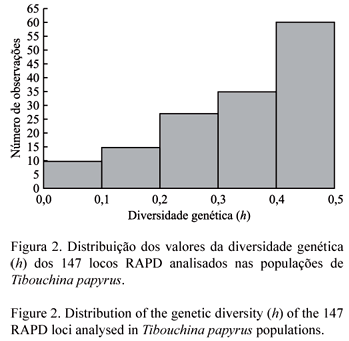
Variabilidade genética, estrutura genética populacional e fluxo gênico Dos 332 indivíduos coletados, somente 327 foram utilizados nas análises, por apresentarem boa quantidade e qualidade de DNA. A matriz de dados binários proveniente da codificação dos seis iniciadores RAPD gerou um total de 147 locos, variando entre 23 e 26 por iniciador (tabela 2). Na figura 1 observa-se o perfil de amplificação de algumas amostras de DNA dos indivíduos das populações de T. papyrus, utilizando-se o iniciador OPB-05.
As seis populações de T. papyrus apresentaram uma variabilidade genética consideravelmente alta, que foi observada para os seis iniciadores RAPD, nos 327 indivíduos analisados. O número de locos polimórficos, por iniciador RAPD, variou entre 121 (P2) e 143 (P1), com um total igual a 147 locos, considerando o total de indivíduos avaliados. Quando todos os locos foram considerados, a porcentagem de locos polimórficos foi igual a 100% e, nas populações, variou entre 82% (P2) e 97% (P1). A diversidade genética (h) ou heterozigose esperada (Nei, 1973), quando avaliada para os 147 locos e em todos os 327 indivíduos, apresentou uma distribuição de frequência que variou entre 0 e 0,5 (figura 2), sugerindo que existem locos que apresentam a máxima diversidade esperada para um loco com dois alelos (h = 0,499) e outros que já perderam variabilidade genética, praticamente fixando um dos alelos (h = 0,025). A diversidade genética geral, considerando todos os locos simultaneamente em todos os indivíduos, foi alta e igual a 0,34, variando nas populações entre 0,24 (P2 e P4) e 0,33 (P1) (tabela 3).
Dessa forma, pode-se verificar que apesar de ser uma espécie endêmica e com distribuição geográfica restrita, para os locos avaliados nesse trabalho, existe uma grande variabilidade genética, provavelmente em função das estratégias reprodutivas adotadas pela espécie ao longo de sua evolução. A atuação dos agentes polinizadores (abelhas do gênero Bombus) é de extrema importância para garantir este processo, uma vez que os mesmos são responsáveis por grande parte da fecundação cruzada que ocorre entre os indivíduos, o que possibilita o aparecimento de novas combinações genotípicas nos descendentes (Lovelless & Hamrick 1984).
Os valores de diversidade genética, encontrados neste trabalho, corroboram a hipótese de que a espécie T. papyrus é uma espécie xenógama facultativa, com maior formação de frutos por polinização cruzada do que por autopolinização, conforme descrito na literatura (Montoro & Santos 2007, J. T. Chaves Filho, dados não publicados). Além disso, o fato de não ser encontrado altos níveis de homozigose, sugere que existem mecanismos que estão evitando, de alguma maneira, a ocorrência de elevadas quantidades de endogamia, que poderia ter um efeito deletério em médio e longo prazo. Essa estratégia reprodutiva, provavelmente, tem garantido a persistência de suas populações com níveis consideráveis de variabilidade genética nas regiões onde a espécie T. papyrus é endêmica e restrita.
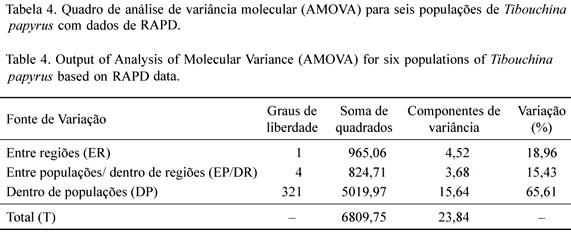
A avaliação da estruturação da variabilidade genética, realizada pela AMOVA (tabela 4), forneceu uma estimativa global de ΦST = 0,3439 (P < 0,00001, com 5.000 permutações), ou seja 34,39% da variação total se encontra entre as populações, sendo que destes, 18,96% referem-se à variação entre regiões (ΦCT) e 15,43% à variação entre populações dentro de regiões. Deste modo, para cada região, pode-se verificar que 84,57% da variância genética se encontram no componente intrapopulacional. Se for desconsiderado o efeito da região o componente da variância genética intrapopulacional passa a assumir um valor igual 65,6%.
Os valores elevados de estruturação da variabilidade genética entre regiões estão de acordo com o esperado, uma vez que as mesmas são geograficamente distantes o bastante para impedir a ocorrência de fluxo gênico atual entre as populações dessas regiões. Neste sentido, uma explicação possível para a similaridade genética compartilhada entre as populações dessas regiões é o efeito fundador compartilhado por populações ancestrais, que está refletindo, portanto, um fluxo gênico histórico, antes separação geológica das serras. O valor de estruturação de populações dentro de cada região, embora seja considerado moderadamente alto (15,43%), é muito semelhante aos encontrados na literatura para outras espécies de plantas do cerrado (Telles et al. 2001, Collevatti et al. 2001, Zucchi et al. 2005, Soares et al. 2008).
A estimativa da proporção de migrantes, por geração, calculada com base no ΦST (0,34), resultou num Nem igual a 0,48 indivíduos. No entanto, como foi verificado que há um elevado e significativo componente de variação genética entre regiões, a estimativa da proporção de migrantes por geração, torna-se melhor estimada a partir da proporção de variação entre populações dentro de regiões (ΦSC = 0,15), que passa a assumir um valor de Nem igual a 1,37 indivíduos. Com base nesses resultados, pode-se inferir que a estimativa de fluxo gênico, obtida a partir do ΦST, reflete com mais intensidade um componente histórico e o obtido com base no ΦSC, se aproxime mais às taxas atuais de fluxo gênico.
Divergência genética e padrão espacial Com base nas análises de variabilidade genética entre e dentro das populações descritas anteriormente pode-se observar que existe diferenciação significativa entre as populações. Neste sentido, pode-se observar que as distâncias genéticas (ΦST) entre as populações (tabela 5) variaram entre 0,095 (SD1 e SD2) e 0,453 (SD1 e P2).
Para visualizar o padrão de divergência entre as populações de T. papyrus, a análise de agrupamento por UPGMA da matriz de distância genética, embora tenha apresentado um valor da correlação cofenética não muito elevado (r = 0,78), permitiu verificar que as ligações no dendrograma refletem corretamente os padrões multivariados de distâncias genéticas entre as populações (figura 3). Esse resultado também confirma a elevada estruturação genética encontrada entre regiões, uma vez que o agrupamento permite a visualização de dois grandes grupos, um contendo as duas populações de Serra Dourada (SD1 e SD2), extremamente semelhantes geneticamente, e o outro as demais populações da Serra dos Pirineus (P1, P2, P3 e P4).
O padrão de divergência genética entre as populações de T. papyrus, também avaliado pelo escalonamento multidimensional não-métrico (NMDS), apresentou um valor de estresse em duas dimensões igual a 0,00007 (figura 4). As duas abordagens possibilitaram a mesma interpretação e sugerem que deve haver uma relação direta entre distância genética e geográfica, pois as populações de Serra Dourada (SD1 e SD2) são sempre mais similares geneticamente em relação às demais que são oriundas da Serra de Pirineus (P1, P2, P3 e P4). Outro ponto interessante de ser observado é que, nas duas análises, a P1 (da Serra de Pirineus) difere geneticamente das demais da mesma região (P2, P3 e P4) e, de fato, os indivíduos da P1 foram coletados na base da Serra, enquanto os demais foram coletados no topo da Serra (muito semelhante à configuração das populações obtida na área de coleta) (figura 4).
Ainda na investigação do padrão espacial da variabilidade genética presente nessas populações, o teste de Mantel apresentou uma correlação matricial entre distância genética (ΦST) e geográfica igual a 0,715 (P = 0,015, com 1.000 permutações) (figura 5). Esses resultados mostram que, de fato, a distância geográfica parece influenciar fortemente na estruturação espacial da variabilidade genética entre essas populações de T. papyrus, que sabidamente estão localizadas em duas regiões descontínuas e isoladas. Essa análise de padrão espacial sugere que esta estrutura tenha se originado seguindo um modelo de diferenciação estocástica (neutro), ou seja, por um balanço entre fluxo gênico a curtas distâncias e deriva genética nas populações. Esses resultados confirmam o que foi assinalado nas análises de estruturação da variabilidade genética reforçando a influência do isolamento geográfico entre as populações das duas regiões e provável interrupção do fluxo gênico atual.
Diversos estudos, disponíveis na literatura, que utilizaram marcadores RAPD em populações de espécies de plantas nativas do cerrado (tabela 6), apresentam valores médios de estruturação genética bem inferiores aos encontrados neste trabalho. No entanto, a maioria dessas espécies possui hábito arbóreo e ampla distribuição geográfica de suas populações nas áreas de cerrado, reforçando a hipótese de que espécies endêmicas e de distribuição restrita, como é o caso da T. papyrus, exibem um modelo de evolução das suas populações muito peculiar, que necessita melhor compreensão, antes que se proceda qualquer tipo de intervenção humana. Outro ponto importante é que as comparações dos valores de estruturação devem ser realizadas com cautela, pois os trabalhos são conduzidos de maneira muito diferentes, exibindo muita variação no número de populações, indivíduos dentro de populações e locos, além de escalas espaciais muito distintas.
Para fins de conservação de germoplasma, com base nestes resultados preliminares, pode-se sugerir a coleta de sementes do maior número possível de populações, e nas duas regiões avaliadas, pois cada uma contém um componente singular da variação genética total.
Agradecimentos Agradecemos ao Dr. José Alexandre Felizola Diniz Filho pelo apoio e sugestões durante o desenvolvimento deste trabalho. Ao CNPq pelas diversas formas de apoio (Pronex proc. 23234156; Universal proc. 471492/2007-8; Bolsa de Produtividade proc. 309404/2007-0) À Capes pelo oportunidade no Programa Nacional de Cooperação Acadêmica (Procad-NF proc. 1418/2007).
(recebido: 17 de abril de 2008; aceito: 25 de março de 2010)
- ALFENAS, A.C. 2006. Eletroforese e marcadores bioquímicos em plantas e microorganismos. Editora UFV, Viçosa.
- ALMEIDA, S.P., PROENÇA, C.E.B., SANO, S.M. & RIBEIRO, J.F. 1998. Cerrado: espécies vegetais úteis. Embrapa-CPAC, Planaltina.
- BERTONI, B.W., ASTOLF FILHO, S., MARTINS, E.R., DAMIAO FILHO, C.F., FRANCA, S.C., PEREIRA, A.M.S., TELLES, M.P.C. & DINIZ-FILHO, J.A.F. 2007. Genetic variability in natural populations of Zeyheria montana Mart. from Brazilian cerrado. Scientia Agricola 64:409-415.
- BORÉM, A. & CAIXETA, E.T. 2006. Marcadores moleculares. Editora UFV, Viçosa.
- COCKERHAM, C.C. 1969. Variance of gene frequencies. Evolution 23:72-84.
- COCKERHAM, C.C. 1973. Analyses of gene frequencies. Genetics 74:679-700.
- COLLEVATTI, R.G., GRATTAPAGLIA, D. & HAY, J.D. 2001. Population genetic structure of the endangered tropical tree species Caryocar brasiliense, based on variability at microsatellite loci. Molecular Ecology 10:349-356.
- EPPERSON, B.K. 2003. Geographical genetics. Princeton University Press, New Jersey.
- EXCOFFIER, L., SMOUSE, P., QUATTRO, J.M. 1992. Analysis of molecular variance inferred from metric distances among DNA haplotypes application to human mitochondrial DNA restriction data. Genetics 13:479-491.
- FERREIRA, M.E. & GRATTAPAGLIA, D. 1998. Introdução ao uso de marcadores moleculares em análise genética. Embrapa Cenargen, Brasília.
- FREELAND, J.R. 2005. Molecular ecology. John Wiley & Sons, Southern Gate.
- KLINK, C.A. & MACHADO, R.B. 2005. A conservação do Cerrado brasileiro. Megadiversidade 1:1-9.
- LACERDA, D.R., ACEDO, M.D.P., LEMOS FILHO, J.P. & LOVATO, M.B. 2001. Genetic diversity and struture of natural populations of Plathymenia reticulata (Mimosoideae), a tropical tree from the Brazilian Cerrado. Molecular Ecology 10:1143-1152.
- LEGENDRE, P. & LEGENDRE, L. 1998. Numerical Ecology. Elsevier, Amsterdam.
- LESSA, E. 1990. Multidimensional analysis of geographic genetic structure. Systematics Zoology 39:242-252.
- LOVELESS, M.D. & HAMRICK, J.L. 1984. Ecological determinants of genetic structure in plant populations. Annual Review of Ecology and Systematics 15: 65-95.
- MANLY, B.F.J. 1997. Randomization, Bootstrap and Monte Carlo methods in biology. Chapman & Hall, London.
- MANTEL, N. 1967. The detection of disease clustering and a generalized regression approach. Cancer Research 27:209-220.
- MONTORO, G.R. & SANTOS, M.L. 2007. Fenologia e biologia reprodutiva de Tibouchina papyrus (Pohl) Toledo no Parque Estadual da Serra dos Pirineus. Pirenópolis, Goiás. Revista de Biologia Neotropical 4:21-29.
- MYERS, N., MITTERMEIER, R.A., MITTERMEIER, C.G., FONSECA, G.A.B. & DE KENT, J. 2000. Biodiversity hotspots for conservation priorities. Nature 403:853-858.
- NAMKOONG, G., BOYLE, T., EL-KASSABY, Y.A., PALBERG-LERCHE, C., ERISSON, G., GREGORIUS, H.R., JOLY, H., KREMER, J.A., SAVOLAINEM, O., WICKNESWARI, R., YOUNG, A., ZEH-NLO, M. & PRABHU, R. 2002. Criteria and indicators sustainable forest managent: assessment and monitoring of genetic variation. FAO, Roma.
- NEI, M. 1973. Analysis of gene diversity in subdivided populations. Proceedings of the National Academy of Sciences 70:3321-3323.
- PRIMACK, R.B. & RODRIGUES, E. 2001. Biologia da conservação. Editora Rodrigues, Londrina.
- RAMOS, J.R., TELLES, M.P.C., DINIZ-FILHO, J.A.F., SOARES, T.N., MELO, D.B. & OLIVEIRA, G. 2008. Optimizing reproducibility evaluation for random amplified polymorphic DNA markers. Genetics and Molecular Research 7:1384-1391.
- REIS, A.M.M. & GRATTAPAGLIA, D. 2004. RAPD variation in a germplasm collection of Myracrodruon urundeuva (Anacardiaceae), an endangered tropical tree: recommendations for conservation. Genetic Resources and Crop Evolution 51:529-538.
- SCHNEIDER, S., ROESSLI, D., EXCOFFIER, L. 2007. Arlequin Ver. 2.000. A software for population genetic data analysis. Universidade de Geneva, Geneva.
- SOARES, T.N., CHAVES, L.J., TELLES, M.P.C., DINIZ-FILHO, J.A.F. & RESENDE, L.V. 2008. Landscape conservation genetics of Dipteryx alata ("baru" tree Fabaceae) from Cerrado region of central Brazil. Genetica 132:9-19.
- TELLES, M.P.C., DINIZ-FILHO, J.A.F., COELHO, A.S.G. & CHAVES, L.J. 2001. Autocorrelação espacial das frequências alélicas em subpopulações de cagaiteira (Eugenia dysenterica DC, Myrtaceae) no Sudeste de Goiás. Revista Brasileira de Botânica 24:145-154.
- TRINDADE, M.G. & CHAVES, L.J. 2005. Genetic structure of natural Eugenia dysenterica DC (Myrtaceae) populations in northeastern Goiás, Brazil, accessed by morphological traits and RAPD markers. Genetics and Molecular Biology 28:407-413.
- VIEIRA, R.F. & MARTINS, M.V.M. 2000. Recursos genéticos de plantas medicinais do cerrado: uma compilação de dados. Revista Brasileira de Plantas Medicinais Botucatu 3:13-36.
- WEIR, B.S. 1996. Genetic data analysis II: methods for discrete population genetic data. Sinauer Associates, Sunderland.
- WRIGTH, S. 1951. The genetical structure of populations. Annals of Eugenics 15:323-354.
- ZUCCHI, M.I., PINHEIRO, J.B., CHAVES, L.J., COELHO, A.S., COUTO, M.A., MORAIS, L.K. & VENCOVSKY, R. 2005. Genetic structure and gene flow of Eugenia dysenterica natural populations. Pesquisa agropecuária brasileira 40:975-980.
Datas de Publicação
-
Publicação nesta coleção
01 Set 2010 -
Data do Fascículo
Jun 2010
Histórico
-
Recebido
17 Abr 2008 -
Aceito
25 Mar 2010