Resumos
O gênero Diadegma compreende espécies que são parasitóides de larvas da traça-das-crucíferas Plutella xylostella, uma das mais importantes pragas das plantas da família Brassicacea. Este gênero possui distribuição mundial. Neste trabalho, três populações de Diadegma spp. oriundas de diferentes países (Brasil, Ilha da Reunião e Malásia) foram caracterizadas geneticamente por meio da técnica de RAPD-PCR. Não foi constatada variabilidade intra-populacional, no entanto o alto coeficiente de similaridade entre populações sugeriram que esses insetos poderiam pertencer a espécies diferentes.
Plutella xylostella; inimigo natural; controle biológico; populações; marcadores moleculares
The genus Diadegma has species that are parasitoids of larvae of diamondback moth, Plutella xylostella, one of the most important pests of Brassicacea. This genus has a worldwide distribution. Representative samples of three Diadegma populations from Brazil, Reunion Island and Malaysia were characterized by RAPD-PCR. No intra-population variability was found. However, the high coefficient of genetic similarity between the populations suggests that they could belong to different species.
Plutella xylostella; natural enemy; biological control; population; molecular marker
PESQUISA
Variabilidade genética de Diadegma sp., parasitóide da traça-das-crucíferas, através de RAPD-PCR
Genetic variability of Diadegma sp., parasitoid of diamondback moth using RAPD-PCR
Rose MonneratI; Soraya Leal-BertioliI; David BertioliII; Tariq ButtIII; Dominique BordatIV
IEmbrapa Recursos Genéticos e Biotecnologia, C. Postal 02372, 70770-900 Brasilia-DF; E-mail: rose@cenargen.embrapa.br
IIUniversidade Católica de Brasília, 70790-160 Brasília-DF
IIIUniversity of Wales Swansea, Singleton Park, Swansea, SA2 8PP, Wales, UK
IVCIRAD-AMIS, BP 5035, 34032 Montpellier Cedex 1, France
RESUMO
O gênero Diadegma compreende espécies que são parasitóides de larvas da traça-das-crucíferas Plutella xylostella, uma das mais importantes pragas das plantas da família Brassicacea. Este gênero possui distribuição mundial. Neste trabalho, três populações de Diadegma spp. oriundas de diferentes países (Brasil, Ilha da Reunião e Malásia) foram caracterizadas geneticamente por meio da técnica de RAPD-PCR. Não foi constatada variabilidade intra-populacional, no entanto o alto coeficiente de similaridade entre populações sugeriram que esses insetos poderiam pertencer a espécies diferentes.
Palavras-chave:Plutella xylostella, inimigo natural, controle biológico, populações, marcadores moleculares.
ABSTRACT
The genus Diadegma has species that are parasitoids of larvae of diamondback moth, Plutella xylostella, one of the most important pests of Brassicacea. This genus has a worldwide distribution. Representative samples of three Diadegma populations from Brazil, Reunion Island and Malaysia were characterized by RAPD-PCR. No intra-population variability was found. However, the high coefficient of genetic similarity between the populations suggests that they could belong to different species.
Keywords:Plutella xylostella, natural enemy, biological control, population, molecular marker.
O gênero
Diadegma (Hymenoptera: Ichneumonidae) compreende espécies que são parasitóides de larvas de insetos de pequeno e médio tamanho e são consideradas muito eficientes para o controle da traça-das-crucíferas
Plutella xylostella, uma das mais importantes pragas das plantas da família Brassicacea (Lepidoptera: Yponomeutidae) (Talekar e Shelton, 1993; Monnerat e Bordat, 1998; Monnerat
et al., 2002; Guilloux
et al., 2003). Este gênero possui distribuição mundial. Na região do Distrito Federal foi constatado que este inseto pode parasitar naturalmente 40% das larvas da traça-das-crucíferas, sendo portanto uma importante ferramenta para o controle biológico em olericultura (Monnerat, 1995; Guilloux
et al., 2003).
Mais de 200 espécies já foram descritas, entretanto a até bem pouco tempo havia muitas dúvidas quanto à identificação segura destes insetos, pois a mesma se baseava em aspectos morfológicos, que não eram suficientes para separar as espécies. Assim, espécies morfologicamente idênticas podiam apresentar diferenças no comportamento, biologia e hospedeiros (Noyes, 1994). Ainda, a existência de híbridos entre espécies reconhecidamente diferentes como D. semiclausum (Hellen) e D. fenestrale (Holmgren) (Hardy, 1938) dificulta a sua identificação.
A fim de controlar P. xylostella, muitas introduções de populações de Diadegma foram realizadas em diferentes regiões do mundo (Oatman, 1978; Ooi, 1992; Talekar e Yang, 1991). Em muitos casos, as introduções foram feitas de forma desordenada, tornando extremamente difícil saber se uma certa espécie era endêmica ou introduzida.
Nesta última década, ferramentas de biologia molecular têm sido empregadas para caracterização de material biológico em diversas áreas. Dentre essas, merece ser destacada a técnica da reação em cadeia da polimerase (PCR) (Steffan e Atlas, 1991). Esta técnica tem permitido estudos de polimorfismo em diferentes populações de insetos (Haymer, 1994; Lima et al., 2000).
Este trabalho teve como objetivo comparar três populações de Diadegma sp. morfologicamente semelhantes, de espécies não determinadas, coletadas em diferentes partes do mundo, por meio da técnica de RAPD-PCR.
MATERIAL E MÉTODOS
Insetos
Os insetos analisados foram coletados na Ilha da Reunião (sudeste da África), Brasil e Malásia, criados em laboratório e estocados a -80ºC até serem utilizados. Os insetos coletados na Malásia foram identificados como D. semiclausum.
Dez indivíduos adultos de cada população foram utilizados para os testes intra-específicos. Dez grupos de dez adultos de cada população foram utilizados para os testes inter-específicos.
Extração e amplificação do DNA
Para a obtenção do DNA foram avaliados os protocolos de extração descritos por Black et al. (1992), Langridge et al. (1991) e Möller et al. (1992). O método descrito pelo primeiro autor foi utilizado para todos os testes. O protocolo consistiu em (1) macerar os insetos em tampão TES (100mM TRIS, pH 8,0, 10 mM EDTA, 2%SDS), (2) deixar o macerado em repouso por 5 minutos, (3) incubar com proteinase K a 37ºC por 15 minutos, (4) adicionar 100 ml de água e incubar por mais 15 minutos a 37ºC, (5) incubar a 94ºC por 5 minutos, (6) centrifugar por 5 minutos a 13.000 rpm, (7) coletar o sobrenadante, (8) estocar a 80ºC.
Um volume de 1 ml da solução contendo o DNA do inseto foi misturado com os componentes: 2,5 ml de tampão de reação, 1,5 ml do oligonucleotídeo (50 ng/ml), 0,25 ml dNTPs a 20 mM, 0,2 ml de DNA Taq Polimerase (5U/ml, Northumbria Biologicals), 19,55 ml de água bidestilada estéril.
Quinze oligonucleotídeos (kit Operon, UK) foram utilizados neste estudo (OPA7, OPB16, OPE6, OPE16, OPF13, OPK1, OPK2, OPK5, OPK7, OPK9, OPK10, OPK11, OPK15, OPK17 e OPK18). As amplificações foram realizadas em termociclador (Hybaid) programado da seguinte forma: (1) desnaturação inicial de 5 minutos a 94ºC, (2) 42 ciclos de desnaturação de 1 minuto a 92ºC, hibridação de 1 minuto a 35ºC e alongamento de 1 minuto a 72ºC, (3) alongamento final de 1 minuto a 72ºC.
Os produtos foram analisados em gel de agarose 0,8% em tampão TBE (45mM TRIS, 45mM de ácido bórico, 1mM EDTA pH 8,0) e as bandas visualizadas em luz ultravioleta.
Análise das bandas
Os perfis eletroforéticos foram analisados por meio do programa estatístico Genstat (Payne et al., 1987) e a matriz de similaridade genética foi calculada pelo coeficiente de similaridade de Jaccard (Sneath e Sokal, 1973).
RESULTADOS E DISCUSSÃO
Três métodos de extração de DNA foram avaliados, pois não havia nenhum método descrito especificamente para parasitóides. Dentre estes, o método de Black et al. (1992), descrito para extração de DNA de afídeos, foi selecionado para a realização do trabalho, pois foi o único que proporcionou uma boa extração de DNA. Os outros métodos, descritos para a extração de DNA de plantas (Langridge et al., 1991) e de fungos filamentosos (Möller et al., 1992), não foram apropriados por não resultarem em um DNA de boa qualidade.
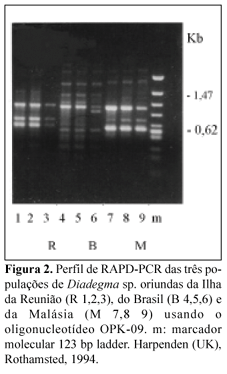
Nenhuma variabilidade genética intra-populacional foi detectada usando os 15 oligonucleotídeos mencionados (100% de similaridade), sugerindo que cada uma das populações são geneticamente homogêneas (Figura 1).
O RAPD-PCR entre as diferentes populações (Figura 2) apresentou 55 produtos diferentes. Houve grande variabilidade genética entre as populações analisadas (23,6 a 47,3%) (Tabela 1). A alta variabilidade genética entre as populações, revelada por RAPD-PCR neste trabalho, sugeriu que as mesmas pertenciam a espécies diferentes. De fato, estudos posteriores baseados nos critérios sugeridos por Azidah et al. (2000) confirmaram que a população isolada do Brasil era D. leontiniae e a população isolada da Ilha da Reunião era D. mollipla.
Os resultados preliminares obtidos com essas três espécies de Diadegma, morfologicamente idênticas, mostraram que esta técnica molecular tem potencial para auxiliar nos trabalhos de taxonomia, incluindo estudos de genética populacional, fluxo gênico e outros.
LITERATURA CITADA
Recebido para publicação em 06 de janeiro de 2003 e aceito em 10 de dezembro de 2003
- AZIDAH, A.A.; FITTON, M.; QUICKE, D.J.J. Identification of the Diadegma species (Hymenoptera: Ichneumonidae, Campopleginae) attaking the diamondback moth, Plutella xylostella (Lepidoptera: Plutellidae). Bulletin of Entomological Research, v.90, n.5, p.375-389. 2000.
- BLACK IV, W.C.; DU TEAU, N.M.; PUTERKA, G.J.; NECHOLS, J.R.; PETTORINI, J.M. Use of random amplified polymorphid DNA polymerase chain reaction (RAPD-PCR) to detect DNA polymorphisms in aphids (Homoptera: Aphididae). Bulletin of Entomological Research, v.82, p.151-159. 1992.
- GUILLOUX, T., MONNERAT, R.; CASTELO BRANCO, M.; KIRK, A; BORDAT, D. Population dynamics of Plutella xylostella (Lep., Yponomeutidae) and its parasitoids in the region of Brasilia. Journal of Applied Entomology, v.127, n.5, p.288-292. 2003.
- HAYMER, D.S. Arbitrary (RAPD) primer sequences used in insect studies. Insect Molecular Biology, v.3, p.191-194. 1994.
- HARDY, J.E. Plutella maculipennis Curt. its natural and biological control in England. Bulletin of Entomological Research, v.29, p.343-372. 1938.
- LANGRIDGE, U.; SCHWALL, A.; LANGRIDGE, P. Squashes of plant tissue as substrate for PCR. Nucleic Acids Research, v.19, p.69-54. 1991.
- LIMA, L.H.; NÁVIA, D.; INGLIS, P.W.; OLIVEIRA, M.R. Survey of Bemisia tabaci (Gennadius) (Hemíptera: Aleyrodidae) biotypes in Brazil using RAPD markers. Genetics and Molecular Biology, v.23, n.4, p.781-785. 2000.
- MÖLLER, E.M.; BAHNWEG, G.; SANDERMANN, H.; GEIGER, H.H. A simple and efficient protocol for isolation of high molecular weight DNA from filamentous fungi, fruit bodies, and infected plant tissues. Nucleic Acids Research, v.20, p.6115-6116. 1992.
- MONNERAT, R.G. Interrelations entre la teigne des cruciferes Plutella xylostella (Lep.: Yponomeitidae), son parasitoide Diadegma sp. (Hym.: Ichneumonidae) et la bacterie entomopathogene Bacillus thuringiensis Berliner. Ecole Nationale Superieure Agronimique de Montpellier, ENSA-M, 1995, 168 p. (Tese doutorado)
- MONNERAT, R.G.; BORDAT, D. Influence of HD-1 Bacillus thuringiensis spp. kurstaki on the developmental stages of Diadegma sp (Hym.: Ichneumonidae) parasitoid of diamondback moth (Lep.: Yponomeutidae). Journal of Applied Entomology, v.122, p.49-51, 1998.
- MONNERAT, R.G.; KIRK, A.A.; BORDAT, D. Biology of Diadegma sp. (Hymenoptera: Ichneumonidae), a parasitoid of Plutella xylostella (L.) (Lepidoptera: Yponomeutidae), from Reunion Island. Neotropical Entomology, v.3, n.2, p.271-274. 2002.
- NOYES, J.S. The reliability of published host-parasitoid records: a taxonomist's view. Norwegian Journal of Agricultural Science, v.16, p.56-69. 1994.
- OATMAN, E.R. Diamondback moth. In: CLAUSEN, C.P. (ed.) Introduced parasites and predators of arthropod pests and weeds: A world review. USDA Agriculture Handbook, 1978. 480 p.
- OOI, P.A.C. Diamondback moth in Malaysia. In: Talekar, N.S. (ed.) Diamonback Moth and other crucifer pests. Proceedings of the Second International Workshop. AVRDC. Taiwan, 1992. p.255-262.
- PAYNE, R.W.; LANE, P.W.; AINSLEY, A.E.; BICKNELL, K.E.; HARDING, P.G.N.; LEECH, P.K.; SIMPSON, H.R.; TODD, A.D.; VERRIER, P.J.; WHITE, R.J.; GOWER, J.C.; WILSON, G.T.; PATTERSON, G. Genstat 5 Reference Manual. Clarendon Press, Oxford. 1987.
- SNEATH, P.H.A.; SOKAL, R.R. Numerical Taxonomy: The principles and practice of numerical taxonomy San Francisco: W.H. Freeman, 1973. 573 p.
- STEFFAN, R.J.; ATLAS, R.M. Polymerase Chain Reaction: Applications in Environmental Microbiology. Annual Review of Microbiology , v.45, p.137-161. 1991.
- TALEKAR, N.S.; SHELTON, A.M. Biology, ecology, and management of the diamondback moth. Annual Review of Entomology, v.38, p.275-301. 1993.
- TALEKAR, N.S.; YANG, J.C. Characteristic of parasitism of Diamondback moth by two larval parasites. Entomophaga, v.36, n.1, p.95-104. 1991.
Datas de Publicação
-
Publicação nesta coleção
26 Maio 2004 -
Data do Fascículo
Mar 2004
Histórico
-
Aceito
10 Dez 2003 -
Recebido
06 Jan 2003