Resumos
Foram obtidas estimativas de herdabilidade e preditas diferenças esperadas na progênie para probabilidade de permanência no rebanho (stayability) de 4180 touros com filhas na base de dados do Programa de Melhoramento Genético da Raça Nelore. Modelo de limiar e modelo linear foram utilizados sob análise Bayesiana via software multiple-trait Gibbs sampler for animal models. A implementação adotada considerou tamanho de cadeia de Gibbs de 225 mil, período de descarte amostral de 25 mil e tomada de amostra a cada mil rodadas. As estimativas de herdabilidade foram de menor magnitude para o modelo linear, 0,065, contra 0,158 sob modelo de limiar. Quando transformadas para escala normal subjacente, o valor obtido ficou em 0,13±0,05, bem próximo àquele encontrado sob modelo de limiar. A correlação entre classificações foi de 97%. O modelo de análise considerado para stayability, sob enfoque bayesiano, parece não influenciar a classificação dos animais quanto aos valores genéticos preditos. Análises sob modelo linear, com reduzido tempo de processamento, poderiam ser preferidas bastando transformar a escala da característica para obter a estimativa de herdabilidade.
bovino; Nelore; permanência no rebanho; Bayesiana; modelo linear
Heritability and expected progeny difference (EPD) for stayability based on progeny records of 4,180 sires from the Programa de Melhoramento Genético da Raça Nelore were estimated. Bayesian analyses for threshold and linear models were performed using the software multiple trait Gibbs sampler for animal model. Gibbs size chain of 225,000, burn-in of 25,000 and thinning interval of 1,000 cycles were considered for implementation purpose. The heritability estimated by linear model (.065) was lower than for threshold model (0.158). The heritability estimate for transformed continuous threshold scale was .13±.05 which is close to the heritability estimate for threshold model. The high rank correlation between the solutions from both models (97%) suggests the stayability analysis under Bayesian perspective does not influence the rank of the animal EPD. Therefore linear model analysis for transformed threshold stayability data, which has a reduced processing time, should be used.
beef cattle; Nellore; stayability; Bayesian; linear model; threshold
ZOOTECNIA E TECNOLOGIA E INSPEÇÃO DE PRODUTOS DE ORIGEM ANIMAL
Comparação entre análises para permanência no rebanho de vacas Nelore utilizando modelo linear e modelo de limiar
Threshold versus linear model analyses in stayability for Nellore cows
C.R. MarcondesI; J.C.C. PanetoI; J.A.II V. SilvaII; H.N. OliveiraIII; R.B. LôboIV, * * Autor para correspondência ( corresponding author) E-mail: cimarcon@rge.fmrp.usp.br
IAluno de Pós-Graduação - Faculdade de Medicina de Ribeirão Preto USP
IIFaculdade de Zootecnia e Engenharia de Alimentos USP Pirassununga, SP
IIIFaculdade de Ciências Agronômicas UNESP Botucatu, SP
IVFaculdade de Medicina de Ribeirão Preto USP - Departamento de Genética - Av. Bandeirantes, 3900 - 14049-900 Ribeirão Preto, SP
RESUMO
Foram obtidas estimativas de herdabilidade e preditas diferenças esperadas na progênie para probabilidade de permanência no rebanho (stayability) de 4180 touros com filhas na base de dados do Programa de Melhoramento Genético da Raça Nelore. Modelo de limiar e modelo linear foram utilizados sob análise Bayesiana via software multiple-trait Gibbs sampler for animal models. A implementação adotada considerou tamanho de cadeia de Gibbs de 225 mil, período de descarte amostral de 25 mil e tomada de amostra a cada mil rodadas. As estimativas de herdabilidade foram de menor magnitude para o modelo linear, 0,065, contra 0,158 sob modelo de limiar. Quando transformadas para escala normal subjacente, o valor obtido ficou em 0,13±0,05, bem próximo àquele encontrado sob modelo de limiar. A correlação entre classificações foi de 97%. O modelo de análise considerado para stayability, sob enfoque bayesiano, parece não influenciar a classificação dos animais quanto aos valores genéticos preditos. Análises sob modelo linear, com reduzido tempo de processamento, poderiam ser preferidas bastando transformar a escala da característica para obter a estimativa de herdabilidade.
Palavras-chave: bovino, Nelore, permanência no rebanho, Bayesiana, modelo linear
ABSTRACT
Heritability and expected progeny difference (EPD) for stayability based on progeny records of 4,180 sires from the Programa de Melhoramento Genético da Raça Nelore were estimated. Bayesian analyses for threshold and linear models were performed using the software multiple trait Gibbs sampler for animal model. Gibbs size chain of 225,000, burn-in of 25,000 and thinning interval of 1,000 cycles were considered for implementation purpose. The heritability estimated by linear model (.065) was lower than for threshold model (0.158). The heritability estimate for transformed continuous threshold scale was .13±.05 which is close to the heritability estimate for threshold model. The high rank correlation between the solutions from both models (97%) suggests the stayability analysis under Bayesian perspective does not influence the rank of the animal EPD. Therefore linear model analysis for transformed threshold stayability data, which has a reduced processing time, should be used.
Keywords: beef cattle, Nellore, stayability, Bayesian, linear model, threshold
INTRODUÇÃO
Para a raça Nelore, no Brasil, existem poucos estudos sobre stayability (Paneto et al., 2002; Silva et al., 2003a,b), principalmente utilizando ferramentas Bayesianas na estimação dos componentes de (co)variância e dos parâmetros genéticos e na predição de valores genéticos. O desenvolvimento de metodologias específicas para análise de dados categóricos surgiu graças às necessidades dos pesquisadores em estudar e aplicar características como dificuldade de parição e probabilidade de permanência no rebanho. Anteriormente, modelos lineares resultavam em estimativas de herdabilidade não diferentes de zero para stayability em bovinos leiteiros e de corte (Hudson e Van Vleck, 1981; Van Doormaal et al., 1985; Short e Lawlor, 1992; Snelling et al., 1995; Mwansa et al., 2002). Segundo Ducrocq et al. (1988), os métodos não lineares poderiam ter maior habilidade para detecção da variabilidade genética do que os métodos lineares.
A melhor predição linear não viesada (BLUP), conforme salientado por Thompson (1979), Gianola (1980; 1982) e Hoeschele (1986), não seria apropriada para as variáveis categóricas, pois elas violariam várias pressuposições existentes nas metodologias de modelos mistos. Os autores identificaram e discutiram alguns problemas nas análises de dados binomiais com modelo linear. Alguns dos problemas mais críticos são: 1) as variâncias das características categóricas são heterogêneas, 2) as soluções do BLUP não tomam em consideração a restrição de que a soma das probabilidades de resposta tenha que ser igual a 1 entre todas as categorias de resposta, 3) a variância genética aditiva na escala observada depende da incidência da característica na população, 4) a possibilidade que o efeito genético não aditivo esteja presente na escala observada no caso em que toda a variação genética é aditiva na escala subjacente, e 5) quando a esperança condicional das predições dado os dados é não linear, as propriedades de classificação do melhor preditor linear não parecem ser otimizados.
Os procedimentos para análise de dados genéticos descontínuos estão divididos em dois grupos, e a distinção entre eles depende se se assume ou não a distribuição contínua subjacente. O primeiro grupo inclui métodos que ignoram a natureza discreta dos dados categóricos e a análise procede ao se usar o procedimento linear como se os dados fossem contínuos. O segundo envolve o conceito de limiar (ou threshold) (Falconer, 1989), que assume uma distribuição normal, não observável, subjacente para a variável discreta mensurada. A conexão da variável discreta e a escala contínua subjacente é gerada por um conjunto de limiares fixos. Enfim, a variável subjacente é descrita pelo modelo linear, mas a relação da subjacente e a externa ou escala observada é não linear. Vários autores (Hoeschele, 1986; Foulley et al., 1987; Soto-Murrillo, 1991) apresentaram e discutiram várias estratégias de análises genéticas de dados discretos em melhoramento animal. Os procedimentos lineares são descritos como não apropriados para quantificar a natureza discreta dos dados categóricos.
Segundo Ramirez-Valverde et al. (2001), o modelo de limiar ofereceria vantagens sobre o modelo linear nas análises sob modelo animal, mas não sob modelo de touro-avô materno, para a característica de dificuldade de parição da raça Gelbvieh americana.
O objetivo do presente trabalho foi comparar estimativas de herdabilidade e correlação entre classificações (rank), obtidas em análises bayesianas sob modelo linear e limiar de touro-avô materno.
MATERIAL E MÉTODOS
O arquivo geral de dados, com vacas nascidas entre 1986 e 1996 em rebanhos participantes do Programa de Melhoramento Genético da Raça Nelore, conduzido pela Faculdade de Medicina de Ribeirão Preto da Universidade de São Paulo (PMGRN-USP) (Lôbo et al., 2002), continha 54.727 registros de produção.
A característica probabilidade de permanência no rebanho (stayability) foi codificada com valor 1 para vacas com, pelo menos, três partos até os 76 meses de idade ou valor 0 para as que tiveram menos de três partos. Definiram-se três partos porque a vaca estaria "pagando" seus custos de cria e recria e 76 meses para que tivesse a chance de parir três vezes, com o primeiro parto em torno de 32 meses. A porcentagem de sucesso (valor 1) foi 37% no arquivo estudado. O grupo de contemporâneos ao nascimento (composto por fazenda e ano de nascimento da vaca) geralmente é o mais usado, por contemplar maior número de animais.
A consistência dos dados, realizada por meio do programa Statistical Analysis System (User's..., 2000), considerou touros com pelo menos duas filhas dentro do mesmo grupo de contemporâneo (GC) e GCs com pelo menos dois touros diferentes. Foram eliminados grupos com médias 0 ou 1 para stayability, isto é, sem variabilidade dentro do grupo de contemporâneos.
Utilizou-se o multiple-trait Gibbs sampler for animal models (MTGSAM for threshold; Van Tassell et al., 1998) sob modelo de limiar unicaráter de touro-avô materno, para obtenção de componentes de (co)variância, estimativas de herdabilidade e soluções para cada touro ((médias a posteriori que transformadas sob escala de probabilidade resultam em diferenças esperadas na progênie (DEPs)). Adotou-se o modelo touro-avô materno, após tentativas de convergência sob modelo animal. Essa ocorrência é normal e há outros relatos dela na literatura (Moreno et al., 1997; Phocas e Laloë, 2003). Os métodos bayesianos, basicamente, são compostos pela resolução de integrais complicadas. A implementação das cadeias de Markov ou MCMC (entre elas, a amostragem de Gibbs) resolveu parte desse problema, gerando outro: a convergência das cadeias nem sempre é possível e geralmente solucionada somente quando a distribuição dos dados é normal (Blasco, 2001). Segundo Van Vleck (1993), o modelo de touro-avô materno assegura predições mais acuradas dos valores genéticos.
O modelo de limiar usado relaciona a resposta observada na escala categórica com uma escala subjacente normal contínua (Silva et al., 2003b), admitindo-se que a escala subjacente tem distribuição normal:
U | q ~ N(Wq, I ), em que:
), em que:
U é o vetor da escala base de ordem r, q' = (b', a') é o vetor dos parâmetros de locação de ordem s com b (definidos sob um ponto de vista freqüentista, como efeitos fixos) e a (como efeito aleatório); W é uma matriz de incidência conhecida de ordem r por s; I é matriz de identidade de ordem r por r; e  é a variância residual.
é a variância residual.
De acordo com o enfoque bayesiano, admite-se que as distribuições a priori dos efeitos genéticos e residuais seguem distribuição normal multivariada:
p (a |  ) ~ N (0, A
) ~ N (0, A )
)
p (e |  ) ~ N (0, I
) ~ N (0, I ), em que:
), em que:
A é a matriz de parentesco e  é a variância genética aditiva. No caso deste estudo, ¼ da
é a variância genética aditiva. No caso deste estudo, ¼ da  foi estimada pela variância de touro (
foi estimada pela variância de touro ( ). Como
). Como  não é estimável (Gianola e Foulley, 1983), valor arbitrário deve ser então atribuído. Conforme sugerido por Silva et al. (2003b), atribuiu-se o valor 1.
não é estimável (Gianola e Foulley, 1983), valor arbitrário deve ser então atribuído. Conforme sugerido por Silva et al. (2003b), atribuiu-se o valor 1.
Após a definição dos parâmetros do modelo, o encadeamento entre as duas escalas (categórica e contínua) pode ser estabelecido inequivocamente, com a contribuição da probabilidade de uma observação que cai na primeira categoria se proporcional a
P(Yv=0 | t, q)= P(Uv< t | t, q) = F ((t W'vq) / se), em que:
Yv, é a variável resposta para a observação v, tomando valores 0 ou 1 se a observação pertence à primeira ou segunda categoria, respectivamente; t é o valor do limiar que, por não ser estimável, será fixado um valor arbitrário; Uv é o valor da variável subjacente para a mencionada observação; F é a função de distribuição cumulativa de uma variável normal padrão; e W'v é um vetor coluna de incidência que une q a observação v. Por serem as observações condicionalmente independentes dado q, a função de verossimilhança será definida pelo produto da contribuição de cada registro.
De acordo com a inferência bayesiana, o produto da distribuição anterior pela função de verossimilhança é proporcional à distribuição posterior conjunta de q e  . Foram então obtidas as distribuições posteriores marginais dos parâmetros usando a amostragem de Gibbs (Gelfand e Smith, 1990).
. Foram então obtidas as distribuições posteriores marginais dos parâmetros usando a amostragem de Gibbs (Gelfand e Smith, 1990).
Por tratar-se de análise unicaráter, as relações entre as variâncias estimadas sob modelo touro-avô materno foram: variância genética aditiva considerada como 4 , a variância ambiental como sendo igual à variância residual (no caso de modelo de limiar, fixa com valor 1), a variância fenotípica como a soma de (
, a variância ambiental como sendo igual à variância residual (no caso de modelo de limiar, fixa com valor 1), a variância fenotípica como a soma de ( +¼
+¼ ) ao 1 e, conseqüentemente, a herdabilidade estimada sob modelo touro como sendo a porção herdável da variância total existente.
) ao 1 e, conseqüentemente, a herdabilidade estimada sob modelo touro como sendo a porção herdável da variância total existente.
A implementação adotada para a base de dados do PMGRN-USP considerou tamanho de cadeia de Gibbs de 225 mil, período de descarte amostral de 25 mil e tomada de amostra a cada mil rodadas.
Considerando-se a mesma implementação e utilizando-se o mesmo software de análise, somente alterando a opção para considerar o modelo como linear (opção 0 no módulo mtgsprep.exe), foi analisada a stayability para, principalmente, comparar a classificação dos touros. A vantagem do modelo linear é a maior velocidade de processamento das análises, porém, como mencionado anteriormente, resulta em estimativas de herdabilidade baixas ou próximas a zero. Uma alternativa para contornar esse problema seria a transformação da herdabilidade para escala normal subjacente, conforme sugerido por Robertson em Dempster e Lerner (1950):
 , em que
, em que
h2icont é a herdabilidade em escala contínua subjacente;
h2iobs é a herdabilidade na escala observada (binária);
pi é a freqüência de saída para a característica i;
f e F relacionados à distribuição acumulada.
A matriz de parentesco continha 4180 touros, 454 endogâmicos (coeficiente de endogamia médio igual a 5%).
A correlação de rank das soluções com modelo linear e limiar foi obtida por meio do programa SAS, opção Spearman.
RESULTADOS E DISCUSSÃO
A Tab. 1 apresenta as estimativas dos componentes genéticos (variância de touro e residual) e da herdabilidade, sob dois modelos diferentes (de limiar, ou threshold, e linear).
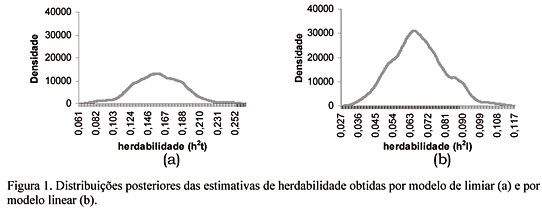
As estimativas de herdabilidade, comparando-se os modelos usados, foram de menor magnitude para o modelo linear (quase um terço do valor estimado sob modelo de limiar), em conformidade aos estudos de Hudson e Van Vleck (1981), Van Doormaal et al. (1985), Short e Lawlor (1992), Snelling et al. (1995) e Mwansa et al. (2002). Observa-se, porém, que a região de credibilidade da herdabilidade para o modelo de limiar (0,10 a 0,22) é muito mais ampla do que para o modelo linear (0,04 a 0,09), devido, respectivamente, às distribuições platicúrtica e leptocúrtica (Fig. 1).
Quando as estimativas de herdabilidade, resultantes das análises por modelo linear, foram transformadas para escala normal subjacente, o valor obtido foi de 0,13±0,05, com região de credibilidade entre 0,06 e 0,21, valores bem próximos àqueles encontrados sob modelo de limiar (Tab. 1).
Na raça Hereford, Martínez et al. (2002) não encontraram diferenças entre estimativas de herdabilidade obtidas por modelo linear misto generalizado para dados binários e modelo linear misto com máxima verossimilhança restrita, utilizando MATVEC (os valores para stayability aos 72 meses de idade foram para limiar e linear, respectivamente, 0,49±0,18 e 0,31±10).
As DEPs preditas e transformadas para escala de probabilidade, conforme explicado anteriormente, estão na Tab. 2.
O modelo de limiar foi mais eficiente na detecção da variabilidade existente entre os touros, dada a maior amplitude entre as DEPs mínima e máxima (26% versus 8% do modelo linear).
A correlação de rank foi de 97%, ou seja, haveria pouca alteração na classificação dos 4180 avaliados.
Quando a mesma correlação foi aplicada sobre um por cento dos melhores touros (42 touros) e sobre um por cento dos piores, o valor ficou acima de 99%. As mudanças de rank dos touros, portanto, ocorreriam nas posições intermediárias. Os resultados estão em acordo com as observações de Matos et al. (1997b) que, em geral, observaram altas correlações entre os valores genéticos dos touros preditos sob modelos linear e de limiar. Perez-Enciso et al. (1993) sugeriram que esse critério poderia não ser verdadeiro e utilizaram a capacidade de ajustamento (denominada goodness of fit), medida pelo quadrado médio do erro e pela correlação entre o valor observado e o valor ajustado, e a habilidade de predição (observações futuras a partir de dados passados baseando-se nos parâmetros de dispersão), para comparar modelos mistos lineares e de Poisson para dados de tamanho de leitegada em suínos.
As conclusões de Matos et al. (1997b), no entanto, foram de que modelos não-lineares como os de limiar, os quais possuem vantagens teóricas sobre os modelos lineares mistos, não se mostraram melhores para características reprodutivas de ovelhas das raças Rambouillet e Finnsheep, não incentivando o uso dos modelos de limiar, especialmente considerando-se as dificuldades computacionais. Outro aspecto interessante, citado por Matos et al. (1997a), seria o de que as vantagens do uso dos modelos de limiar sobre os modelos lineares aumentariam quando as herdabilidades das características utilizadas nos programas de seleção decrescessem. Abdel-Azim e Berger (1999) destacaram que a verdadeira importância do uso de modelos de limiar seria a estimação de componentes de variância e que a acurácia das estimativas aumentaria quando aumentasse o número de categorias na característica, quando a distribuição estivesse mais próxima da normal ou quando houvesse número balanceado de dados.
CONCLUSÕES
Para um programa de melhoramento, cujo principal produto é a avaliação genética dos animais, o modelo de análise considerado para stayability, linear e de limiar sob enfoque Bayesiano, não influenciou a classificação dos animais quanto aos valores genéticos preditos. Assim, análises sob modelo linear, de menor tempo de processamento, poderiam ser preferidas quando houver muitos registros de produção, bastando transformar a escala das estimativas de herdabilidade.
AGRADECIMENTOS
Os autores agradecem o apoio financeiro do PRONEX, FAPESP, CNPq, FINEP, ANCP e aos criadores do PMGRN.
Recebido para publicação em 19 de março de 2004
Recebido para publicação, após modificações, em 10 de setembro de 2004
- ABDEL-AZIM, G.A.; BERGER, P.J. Properties of threshold model predictions. J. Anim. Sci., v.77, p.582-590, 1999.
- BLASCO, A. The Bayesian controversy in animal breeding. J. Anim. Sci., v.79, p.2023-2046, 2001.
- DEMPSTER, E. R.; LERNER, I.M. Heritability of threshold characters. With an appendix by A. Robertson. Genetics, v.35, p.212-236, 1950.
- DUCROCQ, V.; QUAAS, R.L.; POLLAK, E.J. et al. Lenght of productive life of dairy cows. 2. Variance component estimation and sire evaluation. J. Dairy Sci., v.71, p.3071-3079, 1988.
- FALCONER, D.S. Introduction to Quantitative Genetics 3.ed. New York: Longman Scientific and Technical. 1989.
- FOULLEY, J.L.; IM, S.; GIANOLA, D. et al. Empirical Bayes estimation of parameters for n polygenic binary traits. Genet. Sel. Evol., v.19, p.197, 1987.
- GELFAND, A.E.; SMITH, A.F.M. Sampling based approaches to calculating marginal densities. J. Am. Stat. Assoc, v.85, p.398-409, 1990.
- GIANOLA, D. A method of sire evaluation for dichotomies. J. Anim. Sci., v.51, p.1266, 1980.
- GIANOLA, D. Theory and analysis of threshold characters. J. Anim. Sci., v.54, p.1079-1096, 1982.
- GIANOLA, D.; FOULLEY, J.L. Sire evaluation for ordered categorical data with a threshold model. Gen. Sel. Evol, v.15, p.201-224, 1983.
- HUDSON, G.F.S.; VAN VLECK, L.D. Relations between production and stayability in Holstein cattle. J. Dairy Sci., v.64, p.2246-2250, 1981.
- LÔBO, R.B.; BEZERRA, L.A.F.; OLIVEIRA, H.N. et al. Avaliação genética de animais jovens, touros e matrizes. Ribeirão Preto, USP/ FMRP/ GEMAC/ Departamento de Genética, 2002. 76p.
- MARTÍNEZ, G.E.; KOCH, R.M.; CUNDIFF, L.V. et al. Genetic parameters and genetic trends for stayability in Hereford cows. In: WORLD CONGRESS ON GENETICS APPLIED TO LIVESTOCK PRODUCTION, 7., 2002, Montpellier, França. Anais Montpellier, França, 2002. Seção 2, Comunicação 02-32 (CD-ROM).
- MATOS, C.A.P.; THOMAS, D.L.; GIANOLA, D. et al. Genetic analysis of discrete reproductive traits in sheep using linear and nonlinear models: I. Estimation of genetic parameters. J. Anim. Sci., v.75, p.76-87, 1997a.
- MATOS, C.A.P.; THOMAS, D.L.; GIANOLA, D. et al. Genetic analysis of discrete reproductive traits in sheep using linear and nonlinear models: II. Goodness of fit and predictive ability. J. Anim. Sci., v.75, p.88-94, 1997b.
- MORENO, C.; SORENSEN, D.; GARCIA-CORTÉS, L.A. et al. On biased inferences about variance components in the binary threshold model. Genet. Sel. Evol., v.29, p.145-160, 1997.
- MWANSA, P.B.; CREWS, D.H. Jr.; WILTON, J.W. et al. Multiple trait selection for maternal productivity in beef cattle. J. Anim. Breed. Genet., v.119, p.391-399, 2002.
- PANETO, J.C.C.; SILVA, J.A. II V.; BEZERRA, L.A.F. et al. Expected response to selection on stayability and its economic weight in a population of Nelore cattle in Brazil. In: WORLD CONGRESS ON GENETICS APPLIED TO LIVESTOCK PRODUCTION, 7. 2002, Montpellier. Anais Montpellier, França, 2002. Seção 2, Comunicação 02-67 (CD-ROM).
- PEREZ-ENCISO, M.; TEMPELMAN, R.J.; GIANOLA, D. A comparison between linear and Poisson mixed models for litter size in Iberian pigs. Livest. Prod. Sci., v.35, p.303, 1993.
- PHOCAS, F.; LALOË, D. Evaluation models and genetic parameters for calving difficulty in beef cattle. J. Anim. Sci., v.81, p.933-938, 2003.
- RAMIREZ-VALVERDE, R.; MISZTAL, I.; BERTRAND, J.K. Comparison of threshold vs linear and animal vs sire models for predicting direct and maternal genetic effects on calving difficulty in beef cattle. J. Anim. Sci., v.79, p.333-338, 2001.
- SHORT, T.H.; LAWLOR, T.J. Genetic parameters of conformation traits, milk yield and herd life in Holsteins. J. Dairy Sci., v.75, p.1987-1998, 1992.
- SILVA, J.A.II V.; ELER, J.P.; FERRAZ, J.B.S. et al. Heritability estimate for stayability in Nelore cows. Livest. Prod. Sci., v.79, p.97-101, 2003a.
- SILVA, J.A.II V.; ELER, J.P.; OLIVEIRA, H.N. et al. Análise genética da habilidade de permanência em fêmeas da raça Nelore. Rev. Bras. Zootec., v.32, p.598-604, 2003b.
- SNELLING, W.M.; GOLDEN, B.L.; BOURDON, R.M. Within-herd genetic analysis of stayability of beef females. J. Anim. Sci., v.73, p.993-1001, 1995.
- SOTO-MURRILLO, H.W. Methods for checking the goodness of fit of alternative nonlinear mixed models with an application in fertility traits in beef cows 1991. Dissertation (PhD). University of Illinois, Urbana.
- THOMPSON, R. Sire evaluation. Biometrics, v.35, p.339-353, 1979.
- VAN DOORMAAL, B.J.; SCHAEFFER, L.R.; KENNEDY, B.W. Estimation of genetic parameters for stayability in Canadian Holsteins. J. Dairy Sci., v.68, p.1763-1769, 1985.
- VAN TASSELL, C.P.; VAN VLECK, L.D.; GREGORY, K.E. Bayesian analysis of twinning and ovulation rates using a Multiple-Trait Threshold Model and Gibbs Sampling. J. Anim. Sci., v.76, p.2048-2061, 1998.
- VAN VLECK, L.D. Relationships from sires and maternal grandsires of males. In: Selection index and introduction to mixed model methods for genetic improvement of animals, the green book. Boca Raton: CRC, 1993. p.298
Datas de Publicação
-
Publicação nesta coleção
02 Ago 2005 -
Data do Fascículo
Abr 2005
Histórico
-
Aceito
10 Set 2004 -
Recebido
19 Mar 2004