Resumos
Determinou-se o número adequado de repetições na comparação de métodos de seleção tradicionais e associados a marcadores moleculares, com diferentes tamanhos efetivos e sob diferentes sistemas de acasalamento dos reprodutores selecionados, usando simulação com o programa GENESYS. Para comparar os diferentes métodos de seleção utilizaram-se populações com tamanhos efetivos de 18,18 (TE1) e de 66,66 (TE2) e uma, 10 e 30 repetições por geração, avaliando-se os valores fenotípicos médios. Para as situações com apenas uma repetição, os resultados apresentaram incoerências, independentemente do tamanho efetivo (TE1 ou TE2) ou do sistema de acasalamento (RAA - reprodutores acasalados aleatoriamente, EIC - exclusão de irmãos completos ou EICMI - exclusão de irmãos completos e meio-irmãos). Observou-se que a oscilação genética influencia o ganho genético, principalmente, em populações com pequeno tamanho efetivo e que um valor mínimo de 10 repetições por geração é necessário para assegurar a consistência dos resultados obtidos pelos métodos de seleção.
BLUP; marcador molecular; tamanho efetivo de população; sistema de acasalamento
This work was carried out to determine the required number of replicates for comparison of conventional and molecular marker-associated selection methods in distinct effective population sizes and different mating systems, by simulations using the software GENESYS. Effective populational size of 18.18 (ES1) and 66.66 (ES2), and one, 10, and 30 replicates per generation were used to compare the different selection methods, based on mean phenotypic values. Incongruences results were observed when a single replicate was considered independently of effective size (ES1or ES2) and the mating system (random mating; exclusion of full-sibs or exclusion of both full and half-sibs). Genetic oscillation influenced the genetic gain, mainly in populations of small effective size. Furthermore, at least 10 replicates per generation were required to obtain sound consistent results for both selection methods.
BLUP; molecular marker; effective population size; mating system
ZOOTECNIA E TECNOLOGIA E INSPEÇÃO DE PRODUTOS DE ORIGEM ANIMAL
Comparação de metodologias de seleção sob oscilação genética
Comparison of selection methodologies under genetic drift
P.L.S. CarneiroI; C.H.M. MalhadoI; P.R.A.M. AffonsoI; R.F. EuclydesII; A.P.S. CarneiroIII; E.E. CunhaIV; L.G.R. SouzaV
IDepartamento de Ciências Biológicas - UESB - Av. José Moreira s/n - 45200-000 - Jequié, BA
IIDepartamento de Zootecnia - UFV - Viçosa, MG
IIIDepartamento de Informática - UFV - Viçosa, MG
IVDepartamento de Genética - UFRN - Natal, RN
VDepartamento de Botânica - UFPE - Recife, PE
RESUMO
Determinou-se o número adequado de repetições na comparação de métodos de seleção tradicionais e associados a marcadores moleculares, com diferentes tamanhos efetivos e sob diferentes sistemas de acasalamento dos reprodutores selecionados, usando simulação com o programa GENESYS. Para comparar os diferentes métodos de seleção utilizaram-se populações com tamanhos efetivos de 18,18 (TE1) e de 66,66 (TE2) e uma, 10 e 30 repetições por geração, avaliando-se os valores fenotípicos médios. Para as situações com apenas uma repetição, os resultados apresentaram incoerências, independentemente do tamanho efetivo (TE1 ou TE2) ou do sistema de acasalamento (RAA - reprodutores acasalados aleatoriamente, EIC - exclusão de irmãos completos ou EICMI - exclusão de irmãos completos e meio-irmãos). Observou-se que a oscilação genética influencia o ganho genético, principalmente, em populações com pequeno tamanho efetivo e que um valor mínimo de 10 repetições por geração é necessário para assegurar a consistência dos resultados obtidos pelos métodos de seleção.
Palavras-chave: BLUP, marcador molecular, tamanho efetivo de população, sistema de acasalamento
ABSTRACT
This work was carried out to determine the required number of replicates for comparison of conventional and molecular marker-associated selection methods in distinct effective population sizes and different mating systems, by simulations using the software GENESYS. Effective populational size of 18.18 (ES1) and 66.66 (ES2), and one, 10, and 30 replicates per generation were used to compare the different selection methods, based on mean phenotypic values. Incongruences results were observed when a single replicate was considered independently of effective size (ES1or ES2) and the mating system (random mating; exclusion of full-sibs or exclusion of both full and half-sibs). Genetic oscillation influenced the genetic gain, mainly in populations of small effective size. Furthermore, at least 10 replicates per generation were required to obtain sound consistent results for both selection methods.
Keywords: BLUP, molecular marker, effective population size, mating system
INTRODUÇÃO
A resposta à seleção para uma única característica depende da variabilidade genética, intervalo de gerações, intensidade de seleção, tamanho efetivo da população e acurácia da seleção. Dentre estes fatores, o tamanho efetivo da população é fundamental. De acordo com Falconer e Mackay (1996), a resposta genética de um ciclo de seleção para características quantitativas é função da acurácia de seleção, da intensidade de seleção e da variância genética aditiva na população. Para seleção em longo prazo, como citado por Wei et al. (1996), em pequenas populações, outros fatores como oscilação genética, desequilíbrio gamético, variância mutacional, tamanho efetivo de população, estrutura da população e estratégia de seleção devem ser incorporados para predizer a resposta acumulada da seleção.
De acordo com Wei et al. (1996), a seleção individual, o índice de seleção e a seleção com base nos valores genéticos preditos pelo BLUP (melhor preditor linear não viesado) diferem na acurácia da predição dos valores genéticos. A curto prazo, a maior acurácia da avaliação genética aumenta a resposta à seleção. Porém, caso não haja controle sobre os acasalamentos, a seleção de maior número de indivíduos da mesma família, pode aumentar a taxa de endogamia, reduzindo a resposta à seleção. A longo prazo, todos esses métodos de avaliação genética alcançam máxima resposta sem contudo otimizá-la. Belonsky e Kennedy (1988) concluíram que o BLUP proporciona mais alta resposta que a seleção individual para dez gerações de seleção, porque o aumento da acurácia na seleção pelos valores genéticos preditos pelo BLUP é mais importante que a redução no tamanho efetivo da população. Segundo Cunha et al. (2006), no processo seletivo, é importante considerar simultaneamente o método de seleção bem como o tamanho efetivo da população. Verrier et al. (1993) utilizaram diferentes tamanhos de população e mostraram que a seleção individual é mais vantajosa do que o BLUP, quando a população é pequena.
Meuwissen e Woolliams (1994) frisaram o fato de que novos métodos de avaliação genética têm sido desenvolvidos, em melhoramento animal, para aumentar o ganho genético, por exemplo o BLUP e os núcleos MOET (técnicas de ovulação múltipla e transferência de embriões). Entretanto, para estes métodos, as taxas de endogamia aumentam concomitantemente, de modo mais intenso em pequenas populações, como no caso dos núcleos MOET e de pequenos núcleos para conservação genética. Segundo Muir (2000), o primeiro impacto da endogamia é a perda de alelos atribuída à oscilação genética.
Muitos estudos com simulação têm sido realizados comparando-se métodos de seleção em populações de diferentes tamanhos. Entretanto, o número de repetições do processo de simulação utilizado por geração de seleção para minimizar o efeito da oscilação genética não está bem definido. Assim, o objetivo neste trabalho foi determinar o número adequado de repetições do processo de simulação na comparação de métodos de avaliação tradicionais e associados a marcadores moleculares, para populações com diferentes tamanhos efetivos e sob diferentes sistemas de acasalamento dos reprodutores selecionados.
MATERIAL E MÉTODOS
Os dados utilizados foram obtidos a partir do programa de simulação GENESYS (Euclydes e Guimarães, 1997), desenvolvido para o compilador FORTRAN. Esse sistema de simulação baseia-se no princípio de que a expressão de uma característica é determinada pela contribuição de uma série de genes e por efeitos de ambiente.
Para possibilitar o desenvolvimento deste trabalho foi simulado um genoma constituído de uma característica quantitativa com valor de herdabilidade igual a 0,10. As características desse genoma foram: 200 locos quantitativos polialélicos (oito alelos); os locos foram distribuídos ao acaso em 15 pares de cromossomos autossômicos de tamanhos aleatórios, em genoma de dois mil centimorgans de comprimento. Foram simulados apenas efeitos aditivos dos locos quantitativos, segundo a distribuição normal. As freqüências gênicas iniciais foram iguais em ambos os sexos e utilizaram-se três efeitos fixos com 10, 4 e 6 níveis, respectivamente. Para cálculo da matriz de similaridade genética, por meio de marcadores moleculares, foram utilizados 100 marcadores do tipo microssatélite (Simple Sequence Repeat - SSR).
A partir do genoma citado anteriormente, foi simulada uma população-base de 1000 indivíduos (500 machos e 500 fêmeas). A partir dessa população foi construída uma população inicial, pela escolha ao acaso e acasalamento de 10 machos e 100 fêmeas, produzindo cinco descendentes por acasalamento, gerando 500 indivíduos. Em seguida, foram formadas as populações de seleção num total de seis, correspondendo à combinação de dois tamanhos efetivos de população (18,18 e 66,66) e três sistemas de acasalamento praticados entre os reprodutores selecionados para pais da geração seguinte.
A seleção foi praticada durante 20 gerações consecutivas utilizando-se seleção individual (SI) e valores genéticos preditos pelo BLUP (BLUP clássico) e pelo BLUP marcadores (BLUPM), em que se utilizou uma matriz de similaridade genética calculada por meio de marcadores moleculares (Carneiro et al., 2006a). Em todos os métodos foram consideradas 1, 10 e 30 repetições do processo de simulação (R1, R10 e R30). Assim, denominando cada geração de seleção como um evento, no caso de uma repetição, o evento foi simulado uma única vez, e as gerações foram avançadas consecutivamente até um total de 20 para obtenção dos valores fenotípicos. Considerando 10 e 30 repetições, o evento foi simulado por 10 e 30 vezes, respectivamente, de modo que os resultados obtidos por evento, ao término das simulações, eram valores fenotípicos médios; e da mesma forma, foram avançadas 20 gerações.
Para as populações com tamanho efetivo 1 (TE1 = 18,18), em cada geração, foram selecionados cinco machos e 50 fêmeas e gerados cinco descendentes por acasalamento, totalizando 250 indivíduos. Para as populações com tamanho efetivo 2 (TE2 = 66,66) foram selecionados, a cada geração, 20 machos e 100 fêmeas e gerados cinco descendentes por acasalamento, totalizando 500 indivíduos. Para avaliar o efeito da oscilação genética nas diferentes estruturas de populações sob seleção utilizou-se o valor fenotípico médio por geração nos diferentes tamanhos efetivos (TE1 e TE2), números de repetições (uma, 10 e 30) e sistemas de acasalamento dos reprodutores selecionados (RAA = reprodutores acasalados aleatoriamente, EIC = exclusão de irmãos completos, EICMI = exclusão de irmãos completos e meio-irmãos).
RESULTADOS E DISCUSSÃO
Vários autores, ao compararem o BLUP e a SI, com dados simulados ou reais, observaram, nas gerações iniciais, superioridade do BLUP em relação à seleção individual, principalmente para características de baixa herdabilidade (Mabry et al., 1987; Belonsky e Kennedy, 1988; Euclydes e Guimarães, 1997; Carneiro et al., 1999). Carneiro et al. (2006a) avaliaram a seleção tradicional e a seleção associada a marcadores moleculares e observaram que o uso do BLUPM levou a maior ganho nas gerações iniciais (até cinco gerações) que o BLUP clássico e a seleção individual. No entanto, quando se consideraram os ganhos ao longo de 20 gerações de seleção, o BLUP clássico foi superior ao BLUPM, e este superior à SI.
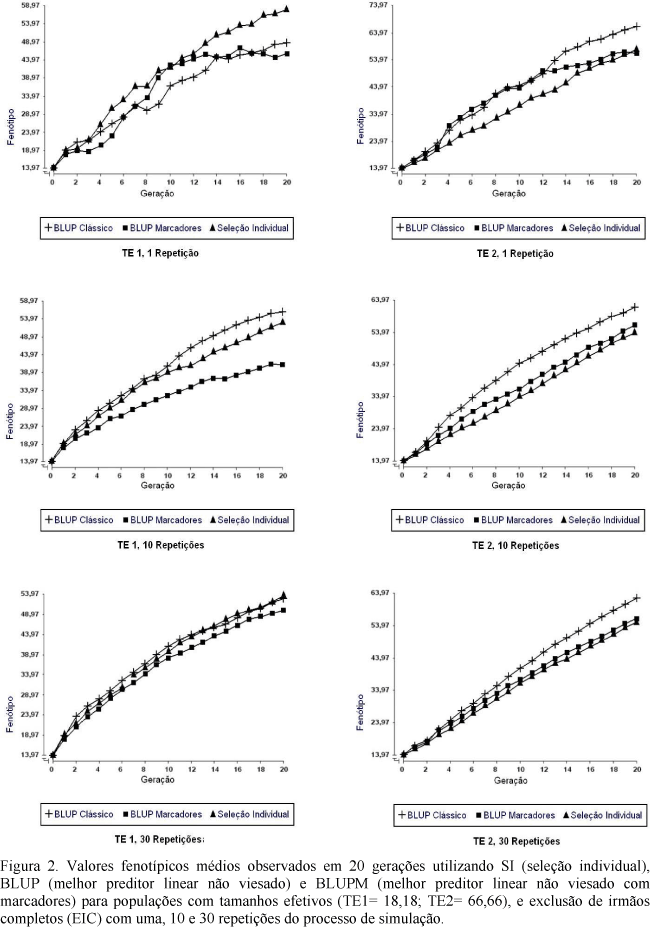
Neste estudo, a eficiência das metodologias de avaliação genética foi afetada pelo número de repetições do processo de simulação dos dados. Para as situações com apenas uma repetição do processo de simulação (Fig. 1, 2 e 3), os resultados apresentaram-se incoerentes, independentemente do tamanho efetivo (TE1 ou TE2) ou do sistema de acasalamento (RAA, EIC ou EICMI). Como exemplos, na seleção em que os reprodutores são acasalados aleatoriamente (Fig. 1), com uma repetição e TE1, na qual a SI teve desempenho semelhante ao BLUP e maior que o BLUPM, e com uma repetição e TE2, na qual a SI foi bem menor que o BLUP. Considerando-se acasalamentos com exclusão de irmãos completos (Fig. 2), com uma repetição e TE1, observou-se que a SI superou tanto o BLUP quanto o BLUPM. Essas incoerências, na comparação das metodologias são realçadas nas populações com menor tamanho efetivo, indicando que, em estudos de comparação de metodologias, devem-se utilizar populações com tamanhos efetivos maiores e média de várias repetições, a fim de minimizar os efeitos da oscilação genética.
Para as populações com TE1 e valores fenotípicos médios de 10 e 30 repetições do processo de simulação (Fig. 1, 2 e 3), houve leve tendência de o BLUP superar a SI, mostrando o grande poder desse método mesmo em pequenas populações. Verificou-se que a SI e o BLUPM não apresentaram distinção relevante entre si, que pudesse ser atribuída ao processo de oscilação genética, e servisse como indicativo de que a seleção com base nos valores genéticos preditos pelo BLUPM conduz a maiores problemas de oscilação genética em pequenas populações, em função do aumento nas taxas de endogamia e de fixação de alelos com redução do limite de seleção.
Por outro lado, observaram-se para as populações com TE2 e valores fenotípicos médios de 10 e 30 repetições (Fig. 1, 2 e 3), diferenças claras com relação aos métodos de seleção, principalmente para as populações com médias de 10 repetições. De modo geral, a seleção baseada nos valores genéticos preditos pelo BLUP superou a dos valores genéticos preditos pelo BLUPM e este método, por sua vez, a SI, nos diferentes sistemas de acasalamento, confirmando os resultados obtidos por Carneiro et al. (2006a). Estes indicam que, para dados de simulação, o uso de 10 ou mais repetições por geração, na comparação de metodologias, já seria aceitável, pois permitiria evidenciar diferenças entre os métodos de seleção.
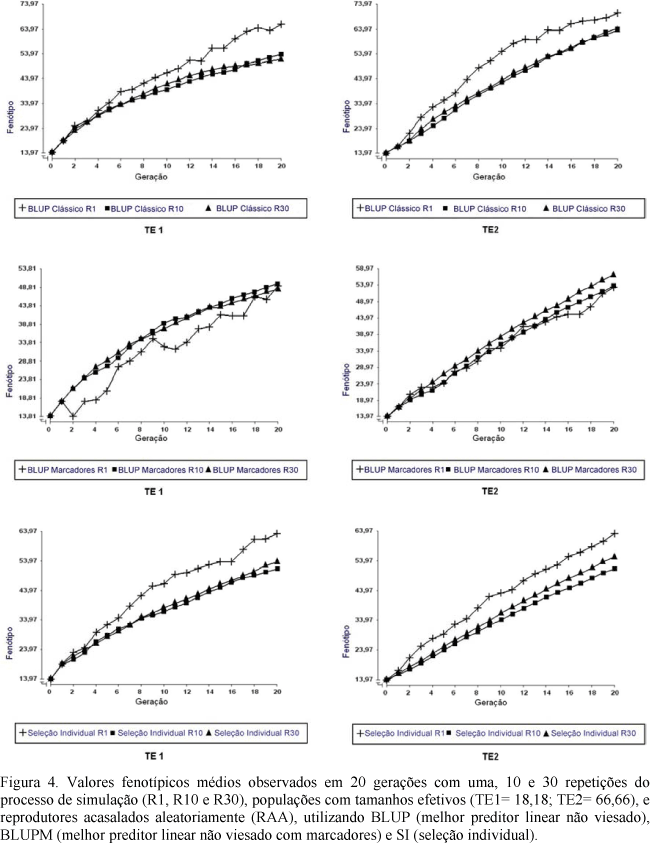
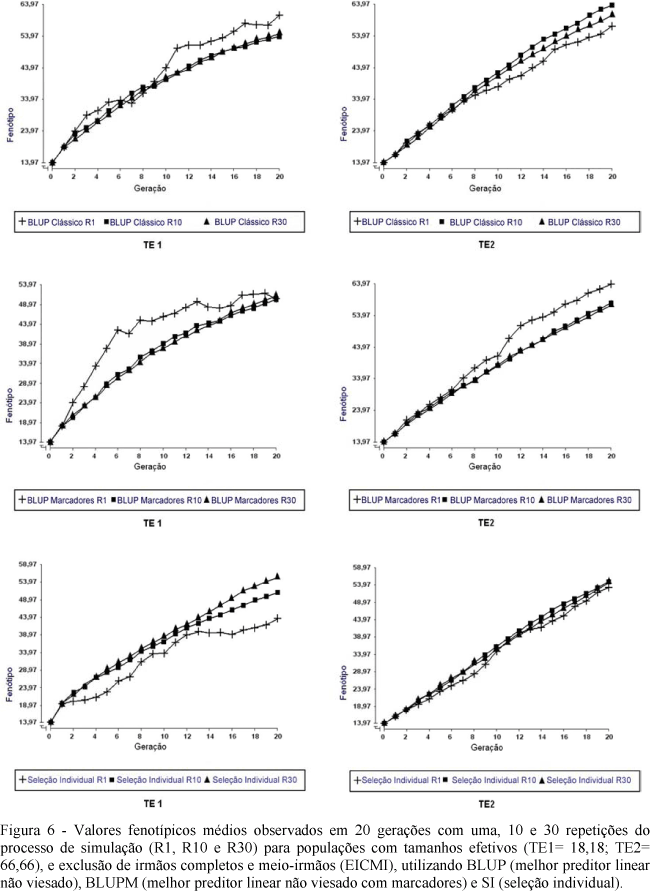
De modo geral, houve aumento nos valores fenotípicos, no decorrer das gerações. Entretanto, nas populações em que se trabalhou com os valores fenotípicos de uma única repetição do processo de simulação (R1) estes se mostraram bastante irregulares em todos os métodos de seleção, principalmente para o TE1 (Fig. 4, 5 e 6), apresentando estabilizações e quedas em algumas gerações, como por exemplo na Fig. 4 (BLUPM), Fig. 5 (BLUP) e Fig. 6 (BLUP e BLUPM).
Para as populações em que se trabalhou com os valores fenotípicos médios de 10 e 30 repetições do processo de simulação (R10 e R30), observou-se comportamento bastante regular, com ganhos crescentes e sem estabilizações e quedas. Esse é o comportamento esperado, visto que, na seleção artificial, ao se escolherem os melhores indivíduos para determinada característica, obtêm-se ganhos cumulativos ao longo das gerações. Resultados semelhantes foram encontrados em diversos trabalhos de simulação (Euclydes e Guimarães, 1997; Carneiro et al., 1999; Carneiro et al., 2001; Fonseca et al., 2001; Corrêa, 2001; Cunha et al., 2004).
É interessante observar que nas populações simuladas com média de 10 e 30 repetições do processo de simulação o comportamento foi bastante semelhante, principalmente quando se usou a SI, método que menos sofreu os efeitos da oscilação genética, por não apresentar perdas tão intensas pela fixação de alelos desfavoráveis e conduzir a taxas menores de endogamia. Esses resultados ressaltam a importância de se trabalhar com grande número de repetições do processo de simulação, em trabalhos com dados simulados, para comparações de metodologias de seleção. Nos casos em que o número de repetições for pequeno, alguma diferença causada por oscilação genética pode ser atribuída a diferenças entre as metodologias avaliadas, culminando em conclusões equivocadas. Carneiro et al. (2006b) afirmaram que em programas de melhoramento que utilizam pequenas populações, os resultados podem ser influenciados pela oscilação genética, podendo ocasionar grandes variações nos ganhos genéticos e que a seleção feita com base nos valores genéticos preditos pelo BLUP e BLUPM apresentou maior oscilação genética.
Para as populações com TE2 também foi observada grande diferença quando se compararam os valores fenotípicos obtidos de uma única repetição (R1) com os valores fenotípicos médios obtidos a partir de 10 e 30 repetições (R10 e R30) (Fig. 4, 5 e 6). Entretanto, o comportamento não foi tão irregular quanto nas populações de TE1, mostrando que, em populações com tamanhos efetivos maiores, os efeitos da oscilação genética não são tão intensos. Este fato foi também observado em trabalho de simulação desenvolvido por Verrier et al. (1993).
Com relação aos sistemas de acasalamento dos reprodutores selecionados (RAA, EIC e EICMI) (Fig. 4, 5 e 6), as diferenças com relação à oscilação genética não mantiveram tendência clara. Parece que os tipos de acasalamento que excluem acasalamentos entre irmãos (EIC e EICMI) com 10 e 30 repetições, principalmente para o BLUPM, permitiram maior controle da oscilação genética.
CONCLUSÕES
Em processos de simulação com apenas uma repetição, a oscilação genética influencia o ganho genético, principalmente em populações com pequeno tamanho efetivo, comprometendo a comparação de metodologias de avaliação genética. A utilização de 30 repetições do processo de simulação permite obter resultados consistentes em estudos comparativos de metodologias de avaliação genética utilizando simulação.
Recebido em 13 de setembro de 2007
Aceito em 14 de abril de 2008
Apoio: CNPq e PAGAB
E-mail: plscarneiro@gmail.com
- BELONSKY, G.M.; KENNEDY, B.W. Selection on individual phenotype and best linear unbiased predictor of breeding value in a closed swine herd. J. Anim. Sci, v.66, p.1124-1131, 1988.
- CARNEIRO, A.P.S.; TORRES, R.A.; EUCLYDES, R.F. et al. Efeito da Conexidade de Dados sobre a Acurácia dos Testes de Progênie e Performance. Rev. Bras. Zootec, v.30, p.342-347, 2001.
- CARNEIRO, P.L.S.; EUCLYDES, R.F.; SILVA, M.A. et al. Efeito de erros de pedigree na seleção. Rev. Bras. Zootec, v.28, p.269-274, 1999.
- CARNEIRO, P.L.S.; MALHADO, C.H.M.M.; EUCLYDES, R.F. et al. Seleção tradicional e associada a marcadores moleculares na avaliação genética animal. Pesq. Agropec. Bras, v.41, p.615-621, 2006a.
- CARNEIRO, P.L.S.; MALHADO, C.H.M.M.; EUCLYDES, R.F. et al. Oscilação genética em populações submetidas a métodos de seleção tradicionais e associados a marcadores moleculares. Rev. Bras. Zootec., v.35, p.84-91, 2006b.
- CORRÊA, F.J.C. Avaliação de métodos de seleção tradicionais, assistida por marcadores moleculares e por genes candidatos, com dados simulados 2001. 54f. Dissertação (Mestrado) - Universidade Federal de Viçosa, Viçosa, MG.
- CUNHA, E.E.; EUCLYDES, R.F.; TORRES, R.A. et al. Efeitos da seleção individual e da seleção baseada no BLUP em populações diferentes, quanto ao tipo de acasalamento. Arq. Bras. Med. Vet. Zootec, v.56, p.94-106, 2004.
- CUNHA, E.E.; EUCLYDES, R.F.; TORRES, R.A. et al. Simulação de dados para avaliação genética de rebanhos de gado de corte. Arq. Bras. Med. Vet. Zootec, v.58, p.381-387, 2006.
- EUCLYDES, R.F.; GUIMARÃES, S.E.F. Associação dos métodos tradicionais de seleção à seleção assistida por marcadores moleculares. Rev. Bras. Reprod. Anim, v.21, p.89-96, 1997.
- FALCONER, D.S.; MACKAY, T.F.C. Introduction to quantitative genetics 4.ed. London: Longman, 1996.
- FONSECA, R.; EUCLYDES, R.F.; TORRES, R.A. et al. Efeito da violação de pressuposições da metodologia de modelos mistos na avaliação genética animal. Arq. Bras. Med. Vet. Zootec., v.53, p.122-129, 2001.
- MABRY, J.W.; BENYSHEK, L.L.; JOHNSON, M.H. et al. A comparison of methods for ranking boars from different central test station. J. Anim. Sci, v.65, p.56-63, 1987.
- MEUWISSEN, T.H.E.; WOOLLIAMS, J.A. Effective sizes of livestock populations to prevent a decline in fitness. Theor. Appl. Genet, v.89, p.1019-1026, 1994.
- MUIR, W.M. The interaction of selection intensity, inbreeding depression, and randon genetic drift on short and long-term response to selection: Results using finite locus and finite population size models incorporating directional dominance. Proceeding of the American society of Animal Science, 2000. Disponível em: <http://www.google.com.br/search?hl=pt&ie=UTF8&oe=UTF8&q=blup+and+genetic+drift&lr >. Acessado em: 27 abr 2006.
- VERRIER, E.; COLLEAU, J.J., FOULLEY, J.L. Long-term effects of seletion based on the animal model BLUP in a finite population. Theor. Appl. Genet, v.87, p.446-454, 1993.
- WEI, M., CABALERO, A.; HILL, W.G. Selection response in finite populations. Genetics, v.144, p.1961-1974, 1996.
Datas de Publicação
-
Publicação nesta coleção
15 Ago 2008 -
Data do Fascículo
Ago 2008
Histórico
-
Aceito
14 Abr 2008 -
Recebido
13 Set 2007