Resumos
Avaliou-se o efeito do sistema seminatural na diversidade genética de um estoque de Brycon orbignyanus, utilizado em programas de repovoamento, com o marcador molecular RAPD. Vinte e quatro reprodutores, 12 machos e 12 fêmeas e 95 larvas da progênie foram analisados. Os nove primers utilizados produziram 90 fragmentos, dos quais 94,4% foram polimórficos. Houve diferença significativa na frequência de 20 dos 90 fragmentos entre os reprodutores e sua progênie sem a presença de fragmentos exclusivos. O índice de diversidade genética de Shannon, a porcentagem de fragmentos polimórficos e a diversidade genética de Nei foram mais altos nos indivíduos da progênie. A similaridade genética foi maior nos indivíduos do estoque de reprodutores. A análise de variância molecular mostrou que a maior parte da variação está dentro de cada grupo (89,1%) e não entre os grupos (10,9%). A identidade e a distância genética entre os estoques foram de 0,944 e 0,057, respectivamente. Assim, a utilização do sistema seminatural evitou a mortalidade de reprodutores B. orbignyanus e conservou a variabilidade genética da progênie.
piracanjuba; programa de repovoamento; RAPD; similaridade genética; variabilidade genética
The effect of the semi-natural system on the genetic diversity of a Brycon orbignyanus stock, used in stock enhancement programs, was evaluated with the RAPD molecular marker. Twenty-four broodstocks - 12 males and 12 females - and 95 larvae of the offspring were analyzed. The nine used primers produced 90 fragments, of which 94.4% were polymorphic. There was significant difference in the frequency of 20 out of the 90 fragments between the broodstocks and their offspring without the presence of exclusive fragments. The Shannon genetic diversity index, the percentage of polymorphic fragments and the Nei gene diversity were higher in the offspring individuals. Genetic similarity was higher in broodstock individuals. The analysis of molecular variance results showed that the major part of the genetic variation is within the groups (89.1%) and not between them (10.9%). The identity and genetic distance between the groups were 0.944 and 0.057, respectively. Like this, the use of the semi-natural system avoided the mortality of B. orbignyanus broodstocks and conserved the genetic variability of the offspring.
piracanjuba; genetic similarity; genetic variability; RAPD; stock enhancement programs
ZOOTECNIA E TECNOLOGIA E INSPEÇÃO DE PRODUTOS DE ORIGEM ANIMAL
Caracterização genética de Brycon orbignyanus utilizando o sistema seminatural
Genetic characterization of Brycon orbignyanus using the semi-natural system
N.M. Lopera-BarreroI; L. VargasII; R.N. SirolIII; R.P. RibeiroII; J.A. PovhIV; C.A. MangolinV
INúcleo de Pesquisa PeixeGen - UEM - Maringá, PR
IIDepartamento de Zootecnia - UEM - Maringá, PR
IIIDuke Energy International - Geração Paranapanema - São Paulo, SP
IVInstituto de Ciências Agrárias e Tecnológicas - UFMT - Rondonópolis, MT
VDepartamento de Biologia Celular e Genética - UEM - Maringá, PR
RESUMO
Avaliou-se o efeito do sistema seminatural na diversidade genética de um estoque de Brycon orbignyanus, utilizado em programas de repovoamento, com o marcador molecular RAPD. Vinte e quatro reprodutores, 12 machos e 12 fêmeas e 95 larvas da progênie foram analisados. Os nove primers utilizados produziram 90 fragmentos, dos quais 94,4% foram polimórficos. Houve diferença significativa na frequência de 20 dos 90 fragmentos entre os reprodutores e sua progênie sem a presença de fragmentos exclusivos. O índice de diversidade genética de Shannon, a porcentagem de fragmentos polimórficos e a diversidade genética de Nei foram mais altos nos indivíduos da progênie. A similaridade genética foi maior nos indivíduos do estoque de reprodutores. A análise de variância molecular mostrou que a maior parte da variação está dentro de cada grupo (89,1%) e não entre os grupos (10,9%). A identidade e a distância genética entre os estoques foram de 0,944 e 0,057, respectivamente. Assim, a utilização do sistema seminatural evitou a mortalidade de reprodutores B. orbignyanus e conservou a variabilidade genética da progênie.
Palavras-chave: piracanjuba, programa de repovoamento, RAPD, similaridade genética, variabilidade genética
ABSTRACT
The effect of the semi-natural system on the genetic diversity of a Brycon orbignyanus stock, used in stock enhancement programs, was evaluated with the RAPD molecular marker. Twenty-four broodstocks - 12 males and 12 females - and 95 larvae of the offspring were analyzed. The nine used primers produced 90 fragments, of which 94.4% were polymorphic. There was significant difference in the frequency of 20 out of the 90 fragments between the broodstocks and their offspring without the presence of exclusive fragments. The Shannon genetic diversity index, the percentage of polymorphic fragments and the Nei gene diversity were higher in the offspring individuals. Genetic similarity was higher in broodstock individuals. The analysis of molecular variance results showed that the major part of the genetic variation is within the groups (89.1%) and not between them (10.9%). The identity and genetic distance between the groups were 0.944 and 0.057, respectively. Like this, the use of the semi-natural system avoided the mortality of B. orbignyanus broodstocks and conserved the genetic variability of the offspring.
Keywords: piracanjuba, genetic similarity, genetic variability, RAPD, stock enhancement programs
INTRODUÇÃO
Brycon orbignyanus (Valenciennes, 1849), conhecido no Brasil como piracanjuba ou bracanjuba (ordem Characiformes, família Characidae, subfamília Bryconinae), é uma espécie migratória nativa das bacias formadas pelos rios Uruguai (Zaniboni-Filho e Schulz, 2003) e Paraná (Ganeco et al., 2001). Em resposta às modificações ambientais geradas por mudanças climáticas e principalmente por ações humanas, nos últimos anos populações naturais desse peixe desapareceram (Zaniboni-Filho et al., 2006), sendo catalogada como espécie em risco de extinção. Por esse motivo, programas que visem à sua conservação estão sendo desenvolvidos em várias regiões do Brasil, destacando-se o repovoamento como estratégia utilizada para atingir esse objetivo (Lopera-Barrero et al., 2007).
Os programas de repovoamento têm destaque por serem estratégias de conservação viáveis para a maioria dos piscicultores (Sirol e Britto, 2006), porém, é necessário um respaldo científico, em áreas como a da genética e a da reprodução que permitam determinar objetivamente quais sistemas reprodutivos, técnicas e estratégias devem ser usados ou recomendados (Lopera-Barrero et al., 2007).
As análises genéticas de estoques de pisciculturas representam informações de grande importância para conseguir ganhos expressivos na produção e na conservação de peixes (Povh et al., 2008). A perda da variabilidade genética dos estoques nas pisciculturas utilizados em programas de repovoamento, em razão do inadequado manejo genético, descrito por Frost et al. (2006) como um problema que leva à endogamia, diminui a adaptabilidade e a sobrevivência das progênies que serão liberadas (Povh, 2007). Isso, consequentemente, pode afetar a população natural de peixes (Sønstebø et al., 2007) e o ecossistema em geral, podendo até mesmo resultar em extinção da espécie (Oliveira et al., 2002). Para analisar a variabilidade genética de estoques de reprodutores e progênies, o marcador molecular Random Amplified Polymorphic DNA (RAPD) tem se mostrado efetivo e vem sendo utilizado com sucesso (Lopera-Barrero et al., 2008b).
Em conjunto com as análises genéticas, deve-se realizar o melhoramento dos processos reprodutivos, pois o manejo errado, resultado de acasalamentos com número insuficiente de reprodutores (Aho et al., 2006; Melo et al., 2006) ou que utiliza sistemas reprodutivos de forma inadequada, pode resultar em perda da variabilidade genética. Alguns trabalhos mostraram que existe menor mortalidade, maior fertilização e melhor conservação da variabilidade genética quando se utiliza o sistema reprodutivo seminatural, em comparação com o sistema por extrusão (Reynalte-Tataje et al., 2002; Povh, 2007). Isso demonstra que, possivelmente, o primeiro sistema reprodutivo pode ser o mais indicado em programas de repovoamento e para peixes susceptíveis ao estresse por manejo e manipulação, como é o caso de B. orbignyanus.
O objetivo deste estudo foi avaliar o efeito do sistema seminatural na diversidade genética de um estoque de B. orbignyanus utilizado em programas de repovoamento do rio Paranapanema, com o marcador molecular RAPD.
MATERIAL E MÉTODOS
Foram utilizados 24 reprodutores, 12 machos e 12 fêmeas de B. orbignyanus, selecionados de um estoque de 136 indivíduos, estocados há seis anos nas instalações da Estação de Aquicultura e Hidrologia da Duke Energy International (Geração Paranapanema), localizada na cidade de Salto Grande, SP. Esses indivíduos são originários da primeira geração de um estoque pertencente a uma piscicultura localizada na cidade de Castilho, SP (Fig. 1).
O experimento foi realizado em novembro de 2006 nas instalações da Estação de Aquicultura da Duke Energy International. Os 24 reprodutores foram conduzidos para o laboratório e induzidos à reprodução com extrato de hipófise de carpa. As fêmeas receberam 5,5mg/kg, divididos em duas aplicações: 10% do total na primeira aplicação e 12 horas depois os 90% restantes. Os machos receberam 2,5mg/kg em dose única.
Os reprodutores foram colocados em um tanque circular com raio de 5,1m e 1,85m de profundidade média, abastecido por fluxo de água contínuo (131L/s) em dois sentidos de vazão. O escoamento da água na porção central permitiu o direcionamento dos ovos para uma incubadora cilindro/cônica de captação de 200L com fluxo contínuo de água (7L/s).
Aproximadamente seis horas depois da última indução (160 horas-grau, 27ºC) iniciou-se a coleta de ovos. Estabeleceu-se um período de coleta de no máximo seis horas, com a retirada dos ovos da incubadora de captação a cada hora, seguida de transferência para incubadoras do tipo cilindro/cônicas, onde ocorreu a incubação. Depois de seis horas, verificou-se, por pressão abdominal, se os machos e fêmeas utilizados desovaram completamente - a falta de saída indicava que ocorreu vazamento total dos gametas. Paralelamente, dos indivíduos foram coletadas amostras de nadadeira caudal (0,5cm2 aproximadamente), que foram armazenadas em microtubos de 1,5mL contendo álcool etílico 100% para a posterior extração e amplificação do DNA. A porcentagem de mortalidade dos reprodutores usados nos acasalamentos foi definida um dia depois da reprodução.
Três dias depois da eclosão dos ovos, aproximadamente 200 larvas, coletadas de forma aleatória das incubadoras em todos os horários, foram armazenadas em microtubos de 1,5mL contendo álcool etílico 100%. Dessas larvas, 95 foram coletadas aleatoriamente para posterior extração e amplificação do DNA.
Para extração de DNA, foi utilizada a metodologia descrita por Lopera-Barrero et al. (2008a). Em microtubos contendo separadamente as larvas e as nadadeiras, foram adicionados 550µL de tampão de lise (50mM Tris-HCl, 50mM EDTA, 100mM NaCl, 1% SDS) e 7µL de proteinase K (200µg/mL). As amostras foram incubadas em banho-maria a 50ºC por 12 horas. O DNA foi precipitado com 600µL de solução de NaCl (5M) e centrifugado por 10min a 12000rpm. O sobrenadante contendo o DNA foi transferido para novos microtubos, precipitado com 700µL de álcool etílico absoluto e incubado por uma hora a -20ºC. O DNA foi centrifugado, lavado com 700µL de álcool etílico 70%, ressuspendido em tampão TE - 10mM Tris pH 8,0 e 1mM EDTA (80µL para nadadeira e 35µL para larva), e tratado com 7µL de RNAse (30µg/mL) em banho-maria a 37ºC por uma hora e, em seguida, estocado em freezer a -20ºC.
O DNA foi quantificado em espectrofotômetro Shimadzu com absorvância de 260nm. As amostras foram diluídas para a concentração de 10ng/µL (reprodutores) e de 5ng/µL (larvas). Para conferir a qualidade do DNA, foi realizada eletroforese em gel de agarose 1%, conduzida em tampão TBE 1X (500mM Tris-HC1, 60mM ácido bórico e 83mM EDTA), por uma hora, a 70V.
O DNA genômico foi amplificado para um volume de reação de 15µL, no qual se utilizou tampão 1X Tris-KCl, 2,5mM de MgCl2, 0,46µM de primer, 0,2mM de cada dNTP, uma unidade de Platinun Taq DNA Polimerase (Invitrogen, EUA) 10ng de DNA molde para os reprodutores e 5ng para as larvas. O DNA foi desnaturado a 94ºC por 4min e, em seguida, foram realizados 40 ciclos, cada um consistindo de: 1min de desnaturação a 94ºC, 90 segundos de anelamento do primer a 40ºC e 2min para extensão a 72ºC. Após realizou-se uma extensão final a 72ºC por 5min. As reações de RAPD foram realizadas em um termociclador Eppendorf Mastercycler Gradient.
Foi avaliada a amplificação de 60 primers de 10 bases dos kits OPA, OPX e OPW (Operon Technologies Ltd) e escolhidos os que apresentaram melhor definição e reprodutibilidade. Os produtos de amplificação foram separados em gel de agarose 1,5%. Foram utilizados 15µL do produto amplificado e 2µL de tampão de amostra (40% sacarose e 0,25% azul de bromofenol) em eletroforese horizontal. A eletroforese foi realizada em tampão TBE 0,5X (45mM Tris-Borato e 1mM EDTA) por quatro horas, a 70V. Os géis de quantificação e amplificação foram visualizados sob radiação UV, depois da coloração em banho de brometo de etídio (0,5µg/mL) por uma hora. A imagem foi fotografada utilizando o programa Kodak EDAS (Kodak 1D Image Analysis 3.5).
O tamanho dos fragmentos obtidos a partir das amplificações foi estimado por comparação com marcador de peso molecular de 100pb (Invitrogen, EUA). A presença ou ausência de fragmentos de tamanhos moleculares idênticos foi usada para a construção de matriz de similaridade com base no cálculo do coeficiente de Jaccard, codificando 1 como presença de fragmento e 0 como ausência. A similaridade genética em cada estoque foi obtida com base no cálculo do coeficiente de similaridade de Jaccard, por meio do programa NTSYS 1.7 (Rohlf, 1989). O índice de diversidade genética de Shannon e a diversidade genética (Nei, 1973) foram obtidos com o programa PopGene 1.31 (Yeh et al., 1999). O programa TFPGA 1.3 (Miller, 1997) foi utilizado para determinar a porcentagem de fragmentos polimórficos (critério de 95%), distância e identidade genética (Nei, 1978) entre os estoques e frequência dos fragmentos pelo teste exato (Raymond e Rousset, 1995). O programa Arlequin 3.0 (Excoffier et al., 2005) foi utilizado para a análise de variância molecular. A significância desse teste foi verificada pelo método de permutações aleatórias com 10000 permutações.
RESULTADOS E DISCUSSÃO
Dos 60 primers testados, nove foram escolhidos para amplificar o DNA genômico. Na Tab. 1, pode-se observar a sequência dos primers selecionados, a porcentagem de G+C, o número de fragmentos e o tamanho dos fragmentos amplificados para os reprodutores e a progênie de B. orbignyanus
A amplificação dos nove primers gerou 90 fragmentos, com tamanho entre 300 e 2200pb. O número total de fragmentos que foram selecionados para serem usados nas análises variou de oito (OPW03 e OPW08) a 14 (OPW02). O maior fragmento (2200pb) foi obtido pela amplificação do primer OPW02 e o menor (300pb) pela amplificação dos primers OPW08 e OPX01. Dos 90 fragmentos obtidos, 85 (94,4%) foram polimórficos.
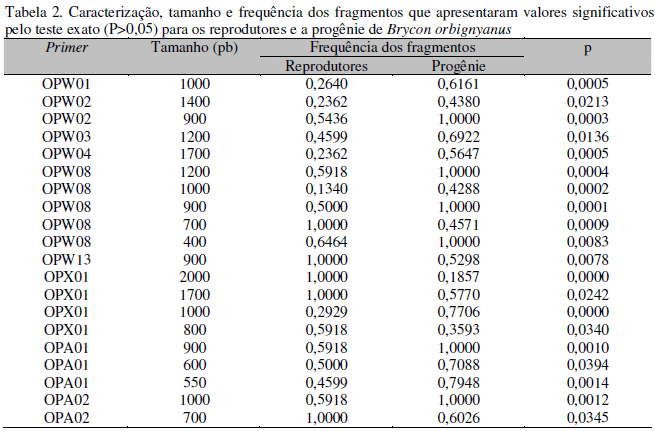
Houve diferença significativa na frequência de 20 dos 90 fragmentos entre os reprodutores e sua progênie (Tab. 2). A ausência de fragmentos exclusivos pode ser decorrente do tempo de estocagem dos reprodutores (seis anos) no qual não houve introdução de novos indivíduos ao estoque, e que se refletiu na progênie.
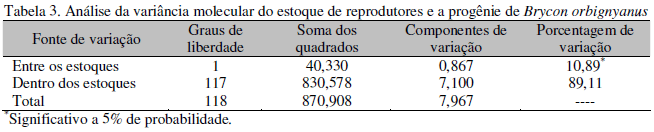
Os resultados de variabilidade genética estimados pelo índice de diversidade genética de Shannon (IS) e pela porcentagem de fragmentos polimórficos (%FP) mostraram alta variabilidade genética no estoque de reprodutores e aumento da variabilidade genética na progênie (Fig. 2). A maior similaridade genética (SG) dos reprodutores (0,838) em relação à progênie (0,828) e a diversidade genética de Nei (DGN = 0,169 e 0,208, respectivamente) confirmam a maior variabilidade genética dos indivíduos da progênie. De acordo com a análise de variância molecular, a maior parte da variação genética está dentro de cada estoque (89,1%) e não entre os estoques (10,9%) de B. orbignyanus (Tab. 3). Esses resultados são confirmados com as estimativas da identidade (0,944) e da distância genética (0,057).
Ao analisar a alta variabilidade genética do estoque de reprodutores, é presumível supor que o efeito gargalo (bottleneck effect) não o influenciou, indicando que houve adequado manejo reprodutivo, em que a formação do estoque com suficiente variabilidade genética e a utilização de sistemas reprodutivos eficientes (Povh, 2007) possivelmente permitiram manter o pool genético do estoque de reprodutores e, consequentemente, da sua progênie. Isso é muito significativo quando se pensa em estoques utilizados em programas de repovoamento, pois o método mais utilizado na formação de novos estoques é a seleção de indivíduos com características visuais favoráveis, o que pode promover a perda de variabilidade genética devido à seleção intencional, apresentando problemas de endogamia pelo grande relacionamento parental (Kang et al., 2006; Moreira et al., 2007; Qin et al., 2007).
Por outro lado, é comum que, nos programas de repovoamento, os acasalamentos sejam realizados com poucos indivíduos - muitas vezes um só macho fertiliza várias fêmeas - pois as espécies migratórias são muito prolíficas e limitam, por falta de uma infra-estrutura adequada, a utilização de número elevado de indivíduos (Povh et al., 2008). No presente estudo, quando foram acasalados 24 reprodutores com alta variabilidade genética (%FP = 41,1%, IS = 0,258, DGN = 0,169), é provável que a contribuição reprodutiva na fertilização dos ovos fosse também alta (óvulos fecundados por diferentes machos), favorecida pela utilização do sistema seminatural, que resultou, dessa forma, em aumento da variabilidade genética na progênie. Apesar de não ter dados de paternidade que confirmem essa afirmação, é factível que a maioria dos reprodutores tenha participado do processo reprodutivo, pois a avaliação física (pressão abdominal), realizada após o cruzamento, mostrou que todas as fêmeas e todos os machos não apresentaram quantidades significativas de gametas, ou seja, houve desova/espermiação dos reprodutores durante o processo reprodutivo.
O sistema reprodutivo seminatural pode ter influenciado também a variabilidade genética da progênie, pois esse sistema reduz a seleção não intencional no processo reprodutivo que acontece normalmente no sistema reprodutivo por extrusão (Povh, 2007) e diminui significativamente a mortalidade causada pelo estresse. Isso possibilita que maior número de reprodutores se reproduza durante os acasalamentos e que exista sincronização na liberação dos gametas (Reynalte-Tataje et al., 2002), permitindo que o pool genético de um estoque pequeno, ou de um acasalamento com poucos indivíduos, seja representado com maior heterogeneidade na progênie (Cacho et al., 2007), devido ao menor efeito seletivo do sistema e à maior taxa de sobrevivência dos reprodutores, o que pode viabilizar a manutenção da variabilidade genética na progênie. No presente estudo, mediante a utilização do sistema reprodutivo seminatural, não houve mortalidade dos reprodutores utilizados (verificado um dia depois do processo reprodutivo) e aconteceu a preservação da variabilidade genética na progênie, demonstrando a efetividade desse sistema reprodutivo. Povh (2007), ao estudar o sistema de reprodução seminatural e por extrusão no acasalamento de 20 reprodutores - 10 machos e 10 fêmeas - de Piaractus mesopotamicus, verificou maior variabilidade genética da progênie no sistema seminatural (IS = 0,415 e %FP = 72,4%) quando comparada com o sistema por extrusão (IS = 0,231 e %FP = 42,7%) e maior mortalidade no sistema por extrusão. Reynalte-Tataje et al. (2002), ao trabalharem na reprodução de Leporinus macrocephalus, não encontraram mortalidade de reprodutores ao utilizarem o sistema seminatural, diferentemente do sistema por extrusão, em que os resultados chegaram a 66,7%.
A partir dessas evidências e dos resultados de diversidade genética dos reprodutores e da progênie de B. orbignyanus, pode-se sugerir que o sistema reprodutivo seminatural é indicado para a reprodução dessa espécie em ambientes controlados, pois diminuem a mortalidade dos reprodutores durante o processo reprodutivo e permitem a manutenção da variabilidade genética. Estes resultados assemelham-se aos de Povh (2007). Segundo esse autor, se o objetivo da piscicultura é a produção de juvenis para programas de repovoamento, deve-se preconizar o sistema reprodutivo seminatural. Por isso, pode-se sugerir que, durante a formação de grupos de acasalamentos destinados à produção, formação de novos estoques ou programas de repovoamento de B. orbignyanus, é necessário utilizar a maior quantidade de reprodutores possível - neste trabalho, 24 reprodutores permitiram conservar a variabilidade genética - durante todo o período reprodutivo, o que, segundo Moreira (2001) e Frost et al. (2006), permitiria obter alta variação e contribuição reprodutiva entre os indivíduos e suas progênies. Essa recomendação confirma o que os resultados obtidos por Yokota et al. (2003) sugeriram, isto é, os autores constataram que o aumento do Ne de 20 (10 machos e 10 fêmeas) para 50 (25 machos e 25 fêmeas) proporcionou maior variabilidade genética na progênie.
A diminuição da variabilidade genética pode tornar um programa de repovoamento ineficiente, por baixa sobrevivência de juvenis no ambiente aquático, e proporcionar impactos genéticos irreversíveis na população natural (Sønstebø et al., 2007; Povh et al., 2008), que podem levar à extinção da espécie. Por esse motivo, os resultados observados neste estudo são de grande importância, pois a partir deles o manejo reprodutivo dos estoques de B. orbignyanus utilizados em programas de repovoamento pode ser orientado corretamente e com maior objetividade. O marcador RAPD permitiu determinar com sucesso a variabilidade genética presente nos reprodutores e nas progênies após o acasalamento ao utilizar o sistema seminatural. Pelo caráter dominante do marcador, não foi possível determinar quantos reprodutores realmente contribuíram com a formação da progênie nos dois sistemas de acasalamento. Contudo, a ausência de mortalidade e a presença de desova/espermiação observada nos reprodutores podem fundamentar que possivelmente todos os machos ou a maioria deles participaram na reprodução durante o acasalamento.
CONCLUSÕES
Nos resultados encontrados neste estudo, ficou evidenciado como a utilização do sistema seminatural evitou a mortalidade de reprodutores e conservou a variabilidade genética durante a reprodução de B. orbignyanus, o que em progênies destinadas para programas de repovoamento pode significar um aumento da adaptabilidade e da sobrevivência ao ambiente.
Recebido em 8 de junho de 2009
Aceito em 2 de fevereiro de 2010
E-mail: nelson.peixegen@gmail.com
- AHO, T.; RÖNN, J.; PIIRONEN, J. et al. Impacts of effective population size on genetic diversity in hatchery reared Brown trout (Salmo trutta L.) populations. Aquaculture, v.253, p.244-248, 2006.
- CACHO, M.S.R.F.; YAMAMOTO, M.E.; CHELLAPPA, S. Mating system of the amazonian cichlid angel fish, Pterophyllum scalare. Braz. J. Biol, v.67, p.161-165, 2007.
- EXCOFFIER, L.; LAVAL, G.; SCHNEIDER, S. Arlequin Ver. 3.0: an integrated software package for population genetics data analysis. Evol. Bioinform. Online, v.1, p.47-50, 2005.
- FROST, L.A.; EVANS, B.S.; JERRY, D.R. Loss of genetic diversity due to hatchery culture practices in barramundi (Lates calcarifer). Aquaculture, v.261, p.1056-1064, 2006.
- GANECO, L.; NAKAGHI, L.; URBINATI, E. et al. Análise morfológica do desenvolvimento ovocitário de piracanjuba (Brycon orbignyanus) durante o ciclo reprodutivo. Bol. Inst. Pesca, v.27, p.131-138, 2001.
- KANG, J.H.; NOH, J.K.; KIM, J.H. et al. Genetic relationship between broodstocks of olive flounder, Paralichthys olivaceus (Temminck and Schlegel) using microsatellite markers. Aquacult. Res, v.37, p.701-707, 2006.
- LOPERA-BARRERO, N.M.; POVH, J.A.; RIBEIRO, R.P. et al. Comparación de protocolos de extracción de ADN con muestras de aleta y larva de peces: extracción modificada con sal (NaCl). Cienc. Invest. Agr, v.35, p.15-24, 2008a.
- LOPERA BARRERO, N.M.; RIBEIRO, R.P.; POVH, J.A. et al. O repovoamento de peixes: uma estratégia multidisciplinar?. Aquicult. Pesca, v.30, p.71-74, 2007.
- LOPERA-BARRERO, N.M.; RIBEIRO, R.P.; SIROL, R.N. et al. Variabilidad genética de lotes de Brycon orbignyanus utilizados en programas de repoblamiento: manejo y conservación. Acta Biol. Col., v.13, p.107-118, 2008b.
- MELO, D.C.; OLIVEIRA, D.A.A.; RIBEIRO, L.P. et al. Caracterização genética de seis plantéis comerciais de tilápia (Oreochromis) utilizando marcadores microssatélites. Arq. Bras. Med. Vet. Zootec, v.58, p.87-93, 2006.
- MILLER, M.P. Tools for population genetic analysis (TFPGA) 1.3: a Windows program for the analysis of allozyme and molecular population genetic data. Logan: Utah State University, 1997. Disponível em: <http://www.marksgeneticsoftware.net/tfpga.htm>. Acessado em: 27 out. 2007.
- MOREIRA, A.A.; HILSDORF, A.W.S.; SILVA, J.V. et al. Variabilidade genética de duas variedades de tilápia nilótica por meio de marcadores microssatélites. Pesq. Agropec. Bras, v.42, p.521-526, 2007.
- MOREIRA, H.L.M. Genética e melhoramento de peixes. In: MOREIRA, H.L.M.; VARGAS, L.; RIBEIRO, R.P.; ZIMMERMANN, S. (Ed.). Fundamentos da moderna aquicultura Porto Alegre: ULBRA, 2001. p.135-147.
- NEI, M. Analysis of gene diversity in subdivided populations. Proc. Natl. Acad. Sci, v.70, p.3321-3323, 1973.
- NEI, M. Estimation of average heterozygosity and genetic distance from a small number of individual. Genetics, v.89, p.583-590, 1978.
- OLIVEIRA, A.V.; PRIOLI, A.J.; PRIOLI, S.M.A.P. et al. Diversity and genetic distance in populations of Steindachnerina in the Upper Paraná River floodplain. Genetica, v.115, p.259-267, 2002.
- POVH, J.A. Avaliação da diversidade genética e do manejo reprodutivo do pacu, Piaractus mesopotamicus 2007. 75f. Tese (Doutorado em Zootecnia) - Universidade Estadual de Maringá, Maringá, PR.
- POVH, J.A.; LOPERA BARRERO, N.M.; RIBEIRO, R.P. et al. Importancia del monitoreo genético de programas de repoblamiento de peces mediante marcadores moleculares. Cienc. Invest. Agr, v.35, p.25-35, 2008.
- QIN, Y.; LIU, X.; ZHANG, H. et al. Effect of parental stock size on F1 genetic structure in the bay scallop, Argopecten irradians (Lamarck, 1819). Aquacult. Res, v.38, p.174-181, 2007.
- RAYMOND, M.; ROUSSET, F. An exact test for population differentiation. Evolution, v.49, p.1280-1283, 1995.
- REYNALTE-TATAJE, D.A.; ESQUIVEL, B.M.; ESQUIVEL, J.R. et al. Reproducción inducida del piauçu, Leporinus macrocephalus Garavello y Britski, 1988 (Characiformes, Anostomidae). Bol. Inst. Pesca, v.28, p.11-18, 2002.
- ROHLF, F.J. NTSYS-PC: numerical taxonomy and multivariate analysis system. Setauket , NY: Exeter Publishers, 1989. 191p.
- SIROL, R.N.; BRITTO, S.G. Conservação e manejo da ictiofauna: repovoamento. In: NOGUEIRA, M.G.; HENRY, R.; JORCIN, A. (Ed.). Ecologia de reservatórios: impactos potenciais, ações de manejo e sistemas em cascatas São Carlos, SP: RiMA, 2006. p.275-284.
- SØNSTEBØ, J.H.; BORGSTRØM, R.; HEUN, M. Genetic structure of brown trout (Salmo trutta L.) from the Hardangervidda mountain plateau (Norway) analyzed by microsatellite DNA: a basis for conservation guidelines. Conserv. Genet, v.8, p.33-44, 2007.
- YEH, F.C.; BOYLE, T.Y.Z.; XIYAN, J.M. PopGene Version 131: Microsoft Window-based freeware for population genetic analysis. Edmonton, AB: University of Alberta and Center for International Forestry Research, 1999. 29p.
- YOKOTA, M.; HARADA, Y.; IIZUKA, M. Genetic drift in a hatchery and the maintenance of genetic diversity in hatchery-wild systems. Fish Sci, v.69, p.101-109, 2003.
- ZANIBONI-FILHO, E.; REYNALTE-TATAJE, D.; WEINGARTNER, M. Potencialidad del género Brycon en la piscicultura brasileña. Rev. Col. Cienc. Pec, v.19, p.233-240, 2006.
- ZANIBONI-FILHO, E.; SCHULZ, U.H. Migratory Fishes of the Uruguay River. In: CAROLSFELD, J.; HARVEY, B.; ROSS, C. et al. (Ed.). Migratory sishes of South America: Biology, fisheries and conservation status. Victoria, BC: World Fisheries Trust, 2003. p.253-271.
Datas de Publicação
-
Publicação nesta coleção
23 Mar 2010 -
Data do Fascículo
Fev 2010
Histórico
-
Aceito
02 Fev 2010 -
Recebido
08 Jun 2009