Resumos
A diversidade genética entre três linhagens de codorna (Coturnix japônica) foi avaliada utilizando-se a técnica de random amplified polymorphic DNA (RAPD). As linhagens selecionadas para produção de ovos foram identificadas como amarela, azul e vermelha por meio de anilhas no pé esquerdo. Seis primers de RAPD amplificaram 55 loci, os quais geraram padrão de bandas intensa e reproduzível em gel de agarose. Os resultados indicaram polimorfismos dentro e entre as linhagens. A similaridade de Jaccard média e o índice de diversidade Shannon revelaram alta diversidade dentro das linhagens de codornas. O teste de Mantel por meio do algoritmo unweighted pair-group method using arithmetic average (UPGMA) e a dispersão de coordenadas principais indicaram diferenciação genética significativa, embora em baixo nível. Os resultados sugerem que a diversidade genética dentro e entre as linhagens de codornas da Universidade Estadual de Maringá são promissoras para uso em programas de melhoramento.
codorna; Coturnix japonica; diversidade genética; RAPD
The genetic diversity among three lineages of quail (Coturnix japonica) was evaluated by the random amplified polymorphic DNA (RAPD) technique. The lineages were selected for egg production and identified with a yellow, blue, or red ring fasten on their left foot. Six selected RAPD primers amplified 55 loci, which generated intense and reproducible bands on agarose gel. The results indicated polymorphism within and among the lineages. The Jaccard similarity average and the Shannon diversity index revealed high diversity values within the quail lineages. The Mantel test, unweighted pair-group method using arithmetic average (UPGMA) algorithm and dispersion of principal coordinates indicated significant genetic differentiation, although at low levels. Overall, the results suggest that the genetic diversity within and among the quail lineags from the State Universidade Estadual de Maringá are promising for use in breeding programs.
quail; Coturnix japonica; genetic diversity; RAPD
ZOOTECNIA E TECNOLOGIA E INSPEÇÃO DE PRODUTOS DE ORIGEM ANIMAL
Diversidade genética entre três linhagens de codorna selecionadas para produção de ovos
Genetic diversity among three lineages of quail, selected for egg production
R.A. PrioliI; E. Gasparino1, * * Autor para correspondência ( corresponding author) E-mail: egasparino@uem.br ; M.A.M. SoaresII; D.S. MarquesI; D.V. BlanckI; S.M.A. PrioliIII
IDepartamento de Zootécnica - UEM Av. Colombo, 5790, 87020-900 - Maringá, PR
IIDepartamento de Genética - UFRRJ - Seropédica, RJ
IIIDepartamento de Biologia Celular e Genética - UEM - Maringá, PR
RESUMO
A diversidade genética entre três linhagens de codorna (Coturnix japônica) foi avaliada utilizando-se a técnica de random amplified polymorphic DNA (RAPD). As linhagens selecionadas para produção de ovos foram identificadas como amarela, azul e vermelha por meio de anilhas no pé esquerdo. Seis primers de RAPD amplificaram 55 loci, os quais geraram padrão de bandas intensa e reproduzível em gel de agarose. Os resultados indicaram polimorfismos dentro e entre as linhagens. A similaridade de Jaccard média e o índice de diversidade Shannon revelaram alta diversidade dentro das linhagens de codornas. O teste de Mantel por meio do algoritmo unweighted pair-group method using arithmetic average (UPGMA) e a dispersão de coordenadas principais indicaram diferenciação genética significativa, embora em baixo nível. Os resultados sugerem que a diversidade genética dentro e entre as linhagens de codornas da Universidade Estadual de Maringá são promissoras para uso em programas de melhoramento.
Palavras-chave: codorna, Coturnix japonica, diversidade genética, RAPD
ABSTRACT
The genetic diversity among three lineages of quail (Coturnix japonica) was evaluated by the random amplified polymorphic DNA (RAPD) technique. The lineages were selected for egg production and identified with a yellow, blue, or red ring fasten on their left foot. Six selected RAPD primers amplified 55 loci, which generated intense and reproducible bands on agarose gel. The results indicated polymorphism within and among the lineages. The Jaccard similarity average and the Shannon diversity index revealed high diversity values within the quail lineages. The Mantel test, unweighted pair-group method using arithmetic average (UPGMA) algorithm and dispersion of principal coordinates indicated significant genetic differentiation, although at low levels. Overall, the results suggest that the genetic diversity within and among the quail lineags from the State Universidade Estadual de Maringá are promising for use in breeding programs.
Keywords: quail, Coturnix japonica, genetic diversity, RAPD
INTRODUÇÃO
O crescimento da coturnicultura no Brasil vem despertando a atenção de pesquisadores da área, no sentido de desenvolver trabalhos que contribuam para o aprimoramento e a fixação dessa cultura como fonte rentável na produção avícola (Furlan et al., 1998). A coturnicultura brasileira é voltada principalmente para a produção de ovos, cujo produto é bem aceito pelos consumidores. A carne é considerada exótica, e o seu consumo ainda é pequeno devido ao preço alto para os padrões brasileiros.
Desde a sua implantação como atividade avícola econômica, a coturnicultura tem encontrado barreiras que por vezes inviabilizam a exploração econômica. Uma dessas barreiras é a falta de material genético que garanta o potencial de produção. No Brasil, os programas de melhoramento genético de codorna são ainda incipientes. A prática corrente tem sido a reprodução do material genético disponível que, pela deficiência de controle e de esquema de seleção, sofre problemas de depressão pela consanguinidade, resultando em redução de postura, queda de fertilidade e aumento de mortalidade.
As metodologias moleculares têm auxiliado na compreensão das relações evolutivas e de taxonomia de uma ampla diversidade de animais e plantas, bem como auxiliado no estudo de divergência genética (Milligan et al, 1994; Fritsch e Rieseberg, 1996). Algumas dessas metodologias são utilizadas na busca de marcadores moleculares, que auxiliam no conhecimento dos padrões de variações genéticas inter e intrapopulacionais que podem ser utilizados como marcador de produção/reprodução, contribuindo para elaboração de estratégias em programas de melhoramento (seleção ou cruzamento). Entre as técnicas desenvolvidas estão a random amplified polymorphic DNA (RAPD) e os microssatélites.
A técnica de RAPD baseia-se na amplificação de segmentos de DNA genômico a partir de oligonucleotídeos iniciadores curtos e aleatórios (Williams et al., 1990). A análise de variação de DNA por meio dessa técnica vem sendo aplicada, com s.ucesso, em diversas espécies, inclusive em codornas. A técnica RAPD tem sido usada para caracterizar fingerprinting de espécies de peixes e aves, e para genotipagem individual dentro das espécies, provando a eficiência e a sensibilidade desse método. Pode ainda ser usada para estimar variabilidade genética, parentesco e níveis de consanguinidade, identificar espécies ou linhagens, realizar análise de progênie e estabelecer estratégia econômica em peixes (Dinesh et al., 1993).
O objetivo deste trabalho foi avaliar a variabilidade genética existente entre três linhagens de codornas (Coturnix japonica), selecionadas para a produção de ovos.
MATERIAL E MÉTODOS
As codornas (Coturnix japonica) utilizadas pertencem ao programa de desenvolvimento de linhagens de codornas da Universidade Estadual de Maringá. Foram avaliadas três linhagens para postura, denominadas amarela, azul e vermelha, de acordo com a anilha de marcação usada para distingui-las. Para as avaliações genéticas, foram utilizados 20 animais de cada linhagem, sendo 10 machos e 10 fêmeas, totalizando 60 animais.
Amostras de DNA foram extraídas de células sanguíneas, utilizando-se o protocolo proposto por Regitano e Coutinho (2001), com modificações. Foi acrescentada uma lavagem com fenol, seguida de precipitação do DNA com sal e lavagem com álcool. O DNA obtido foi ressuspendido em 50μL em tampão TE diluído (Tris 1mM, EDTA 0,1mM pH 8.0) contendo RNAase (20μg/mL). Alíquotas de DNA foram usadas para quantificação por comparação com quantidades conhecidas de DNA de fago λ, em gel de agarose 0,8%. Após a quantificação, as amostras de DNA foram diluídas para 50ng/μL e armazenadas em freezer -20ºC. O estoque concentrado foi armazenado em freezer -20ºC para uso posterior.
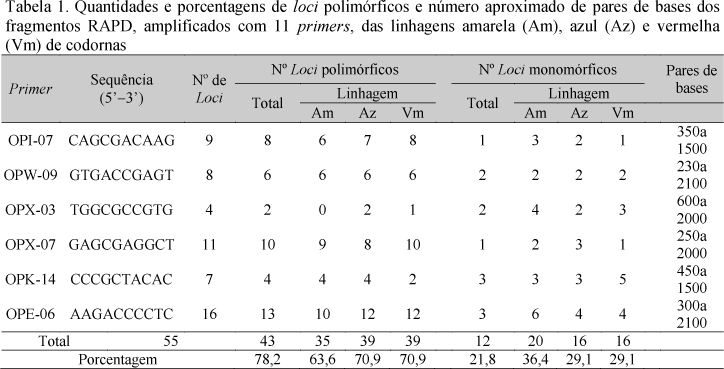
Foram feitos testes com 15 primers RAPD (Operon Technologies Inc. Alameda, CA, USA). Os primers OPW-09, OPI-07, OPX-03, OPX-07, OPK-14 e OPE-06 apresentaram os melhores padrões de bandas e foram selecionados. A mistura de reação de amplificação de cada um dos primers selecionados consistiu de tampão Tris-KCl (Tris-HCl 20mM pH 8,4 e KCl 50mM), MgCl2 2mM, primer 0,46μM, dNTP 0,19mM, 1U/reação de Taq DNA polimerase (Invitrogen), DNA (10ng) e água suficiente para completar 13μL. As reações de amplificação foram realizadas no termociclador MJ Research Inc. programado para 1 ciclo de 4min a 92°C, 40 ciclos de 1min a 92°C, 1min. 30s a 40°C e 2min a 72°C. Imediatamente após o último ciclo de amplificação, a mistura de reação foi mantida durante 5min a 72°C e resfriada durante 20min a 20°C. Controles negativos, sem molde de DNA, foram incluídos em cada reação em cadeia pela polimerase (PCR). Após cada amplificação, os produtos de PCR foram fracionados por eletroforese em gel de agarose 1,4%. Os perfis eletroforéticos foram fotografados, sob luz ultravioleta (UV), com o sistema Kodak EDAS-290. Os tamanhos dos fragmentos de DNA foram estimados por comparação com padrão Ladder 100pb (Invitrogen Life Technologies).
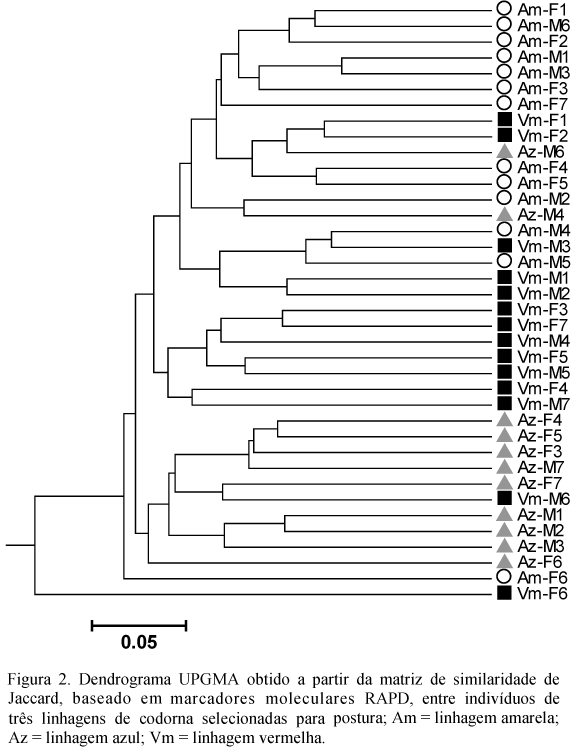
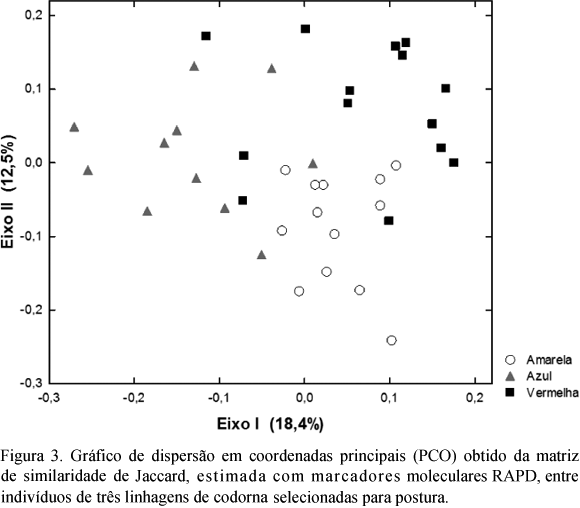
O programa Numerical taxonomy and multivariate analysis system (NTSYS 1.7) (Rohlf, 1989) foi utilizado para obtenção da matriz de similaridade de Jaccard entre indivíduos, a partir da presença ou ausência de bandas de tamanhos moleculares idênticos (mesmos loci). O NTSYS também foi utilizado para realização de outros procedimentos, tendo como ponto de partida a matriz de similaridade de Jaccard. O teste de Mantel (1967) foi utilizado para a detecção de diferenciação genética entre as linhagens de codornas. Com este procedimento, foi testada a significância da correlação entre a matriz de complementos de Jaccard entre indivíduos e uma matriz modelo, construída com base nas linhagens dos pares de indivíduos (Rodrigues et al., 2002). O padrão de divergência genética contido na matriz de similaridade de Jaccard foi representado graficamente, utilizando-se duas modalidades de análises multivariadas. Primeiramente, foi construído um dendrograma com o algoritmo de agrupamento Unweighted Pair-Group Method Using Arithmetic Average (UPGMA). Em uma análise de ordenação, foi construído um gráfico de dispersão em coordenadas principais. O índice de diversidade de Shannon (Zar, 1974) entre as linhagens foi estimado pelo programa computacional PopGene 1.31 (Yeh et al., 1999).
RESULTADOS E DISCUSSÃO
Na Fig. 1, estão ilustrados os perfis eletroforéticos, obtidos com o primer OPE-06, de indivíduos das linhagens de codorna amarela e vermelha. Pode ser constatado o polimorfismo tanto dentro quanto entre as linhagens.
Cada um dos primers selecionados amplificou de 4 a 16 fragmentos, de 230 a 2100pb. Para as análises, foram escolhidos 55 loci RAPD cujos fragmentos formaram bandas intensas (Tab. 1). Quando se consideram as três linhagens simultaneamente, desses 55 loci RAPD, 43 (78,2%) foram polimórficos e 12 (21,8%), monomórficos. Todos os primers produziram, pelo menos, um fragmento monomórfico quando se consideraram todas as linhagens simultaneamente. Por outro lado, nenhum primer produziu banda de DNA encontrada exclusivamente em uma das linhagens. A não ocorrência de bandas exclusivas é um indicativo de que não há diferenciação acentuada entre as linhagens de codorna estudadas.
A diversidade dentro das linhagens foi quantificada pelo número de loci polimórficos, similaridade de Jaccard, e pelo índice de diversidade de Shannon. Na Tab. 1, está indicado que a frequência de loci polimórficos na linhagem amarela foi de 63,6%, enquanto as linhagens azul e vermelha apresentaram 70,9%. As menores estimativas do índice de diversidade de Shannon foram 0,36 para a linhagem amarela e 0,38 referente à azul, enquanto que para a linhagem vermelha esse índice foi de 0,41. As médias das estimativas dos coeficientes de Jaccard dentro das linhagens são mostradas na Tab. 2. Os níveis de similaridade dentro das linhagens foram semelhantes, sendo que a amarela apresentou o maior coeficiente (0,70). Quanto às similaridades intralinhagens, o menor coeficiente (0,61) foi observado entre as linhagens azul e vermelha, enquanto o maior (0,64).
Os três indicadores, frequência de loci polimórficos, índice de diversidade de Shannon e similaridade de Jaccard intralinhagens, são concordantes em revelar a existência de variabilidade genética dentro das linhagens de codorna amarela, azul e vermelha. De acordo com estes resultados, a amarela apresenta diversidade um pouco menor quando comparada com as linhagens azul e vermelha. Portanto, os valores obtidos sugerem que dentro das três linhagens existe variabilidade em níveis que podem ser considerados altos e adequados para exploração em programas de melhoramento.
A diferenciação genética entre as três linhagens foi analisada pelo teste de Mantel, baseado no complemento do coeficiente de similaridade de Jaccard. Foram encontradas correlações significativas em todas as combinações das linhagens estudadas (Tab. 3). A maior correlação encontrada foi 0,39, entre as linhagens amarela e azul, e a menor, 0,29, entre a amarela e a vermelha. Apesar de significativos, os valores de correlação encontrados foram baixos, consequentemente, os dados sugerem que há diferenciação genética entre as linhagens, mas em níveis baixos.
Adicionalmente, a matriz de complementos da similaridade de Jaccard, entre indivíduos, foi utilizada para análise da divergência genética por agrupamento UPGMA e ordenação em coordenadas principais. Na Fig. 2, o dendrograma revela que os indivíduos de uma mesma linhagem tendem a se agrupar. Essa configuração sugere que há baixa divergência entre as linhagens. Da mesma forma, o gráfico em coordenadas principais, representado na Fig. 3, indicou que há diferenciação genética entre as linhagens, com essas formando agrupamentos. Porém, pode ser constatado que há sobreposição dos três grupos, sugerindo baixa divergência.
De modo geral, os três métodos de análises são congruentes em apontar que, embora em níveis pouco acentuados, essas três linhagens de codorna preservam divergência genética. As linhagens amarela e azul são as mais diferenciadas e a amarela e a vermelha, as mais semelhantes geneticamente. Essa baixa divergência genética entre as linhagens, detectada em todas as modalidades de análises empregadas, poderia ser explicada por possível origem em uma população base comum. Caso as linhagens tenham origem em populações divergentes geneticamente, outra possibilidade seria o fluxo gênico entre as linhagens, por cruzamentos em gerações ancestrais.
Os resultados obtidos são reforçados por análises de outros autores. Sharma et al. (2000) e Ali et al. (2002), ao utilizarem RAPD, encontraram variabilidade genética semelhante dentro e entre linhagens de codorna. Em conjunto, os dados da literatura e os deste trabalho sugerem que a técnica RAPD é consistente para a análise de linhagens de codornas. Além disso, Smith et al. (1996) ao trabalharem com RAPD para estimar variabilidade genética dentro e entre populações de perus e galinhas, mostraram que a tecnologia de RAPD pode ser aplicada com sucesso em diferentes espécies.
Evidentemente, o desenvolvimento de linhagens implica a estimação de parâmetros genéticos de características biológicas que são tradicionalmente utilizadas em programas de melhoramento genético. O conhecimento gerado pela genética molecular, associado aos dados obtidos em avaliações genéticas em testes de desempenho, pode contribuir para orientar as decisões durante os procedimentos para a seleção para características de interesse para a coturnicultura.
CONCLUSÕES
Existe alta variabilidade genética dentro das linhagens amarela, azul e vermelha de codornas poedeiras do plantel da Universidade Estadual de Maringá. A divergência genética entre as três linhagens é baixa, mas estatisticamente significativa. A quantidade e a distribuição da diversidade genética revelam potencial para exploração por cruzamentos entre linhagens e seleção.
AGRADECIMENTOS
Aos professores Alberto José Prioli, pelo apoio técnico-científico e ao professor Elias Nunes Martins, pela liberação das aves utilizadas neste experimento.
Recebido em 4 de setembro de 2009
Aceito em 10 de junho de 2010
- ALI, B.A.; AHMED, M.M.M.; BAHIE El-DEEN, M. et al. Genetic variability in the 17th generation of Japanese quail selected for high eggs and meat production. Egyptian Poult. Sci. J., v.22, p.59-71, 2002.
- DINESH, K.R.; LIM, T.M.; CHUA, K.L. et al. RAPD analysis: an efficient of DNA fingerprinting in fisher. Zool. Sci., v.10, p.849-854, 1993.
- FRITSCH, P.; RIESEBERG, L.H. The use of random amplified polymorphic DNA (RAPD) in conservation genetics. In: SMITH, T.B.; WAYNE, R.K. (Eds). Molecular genetic approaches in conservation New York: Oxford University, 1996. p.54-73.
- FURLAN, A.C.; ANDREOTTI, M.O.; MURAKAMI, A.E. Valores energéticos de alguns alimentos determinados com codornas japonesas (Coturnix coturnix japonica). Rev. Bras. Zootec., v.27, p.1147-1150, 1998.
- MANTEL, N. The detection of disease clustering and a generalized regression approach. Cancer Res., v.27, p.209-220, 1967.
- MILLIGAN, B.G.; LEEBENS-MACK, J.; STRAND, A.E. Conservation genetics: beyond the maintenance of marker diversity. Mol. Ecol., v.3, p.423-435, 1994.
- REGITANO, L.C.A.; COUTINHO, L.L. Biologia molecular aplicada à produção animal Brasília: Embrapa Informação Tecnológica, 2001. p.183-184.
- RODRIGUES, F.M.; DINIZ-FILHO, J.A.F.; BATAUS, L.A.M. et al. Hypothesis testing of genetic similarity based on RAPD data using Mantel tests and model matrices. Gen. Mol. Biol., v.25, p.435-439, 2002.
- ROHLF, F.J. NTSYS-PC Numerical taxonomy and multivariate analysis system New York: Exeter Publishers, 1989.
- SHARMA, D.; APPA RAO, K.B.; TOTEY, S.M. Measurement of within and between population genetic variability in quails. Poult. Sci., v.41, p.29-32, 2000.
- SMITH, E.J.; JONES, C.P.; BARTLETT, J. et al. Use of randomly amplified polymorphic DNA markers for the genetic analysis of relatedness and diversity in chickens and turkeys. Poult. Sci., v.75, p.579-584, 1996.
- WILLIAMS, J.G.; KUBELIK, A.R.; LIVAK, K.J. et al. DNA polymorphisms amplified by arbitrary primers are useful as genetic markers. Nucleic Acids Res., v.18, p.6531-6535, 1990.
- YEH, F.C.; YANG, R.; BOYLE, T. POPGENE Version 1.31: Microsoft Windows-based freeware for population genetic analysis. Alberta: University of Alberta and Centre for International Forestry Research, 1999.
- ZAR, J.H. Biostatistical Analisys Englewood Cliffs: Prentice-Hall, 1974.
Datas de Publicação
-
Publicação nesta coleção
04 Ago 2010 -
Data do Fascículo
Jun 2010
Histórico
-
Aceito
10 Jun 2010 -
Recebido
04 Set 2009