Resumos
Foram simuladas diferentes estratégias de seleção para estimar o desempenho fenotípico e a endogamia média na seleção assistida por marcadores, em características quantitativas com valores de herdabilidade de 0,10; 0,40 e 0,70. O sistema de simulação genética (Genesys) foi utilizado para a simulação de três genomas (cada qual constituído de uma única característica cuja distinção estava no valor da herdabilidade), e das populações base e inicial. Cada população inicial foi submetida à seleção assistida por marcadores por 20 gerações consecutivas. Avaliaram-se estratégias de acasalamento entre os genitores selecionados, em diferentes intensidades de seleção (tamanhos populacionais), por meio do acasalamento seletivo entre os melhores e os piores, acasalamento apenas entre os melhores e/ou entre os piores e acasalamento aleatório. Em todos os cenários combinando herdabilidade e intensidade, o acasalamento estratégico utilizando a metodologia da genotipagem seletiva foi superior aos demais, tornando-se mais eficaz na detecção de QTL e, consequentemente, no incremento do valor fenotípico e na minimização das médias endogâmicas ao longo das gerações. Ao utilizar a estratégia seletiva de amostragem, menor tamanho populacional é requerido para otimizar a detecção de QTL à medida que o valor da herdabilidade da característica aumenta.
herdabilidade; seleção estratégica; simulação; QTL
Different strategies of selection were simulated to estimate the phenotypic performance and average inbreeding in selection assisted by markers, for quantitative traits with heritability values of 0.10, 0.40, and 0.70. The genetic simulation system (Genesys) was used for the simulation of three genomes (each one consisting of a single characteristic which distinction was the value of heritability) and base and original populations. Each initial population was subjected to selection assisted by markers for 20 consecutive generations. Strategies of mating between the parents selected in different intensities of selection (population size) were evaluated by selective mating between the best and worst, mating only among the best and/or among the worst and random mating. In all scenarios combining heritability and intensity, the strategic mating using the methodology of selective genotyping was superior to the others, becoming more effective in detecting QTL and, consequently, in the increase of phenotypic value and in the minimization of the inbred average over the generations. By using the selective sampling strategy, smaller population size to optimize the detection of QTL is required, since the value of the heritability of the characteristic increases.
heritability; strategic selection; simulation; QTL
ZOOTECNIA E TECNOLOGIA E INSPEÇÃO DE PRODUTOS DE ORIGEM ANIMAL
M. JangarelliI, V; R.F. EuclydesII, V; A.P.S. CarneiroIII; P.R. CeconIII, V; C.D. CruzIV, V
IAluno de pós-graduação - UFV - Viçosa, MG
IIDepartamento de Zootecnia - UFV - Viçosa, MG
IIIDepartamento de Informática - UFV - Viçosa, MG
IVDepartamento de Biologia Geral - UFV - Viçosa, MG
VBolsista do CNPq
RESUMO
Foram simuladas diferentes estratégias de seleção para estimar o desempenho fenotípico e a endogamia média na seleção assistida por marcadores, em características quantitativas com valores de herdabilidade de 0,10; 0,40 e 0,70. O sistema de simulação genética (Genesys) foi utilizado para a simulação de três genomas (cada qual constituído de uma única característica cuja distinção estava no valor da herdabilidade), e das populações base e inicial. Cada população inicial foi submetida à seleção assistida por marcadores por 20 gerações consecutivas. Avaliaram-se estratégias de acasalamento entre os genitores selecionados, em diferentes intensidades de seleção (tamanhos populacionais), por meio do acasalamento seletivo entre os melhores e os piores, acasalamento apenas entre os melhores e/ou entre os piores e acasalamento aleatório. Em todos os cenários combinando herdabilidade e intensidade, o acasalamento estratégico utilizando a metodologia da genotipagem seletiva foi superior aos demais, tornando-se mais eficaz na detecção de QTL e, consequentemente, no incremento do valor fenotípico e na minimização das médias endogâmicas ao longo das gerações. Ao utilizar a estratégia seletiva de amostragem, menor tamanho populacional é requerido para otimizar a detecção de QTL à medida que o valor da herdabilidade da característica aumenta.
Palavras-chave: herdabilidade, seleção estratégica, simulação, QTL
ABSTRACT
Different strategies of selection were simulated to estimate the phenotypic performance and average inbreeding in selection assisted by markers, for quantitative traits with heritability values of 0.10, 0.40, and 0.70. The genetic simulation system (Genesys) was used for the simulation of three genomes (each one consisting of a single characteristic which distinction was the value of heritability) and base and original populations. Each initial population was subjected to selection assisted by markers for 20 consecutive generations. Strategies of mating between the parents selected in different intensities of selection (population size) were evaluated by selective mating between the best and worst, mating only among the best and/or among the worst and random mating. In all scenarios combining heritability and intensity, the strategic mating using the methodology of selective genotyping was superior to the others, becoming more effective in detecting QTL and, consequently, in the increase of phenotypic value and in the minimization of the inbred average over the generations. By using the selective sampling strategy, smaller population size to optimize the detection of QTL is required, since the value of the heritability of the characteristic increases.
Keywords: heritability, strategic selection, simulation, QTL
INTRODUÇÃO
No mapeamento genético de lócus que controlam características quantitativas (Quantitative Trait Loci - QTL), busca-se uma associação entre o caráter de importância econômica e os marcadores moleculares. A genotipagem de um grande número de indivíduos se faz necessária para a detecção e o mapeamento de QTL, visto que seus efeitos são geralmente pequenos, dificultando sua identificação (Soller et al., 1976).
A estratégia de genotipagem seletiva surge como uma metodologia capaz de aumentar o poder de detecção de QTL. Nela os indivíduos presentes no extremo superior e inferior da distribuição normal dos fenótipos de uma população experimental disponível são genotipados e acasalados, segundo afirmaram Lander e Botstein (1989), Darvasi e Soller (1992), Muranty et al. (1997) e Bovenhuis e Spelman (2000). Ela permite uma análise preliminar de possíveis marcadores que estariam associados aos QTL.
Segundo Lander e Botstein (1989), os indivíduos de uma parte da progênie contribuem mais para a informação de ligação marcador e QTL do que outros. Os indivíduos mais informativos são aqueles cujo genótipo pode ser inferido a partir do seu fenótipo, ou seja, representam os que possuem em seu genótipo os alelos mais favoráveis ou desfavoráveis para a característica avaliada. Os indivíduos centrados próximo à média fenotípica da população pouco contribuem (van Ooijen, 1992). Em uma distribuição normal para um determinado caráter quantitativo, as progênies com valores fenotípicos de mais de um desvio-padrão da média representam aproximadamente 33% de toda a população. Esse percentual contribui com 81% de toda a informação necessária para a ligação do marcador e QTL (van Gestel et al., 2000).
Essa metodologia seletiva de genotipagem e acasalamento representa uma técnica de amostragem que possibilita inferências sobre um universo, a partir do estudo de uma pequena parcela de seus componentes. Ela deve ser realizada adequadamente para garantir a representatividade da população em estudo (Bolfarine e Bussab, 2005).
Este trabalho teve o objetivo de avaliar a estratégia de seleção seletiva na detecção de QTL, em que os acasalamentos foram realizados entre os melhores e os piores genitores selecionados, em analogia às seleções nas quais os acasalamentos dos indivíduos selecionados procederam-se aleatoriamente ou apenas entre os melhores e/ou entre os piores.
MATERIAL E MÉTODOS
Foram utilizados dados simulados pelo sistema de simulação genética Genesys (Genetic System), versão 2009, desenvolvido por Euclydes (1996). Este sistema, escrito na linguagem de programação FORTRAN, permite a criação de genomas complexos, possibilitando a formação de populações seguindo pressuposições genéticas e estatísticas de interesse.
Foram simulados três genomas hipotéticos, separadamente, cuja distinção estava no valor da herdabilidade da característica. Cada genoma foi constituído de uma característica quantitativa com herdabilidade de 0,10; 0,40 e 0,70, respectivamente.
Cada genoma proposto estava caracterizado geneticamente: apresentava 958 centiMorgan (cM) de extensão; os marcadores moleculares foram dispostos estrategicamente a cada cinco cM, totalizando 191 marcadores; 200 lócus quantitativos (QTL) associados à característica, distribuídos ao longo de 40 cromossomos de tamanho aleatório; os efeitos aditivos dos QTL foram simulados seguindo a distribuição normal dos dados fenotípicos; os lócus quantitativos foram dialélicos e não possuíram desvios de dominância e nem epistasia; as frequências gênicas iniciais foram iguais para ambos os sexos; as frequências gênicas iniciais para os marcadores moleculares seguiram distribuição normal, apresentando valores próximos a 0,5; os efeitos de ambiente foram simulados conforme a distribuição normal; os dados fenotípicos simulados apresentaram média de 10,00 unidades e desvio-padrão 2,00 unidades.
Para cada estrutura genômica simulada, foi construída uma população base composta de 500 machos e 500 fêmeas (1.000 indivíduos), todos heterozigotos, não aparentados. Com os 1.000 descendentes escolhidos aleatoriamente em cada população base, obtidos do cruzamento de 100 machos e 100 fêmeas (uma fêmea/macho), produzindo 10 filhos/fêmea/macho (1.000 indivíduos), formaram-se as populações iniciais. Cada população inicial foi submetida à seleção assistida por marcadores (Marker Assisted Selection - MAS) por 20 gerações consecutivas com 20 repetições, visando minimizar os efeitos da flutuação genética. A seleção foi conduzida com a finalidade de incrementar o valor fenotípico.
A partir de cada uma das três populações iniciais, os reprodutores foram selecionados com base nos genótipos de um número de marcadores moleculares que estariam estatisticamente associados a lócus quantitativos. A cada geração, os 10 machos e as 10 fêmeas (uma fêmea/macho) que obtiveram os melhores desempenhos foram acasalados. O número de progênies em cada cruzamento foi dependente da intensidade de seleção admitida. Esses descendentes formavam a geração seguinte. Na MAS foram comparadas três estratégias de acasalamento entre os reprodutores, em cinco cenários (intensidades de seleção) diferentes.
Entre as estratégias de seleção, adotou-se o acasalamento seletivo entre os reprodutores, seguindo a metodologia da genotipagem seletiva. Nele, os machos e as fêmeas selecionados foram ranqueados separadamente, com base em seus genótipos. O acasalamento foi realizado entre os indivíduos posicionados nos extremos opostos, ou seja, procedeu-se ao cruzamento entre os melhores machos (localizados no extremo superior da classificação) e as piores fêmeas (localizadas no extremo inferior da classificação), e vice-versa (piores machos vs melhores fêmeas).
Para verificar a eficácia da metodologia da genotipagem seletiva, outras duas estratégias de acasalamento foram comparadas. Na primeira estratégia, entre os indivíduos selecionados, acasalaram-se os melhores machos com as melhores fêmeas e, consequentemente, os piores machos com as piores fêmeas. Na segunda estratégia, os reprodutores selecionados foram acasalados aleatoriamente.
Foram admitidos cinco diferentes tamanhos de famílias (intensidades de seleção), que representavam 10, 20, 30, 40 e 50% dos indivíduos disponíveis na população experimental (inicial). Assim, um número de 100, 200, 300, 400 e 500 indivíduos foi mantido em cada geração sob MAS, oriundos de 10 acasalamentos (10 machos e 10 fêmeas - uma fêmea/macho), resultando cada qual 10, 20, 30, 40 e 50 descendentes, respectivamente. - mbj
Logo, obtida a população inicial para cada genoma (nível de herdabilidade), foram praticadas 15 seleções assistidas por marcadores, combinando as três estratégias de acasalamento e as cinco intensidades de seleção, todas partindo do mesmo valor fenotípico.
A identificação de associações entre marcadores e QTL na seleção assistida por marcadores deu-se por meio do método da marca simples. Este método verifica a associação entre cada marcador e a característica de interesse. A análise de regressão linear entre os genótipos dos marcadores flanqueadores do QTL e os valores fenotípicos dos descendentes dos acasalamentos foram o teste estatístico utilizado no método. Adotou-se o nível de significância genômica de 5% na regressão linear simples.
Para comparação das estratégias de acasalamento adotadas na MAS, foram estimados os valores fenotípicos em cada geração. Também foram estimadas as médias dos valores da endogamia e do fenótipo durante as 20 gerações. As comparações foram feitas dentro do mesmo nível de herdabilidade e mesma intensidade de seleção.
RESULTADOS E DISCUSSÃO
Na Tab. 1, estão apresentados os valores fenotípicos médios e seus respectivos desvios-padrão obtidos para a característica de baixa herdabilidade (h2 = 0,10) ao longo de 20 gerações sob seleção assistida por marcadores. Todos os processos de seleção partiram do mesmo valor fenotípico na população inicial (10,00 unidades).
Até aproximadamente a sexta geração, observa-se semelhança nos ganhos fenotípicos entre as estratégias de acasalamento, em todas as intensidades consideradas. Entretanto, a partir da sétima geração, observou-se maior incremento ao se adotar o acasalamento entre os melhores e os piores reprodutores selecionados (MP). Comparando as estratégias em que os cruzamentos davam-se ao acaso (AA) e entre os melhores e/ou entre os piores (MM), ambas foram mais similares, especialmente para as intensidades de 20 e 30%. Porém, para as proporções de 10, 40 e 50%, a metodologia MM leva uma pequena vantagem, como pode ser exemplificado pelas médias gerais apresentas na Tab. 2. O acréscimo médio no fenótipo ao final das 20 gerações foi superior ao se aplicar o acasalamento seletivo, no qual os machos e as fêmeas localizados em extremos opostos de uma distribuição se acasalavam.
Em razão de o poder de detecção da ligação marcador e QTL estar relacionado com o número de indivíduos e sua divergência genotípica, Schuster e Cruz (2004) afirmaram que a variabilidade genética favorecida em acasalamentos que envolvam indivíduos contrastantes contribui para melhorias nos programas de seleção. Dessa forma, segundo Lander e Botstein (1989), Darvasi e Soller (1992), Muranty et al. (1997) e Bovenhuis e Spelman (2000), é aceitável que a aplicação da metodologia utilizada na genotipagem seletiva proporcione melhores resultados para a característica sobre a qual os indivíduos foram selecionados.
Analisando-se o parâmetro endogamia, constata-se média mais baixa para o acasalamento seletivo, em todos os cenários admitidos. Equivalência nas médias endogâmicas também é observada entre as outras duas estratégias (AA e MM). A metodologia seletiva (MP) favorece o retardamento da cosanguinidade entre os indivíduos, beneficiando a detecção de QTL e, consequentemente, as respostas a serem obtidas na seleção assistida por marcadores. O acasalamento entre indivíduos divergentes quanto à frequência gênica favorece o desequilíbrio de ligação (Linkage Disequilibrium - LD). O LD representa uma associação não aleatória entre alelos de diferentes lócus em uma população (Flint-Garcia et al., 2003). A escolha do delineamento experimental apropriado envolvendo o cruzamento de indivíduos distintos otimiza o LD (van der Beek et al., 1995).
Dessa forma, a estratégia de acasalar indivíduos contrastantes, ao se considerar a metodologia da genotipagem seletiva, favorece a variabilidade genética e a maximização do desequilíbrio de ligação. A aplicação dessa metodologia, em comparação às demais (AA e MM), beneficia a diversidade entre os indivíduos, mesmo em famílias com grande número de descendentes (intensidades de 40 e 50%). Ela retarda os níveis endogâmicos na população, possibilitando variação genética suficiente para viabilizar a detecção de ligação entre QTL segregante e marcador genético. Nesta característica de baixa herdabilidade, seus benefícios na resposta fenotípica aumentam proporcionalmente com o acréscimo do tamanho da população (intensidade de seleção).
Ressalta-se que a seleção com base no acasalamento seletivo entre os melhores e os piores reprodutores é mais eficiente que as demais (AA e MM), mesmo admitindo diferentes tamanhos populacionais. Ela possibilita incrementos fenotípicos similares ou superiores em populações com menor número de indivíduos. Como exemplo, com apenas 200 descendentes (intensidade de 20%), a partir da 12ª geração, seus ganhos foram mais altos que os obtidos em um programa que utiliza 300 indivíduos e as estratégias de AA e MM. Considerando-se gerações mais avançadas, seu progresso fenotípico pode superar até as intensidades de 40 e 50% que não adotaram a seleção estratégica (MP).
Submetendo-se uma população inicial com a mesma estrutura genômica às estratégias de acasalamento sob MAS, porém considerando-se uma característica quantitativa de média herdabilidade (h2 = 0,40), tem-se na Tab. 3 média geral dos parâmetros valor fenotípico e endogamia, juntamente com seus desvios-padrão. Os valores fenotípicos médios e os respectivos desvios-padrão são apresentados na Tab. 4.
Constata-se, novamente, a eficiência do acasalamento seletivo (MP) sobre as demais estratégias de amostragem, face aos incrementos fenotípicos superiores. Observa-se que o ganho fenotípico médio foi maior em analogia à característica anterior (h2 = 0,10), por considerar um caráter com maior herança genética. Consequentemente, as respostas dos processos seletivos serão superiores. As médias endogâmicas evidenciaram valores ligeiramente mais baixos que o caráter de baixa herdabilidade, embora as diferenças entre os tipos de acasalamento, de acordo com a intensidade, tenham se mantido semelhantes. Estes resultados seguiram os mesmos padrões da característica de baixa herdabilidade, com supremacia da estratégia seletiva, propiciando maiores progressos fenotípicos e simultaneamente maior controle na elevação das taxas endogâmicas.
Os benefícios do acasalamento seletivo não se restringem a uma mesma intensidade de seleção. Conforme mencionado, sua vantagem estende-se para diferentes tamanhos, viabilizando a condução de programas de seleção, face aos menores recursos a serem dispendidos em experimentos com menor número de indivíduos.
Para esta característica, com herdabilidade 0,40, a estratégia seletiva mostrou-se mais eficiente já a partir das primeiras gerações sob MAS, em todas as intensidades. Admitindo tamanhos amostrais distintos, ela supera as demais no incremento fenotípico, requerendo menor número de indivíduos para um mesmo poder de detecção de QTL, a exemplo do que ocorre ao se comparar as proporções de 10 e 20% sob MP com as demais intensidades de maior magnitude, todavia admitindo-se as estratégias AA e MM. É válido ressaltar que nesta característica o acasalamento seletivo proporcionou otimização nos valores fenotípicos para as intensidades de 30 e 40%. O maior vínculo genético do caráter sob seleção favorece melhor performance com a utilização de menor número de indivíduos, devido ao caráter preditivo da herdabilidade na acurácia do valor fenotípico como indicador do valor genético (Falconer e Mackay, 1996).
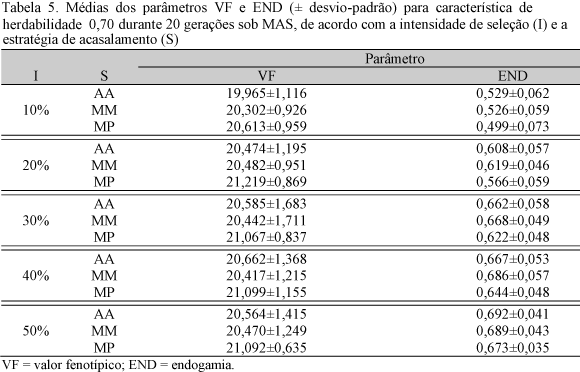
Considerando-se a seleção para característica de alta herdabilidade (0,70), na Tab. 5 apresentam-se as médias gerais dos parâmetros avaliados, com seus desvios-padrão. Na Tab. 6, encontram-se os valores fenotípicos médios e os respectivos desvios-padrão.
Devido à maior acurácia da herdabilidade na predição dos valores fenotípicos, o progresso foi mais acentuado. As diferenças nas médias endogâmicas entre as três estratégias de acasalamento, dentro de cada intensidade, mostraram-se de magnitude inferior. Metodologias que visam implementar a seleção genômica, como as estratégias seletivas, terão mais impacto em características de baixa herdabilidade (Dekkers, 2004; Li et al., 2005). Nelas, a MAS torna-se mais relevante face à importância do ambiente na determinação do valor fenotípico. Todavia, apesar da menor magnitude nas diferenças para minimizar (retardar) os níveis endogâmicos ou maximizar a detecção de QTL (ganhos fenotípicos), a amostragem seletiva foi superior às demais. Em todos os cenários ela proporcionou, em média, melhores respostas nos parâmetros avaliados.
Novamente, os benefícios do acasalamento seletivo estendem-se para tamanho amostral distinto, a exemplo da supremacia no incremento da média fenotípica da intensidade de 20% sob acasalamento seletivo, em comparação às proporções de 30, 40 e 50% em que se adotaram acasalamentos distintos. Este alto vínculo genético da característica sob MAS requer menor número de indivíduos, conforme a maximização nos valores fenotípicos obtidos para a intensidade de 20% sob estratégia seletiva.
CONCLUSÕES
A seleção estratégica, ao acasalar os melhores e os piores genitores selecionados, de acordo com a metodologia da genotipagem seletiva, mostrou-se eficiente na detecção de QTL. O acasalamento seletivo foi superior aos demais (acasalamento aleatório e acasalamento apenas entre os melhores e/ou entre os piores reprodutores) em todas as intensidades de seleção, nos três níveis de herdabilidade. Ao se utilizar a estratégia seletiva de amostragem, menor tamanho populacional é necessário para otimizar a detecção de QTL à medida que o valor da herdabilidade da característica aumenta.
Recebido em 23 de julho de 2009
Aceito em 16 de julho de 2010
E-mail: gmejanga@hotmail.com
- BOLFARINE, H.; BUSSAB, W.O. Elementos de amostragem São Paulo: Edgar Blucher, 2005. 274p.
- BOVENHUIS, H.; SPELMAN, R.J. Selective genotyping to detect quantitative trait loci for multiple traits in outbred populations. J. Dairy Sci, v.83, p.173-180, 2000.
- DARVASI, A.; SOLLER, M. Selective genotyping for determination of linkage between a marker locus and a quantitative trait locus. Theor. Appl. Genet., v.85, p.353-359, 1992.
- DEKKERS, J.C.M. Commercial application of marker-and gene-assisted selection in livestock: Strategies and lessons. J. Anim. Sci., v.82, p.313-328, 2004.
- EUCLYDES, R.F. Uso do sistema para simulação Genesys na avaliação de métodos de seleção clássicos e associados a marcadores moleculares 1996. 149f. Tese (Doutorado) - Universidade Federal de Viçosa, Viçosa, MG.
- FALCONER, D.S.; MACKAY, T.F.C. Introduction to quantitative genetics 4.ed. London: Longman, 1996. 464p.
- FLINT-GARCIA, S.A.; THORNSBERRY, J.M.; BUCKLER, E.S. Structure of linkage disequilibrium in plants. 4., Annu. Rev. Plant. Biol., v.54, p.357-374, 2003.
- LANDER, E.S.; BOTSTEIN, D. Mapping Mendelian factors underlying quantitative traits using RFLP linkage maps. Genetics, v.121, p.185-199, 1989.
- LI, H.; DEEB, N.; ZHOU, H. et al. Chicken quantitative trait loci for growth and body composition associated with the very low density apolipoprotein-II gene. Poult. Sci., v.84, p.697-703, 2005.
- MURANTY, H.; GOFFINET, B.; SANTI, F. Multitrait and multipopulation QTL search using selective genotyping. Genet. Res., v.70, p.259-265, 1997.
- SCHUSTER, I.; CRUZ, C.D. Estatística genômica aplicada a populações derivadas de cruzamentos controlados Viçosa: Editora UFV, 2004. 568p.
- SOLLER, M.; BRODY, T.; GENIZI, A. On the power of experimental designs for the detection of linkage between marker loci and quantitative loci in crosses between inbreed lines. Theor. Appl. Genet., v.47, p.35-39, 1976.
- van der BEEK.; van ARENDONK, J.A.M.; GROEN, A.F. Power of two and three-generation QTL, mapping experiments in an outbred population containing full-sib or half-sib families. Theor. Appl. Genet., v.91, p.1115-1124, 1995.
- van GESTEL, S.; HOUWING-DUISTERMAAT, J.J.; ADOLFSSON, R. et al. Power of selective genotyping in genetic association analyses of quantitative traits. Behav. Genet., v.30, p.141-146, 2000.
- van OOIJEN, J.W. Accuracy of mapping quantitative trait loci in autogamous species. Theor. Appl. Genet., v.84, p.803-811, 1992.
Seleção, acasalamento e genotipagem seletiva e outras estratégias de amostragem na detecção de QTL
Datas de Publicação
-
Publicação nesta coleção
02 Fev 2011 -
Data do Fascículo
Ago 2010
Histórico
-
Recebido
23 Jul 2009 -
Aceito
16 Jul 2010