Resumos
O objetivo desta pesquisa foi avaliar a qualidade microbiológica e o perfil ácido-láctico do queijo de coalho artesanal. Todas as amostras de queijo apresentaram coliformes totais, termotolerantes e presença de Escherichia coli, porém com os valores dentro dos padrões estabelecidos pela legislação vigente no país. O perfil ácido-láctico estudado mostrou uma microbiota heterogênea, constituída por lactobacilos, lactococos, estreptococos e enterococos, confirmadas as espécies Enterococcus faecalis, Enterococcus faecium, Streptococcus thermophilus e Lactococcus lactis.
queijo de coalho artesanal; reação em cadeia da polimerase; bactérias ácido-lácticas
The aim of this study was to evaluate the microbiological quality and lactic-acid profile of artisanal coalho cheese. All cheese samples analyzed showed total coliforms, were thermotolerant, and had Escherichia coli, but all the values were within the standards established by current legislation in the country, and could be considered a food fit for human consumption. The cheese showed a heterogeneous microbiota, being constituted of all tested genus, such as lactobacilli, lactococcus, streptococcus and enterococcus, and confirmed the species: Enterococcus faecalis, Enterococcus faecium, Streptococcus thermophilus and Lactococcus lactis.
artisanal coalho cheese; polymerase chain reaction; lactic-acid bacteria
ZOOTECNIA E TECNOLOGIA E INSPEÇÃO DE PRODUTOS DE ORIGEM ANIMAL ANIMAL SCIENCE AND TECHNOLOGY AND INSPECTION OF ANIMAL PRODUCTS
Avaliação da microbiota bacteriana do queijo de coalho artesanal produzido na região Agreste do estado de Pernambuco
Evaluation of the bacterial microbiota of the artisanal "Coalho" of cheese produced in Agreste Region of the State of Pernambuco
R.A. SilvaI,II; P.A. BismaraII; R.B. MouraII; J.L. Lima FilhoII; A.L.F. PortoI,II; M.T.H. CavalcantiI,II,** Autor para correspondência (corresponding author), E-mail: mtcvsoares@yahoo.com.br
IUniversidade Federal Rural de Pernambuco ‒ UFRPE ‒ Recife, PE
IIUniversidade Federal de Pernambuco ‒ UFPE ‒ Recife, PE
RESUMO
O objetivo desta pesquisa foi avaliar a qualidade microbiológica e o perfil ácido-láctico do queijo de coalho artesanal. Todas as amostras de queijo apresentaram coliformes totais, termotolerantes e presença de Escherichia coli, porém com os valores dentro dos padrões estabelecidos pela legislação vigente no país. O perfil ácido-láctico estudado mostrou uma microbiota heterogênea, constituída por lactobacilos, lactococos, estreptococos e enterococos, confirmadas as espécies Enterococcus faecalis, Enterococcus faecium, Streptococcus thermophilus e Lactococcus lactis.
Palavras-chave: queijo de coalho artesanal, reação em cadeia da polimerase, bactérias ácido-lácticas
ABSTRACT
The aim of this study was to evaluate the microbiological quality and lactic-acid profile of artisanal coalho cheese. All cheese samples analyzed showed total coliforms, were thermotolerant, and had Escherichia coli, but all the values were within the standards established by current legislation in the country, and could be considered a food fit for human consumption. The cheese showed a heterogeneous microbiota, being constituted of all tested genus, such as lactobacilli, lactococcus, streptococcus and enterococcus, and confirmed the species: Enterococcus faecalis, Enterococcus faecium, Streptococcus thermophilus and Lactococcus lactis.
Keywords: artisanal coalho cheese, polymerase chain reaction, lactic-acid bacteria
INTRODUÇÃO
A produção de queijo de coalho representa uma atividade bastante significativa para a economia regional, visto que em determinadas localidades é a principal fonte de renda e sobrevivência da população (Almeida et al., 2010). No Nordeste do Brasil a maior parte da produção de queijo de coalho é realizada em pequenas e médias queijarias, as quais movimentam, mensalmente, algo em torno de 10 milhões de reais, o que torna essa atividade importante tanto no âmbito social quanto no econômico (Perry, 2004).
De acordo com Almeida et al. (2010), o queijo de coalho é uma representação genuína da tradição e cultura do estado de Pernambuco, onde está sendo desenvolvido um trabalho de certificação desse queijo visando a uma identidade própria que irá diferenciá-lo dos demais produzidos na região Nordeste.
As propriedades organolépticas típicas e o aroma particular dos queijos obtidos com leite cru, tais como o queijo de coalho artesanal, estão associados com atributos do próprio leite, relacionados à raça e ao tipo de nutrição das vacas, ao processo de fabricação básico do queijo tradicional e à microbiota natural autóctone, responsáveis pela fermentação e maturação próprias da região produtora (Beresford et al., 2001; Giannino et al., 2009). O conhecimento sobre essa microbiota ácido-láctica especial é importante para assegurar qualidade, autenticidade e rastreabilidade do produto artesanal, com indicação de origem e proteção de origem controlada (Giannino et al., 2009).
O estudo da diversidade microbiana desses produtos artesanais está sendo realizado com o auxílio de técnicas que utilizam o DNA, entre elas a reação em cadeia da polimerase (PCR), e independentemente de cultura. A amplificação de regiões específicas por meio de primer promove a identificação de gêneros e espécies, garantindo maior rapidez nos resultados (Dolci et al., 2008a; Giannino et al., 2009).
Sendo assim, o objetivo desta pesquisa foi analisar a microbiota bacteriana do queijo de coalho artesanal da região Agreste de Pernambuco, quanto à qualidade microbiológica e ao perfil ácido-láctico.
MATERIAL E MÉTODOS
O queijo de coalho artesanal foi coletado em unidades fabris com selo da Adagro-PE (Agência de Defesa e Fiscalização Agropecuária de Pernambuco), pronto para a comercialização, nos meses de março e julho de 2008, nos municípios de Venturosa, Correntes, Capoeiras e Arcoverde, e em duas unidades sem registro, situadas em São Bento do Una e Cachoeirinha, todos localizados na região Agreste de Pernambuco. O produto foi acondicionado em caixas isotérmicas e imediatamente transportado para o laboratório, onde estas foram mantidas a 10°C, até o momento das análises.
As amostras de queijo utilizadas foram analisadas quanto à qualidade microbiológica para coliformes totais AOAC/2002 (Association..., 2002a) e para coliformes termotolerantes AOAC/2002 (Association..., 2002b).
Para avaliar o perfil das bactérias ácido-lácticas das amostras coletadas, os queijos foram dissolvidos em solução citrato trisódico esterilizado, 2% p/v, na proporção de 1g de queijo para 9mL de solução, maceradas manualmente com a ajuda de um almofariz e pistilo até se obter um homogenato, que foi posteriormente centrifugado a 25.758xg. O sobrenadante foi descartado e o precipitado foi novamente macerado. Essa operação foi realizada por três vezes. O precipitado encontrado ao final foi utilizado para identificar o perfil de bactérias ácido-láticas com primer espécie-específicos (Tab. 1) na técnica de amplificação do DNA pela reação em cadeia da polimerase, incluindo as etapas de desnaturação inicial a 94ºC por 3mim, 30 ciclos completos de 1 minuto ao total, divididos em desnaturação a 94ºC, anelamento com temperatura variável de acordo com o primer usado e extensão a 72ºC; e um passo final de extensão a 72ºC por 5min. Após a amplificação, o material foi visualizado por meio de eletroforese, preparada na concentração de 1% de agarose dissolvida em tampão TEB (89 mM Trizma base, 89 mM ácido bórico, 20 mM EDTA) pH 8,0, corado com Gel Red (Uniscience do Brasil, São Paulo Ltda.).
RESULTADOS E DISCUSSÃO
Os resultados obtidos na avaliação da qualidade microbiológica do queijo de coalho estão apresentados na Tab. 2. Todas as amostras de queijos avaliadas apresentaram coliformes totais e termotolerantes, sendo confirmada a presença de Escherichia coli em 100% delas. Os coliformes totais variaram no mínimo de 2,82 x 102 NMP/g para o queijo do município de Arcoverde e no máximo de 1,09 x 103 NMP/g para o queijo de Capoeiras. Os coliformes termotolerantes apresentaram valores no mínimo de 1NMP/g para os municípios de Arcoverde, Correntes e São Bento do Una, e no máximo de 8,78 NMP/g para Cachoeirinha. Entretanto, todos os valores estão dentro dos limites estabelecidos pela legislação vigente RDC 12 (Brasil, 2001), que apresenta o limite de tolerância para coliformes termotolerantes de 5,0 x 102 NMP/g de amostra e 5,0 x 102 NMP/g para coliformes totais. Resultados semelhantes foram encontrados por Feitosa et al. (2003), que, ao estudarem microrganismos indicadores de higiene em queijos produzidos no Rio Grande do Norte, constataram a presença de coliformes termotolerantes em 36,4% das amostras estudadas, 3 a 7NMP/g, dentro do limite descrito na legislação. Duarte et al. (2005), quando estudaram microrganismos indicadores de higiene em queijos de coalho produzidos e comercializados em Pernambuco, encontraram coliformes termotolerantes em valores acima do aceitável em 44,10% das amostras avaliadas; Oliveira et al. (2010), quando estudaram a qualidade microbiológica do queijo de coalho comercializado no município do Cabo de Santo Agostinho, em Pernambuco, encontraram 80,95% das amostras estudadas com valores superiores a 500NMP/g, acima do limite estabelecido pela legislação vigente, sendo observada a presença de E. coli em 64,29% dessas amostras. Todos os autores citados acima atribuíram os resultados obtidos à falta de condições higiênico-sanitárias adequadas na produção e/ou na comercialização desses produtos.
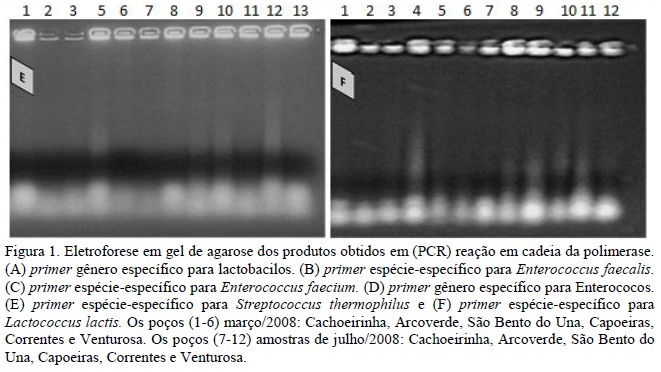
Com o objetivo de conhecer quais as principais espécies que interagem promovendo o sabor inigualável do queijo de coalho artesanal da região Agreste do estado de Pernambuco, a sua microbiota ácido-láctica foi avaliada. Os resultados obtidos da avaliação da microbiota lática estão apresentados nas Fig. 1A a 1F. A população bacteriana desse queijo é heterogênea, haja vista que todos os gêneros e espécies testados foram encontrados, sendo, portanto, o sabor diferenciado presente no produto em decorrência dessa associação microbiana. Além disso, a intensidade das bandas encontradas está relacionada à quantidade de bactérias de cada tipo, pois a própria população bacteriana extraída diretamente do queijo foi usada como amostra para a identificação por biologia molecular.
Esse tipo de interação, entre gêneros e espécies, não é privilégio do queijo de coalho, mas está presente em vários produtos artesanais. Assim, de acordo com Ogier et al. (2004), vários produtos lácteos representam um ecossistema microbiano complexo, associados à grande diversidade de sabores, aromas e texturas. Muitas bactérias contribuem para a qualidade organoléptica dos queijos ou leites fermentados.
Tem se estudado a diversidade bacteriana sempre associando um ecossistema complexo às características organolépticas diferenciadas nestes produtos, semelhante ao estudo deste trabalho, em que os principais gêneros de bactérias ácido-láticas estão presentes e formam uma comunidade estrategicamente organizada. Serhan et al. (2009), ao estudarem a diversidade bacteriana presente em um queijo artesanal libanês produzido com leite cru de cabra, afirmaram que, entre as bactérias ácido-láticas, os gêneros mais encontrados foram Lactobacillus, Streptococcus, Enterococcus, Lactococcus e Leuconostoc, e que queijos produzido com leite cru e seguindo processos tradicionais de fabricação apresentam diversidade microbiológica, que precisa ser conhecida para que haja a manutenção de produtos artesanais típicos com tradição cultural. Vale ressaltar que essa mesma associação foi observada nas amostras de queijo de coalho deste estudo.
A Fig. 1A corresponde aos resultados obtidos para o gênero Lactobacillus, obtendo-se confirmação em 100% das amostras analisadas. A presença desse microrganismo é comum em produtos derivados de leite, sendo importante na qualidade organoléptica deles. No estudo de Serhan et al. (2009), 44,9% das amostras isoladas e identificadas por métodos clássicos foram Lactobacillus curvatus e L. plantarum, sendo este último confirmado por eletroforese com gradiente de temperatura (TGGE) como a principal espécie de lactobacilos no queijo Darfiyeh. Na sequência, as Fig. 1B, C e D apresentam os resultados obtidos na eletroforese dos produtos de PCR para Enterococcus faecalis e E. faecium e o gênero Enterococcus, com 100% de confirmação nas amostras analisadas.
Estes resultados se assemelham aos de vários relatos sobre a análise da microbiota de queijos de origem artesanal em várias partes do mundo, como o queijo São Jorge, produzido em Portugal (Kongo et al., 2007), queijo Raschera, proveniente da Itália (Dolci et al., 2008b), Darfiyeh, encontrado na Líbia (Serhan et al., 2009), queijos espanhóis artesanais de leite de cabra (Martín-Platero et al., 2009), queijo Fontina, originado da Itália (Giannino et al., 2009), e queijo Istriano, presente na Croácia (Fuka et al., 2010). Esses autores sempre relacionaram as espécies do gênero enterococos aos aspectos organolépticos dos produtos artesanais, ressaltando a participação na maturação dos queijos. Segundo Beresford et al. (2001), especialmente nos queijos da região mediterrânea, há efeito forte e positivo sobre o desenvolvimento de aromas e sabores.
A interação das espécies E. faecalis e E. faecium com outras bactérias ácido-láticas também é importante para a formação dos produtos artesanais, como observaram Kongo et al. (2007), que identificaram as espécies de bactérias ácido-lácticas dominantes presentes no queijo São Jorge, um queijo típico português. Os resultados indicaram que Lactobacillus seguidos pelos Enterococus foram os gêneros dominantes. As espécies mais frequentes foram Lactobacillus paracasei, L. rhamnosus, E. faecalis e E. faecium. Outro queijo artesanal em que há associação dos enterococos com outras bactérias ácido-lácticas é o queijo Darfiyeh, estudado por Sehran et al. (2009). Esses autores encontraram os enterococos fazendo parte da microbiota do produto analisado, apresentando esse fato como comum para os queijos da região mediterrânea produzidos com leite cru de várias espécies animais. Esses autores citam o E. faecium e o Lactococcus lactis subs. lactis como os principais microrganismos responsáveis pelas características peculiares desse tipo de queijo. Fuka et al. (2010) também encontraram os enterococos participando ativamente da microbiota do queijo Istriano e reconheceram a importância desse gênero como responsável pelo sabor e aroma típicos desse produto.
Os resultados apresentados na Fig. 1E demonstram que em 100% das amostras de queijo testadas para Streptococcus thermophilus foi encontrado DNA dessa bactéria. Esse microrganismo também está presente em outros queijos artesanais, tais como no queijo italiano Toma piemontese de denominação de origem. Fortina et al. (2003), ao utilizarem o mesmo primer descrito neste trabalho, identificaram S. thermophilus como componente minoritário da microbiota característica, e Ercolini et al. (2008) encontraram no queijo Caciocavalo silano, um queijo italiano artesanal com denominação de origem protegida (PDO), S. thermophilus como parte da microbiota específica do produto.
Os resultados obtidos para lactococos mostraram a presença em 100% das amostras testadas (Fig. 1F), sendo a espécie Lactococcus lactis subs. lactis confirmada por meio de PCR. Este dado é importante, pois, de acordo com Flórez et al. (2006), o L. lactis é uma bactéria ácido-láctica usada mundialmente como um organismo iniciador em indústrias lácticas para a fabricação e maturação de queijos e outros produtos fermentados. O desenvolvimento do L. lactis no leite proporciona ótimas condições para a formação do coágulo na coagulação do leite, previne o crescimento de bactérias patogênicas e cria condições bioquímicas ideais para a maturação. Além disso, L. lactis participa no desenvolvimento da textura e do sabor do queijo por via proteolítica e de sistemas catabólicos de aminoácidos.
A presença de L. lactis subs. lactis é comum no leite e em produtos frescos como o queijo de coalho, pois, durante o processo de maturação, essas bactérias apresentam dificuldades fisiológicas de sobrevivência em razão de certas condições ambientais, como diminuição da atividade de água e acidificação e proteólise intensas produzidas por outras bactérias do ecossistema. Esse fato foi ressaltado por Dolci et al. (2008a), quando avaliaram a dinâmica da microbiota dominante durante o processo de fabricação e maturação do queijo Castelmagno (denominação protegida na origem). Esses autores analisaram amostras do leite, da massa e do queijo em diferentes estágios de maturação, sendo a identificação das espécies realizadas por PCR-DGGE, em que o L. lactis subs. lactis foi a espécie mais frequentemente encontrada durante o processo de fabricação, enquanto L. plantarum e L. paracasei foram isolados com maior frequência nos queijos maturados. Entretanto, Dolci et al. (2008b), quando estudaram o queijo artesanal italiano Raschera (denominação protegida na origem), encontraram L. lactis subs. lactis como bactéria principal durante todo o processo de fabricação e maturação. Dessa forma, sugere-se que a presença dessa bactéria ao longo de todo o processo de fabricação e maturação deve ser sempre analisada, pois, dependendo das condições intrínsecas do produto, ela pode ser eliminada.
CONCLUSÕES
Esse estudo sugere que as características organolépticas do produto estão baseadas em um ecossistema heterogêneo com a interação de todos os gêneros testados, estando a qualidade sanitária dos queijos de coalho utilizados como amostras dentro da legislação vigente.
AGRADECIMENTOS
À Fundação de Amparo a Ciência e Tecnologia do Estado de Pernambuco (FACEPE); ao Laboratório de Imunopatologia Keizo Asami (LIKA); à Universidade Federal Rural de Pernambuco (UFRPE); à Universidade Federal de Pernambuco (UFPE); aos laticínios produtores de queijo de coalho dos municípios estudados do Agreste do estado de Pernambuco; aos técnicos em laticínio Benoit Paquereau e Torquato Marques dos Santos.
REFERÊNCIAS
ALMEIDA, S.L.; JÚNIOR, P.G.F.; GUERRA, J.R.F. A estratégia de internacionalização de negócios na perspectiva da tradução cultural: o caso da indicação geográfica no agronegócio. RIAE - Rev. Ibero-Americana de Estratégia, v.9, p.74-97, 2010.
ASSOCIATION of oficial analytical chemists-AOAC (991.14). Coliforms and Escherichia coli Counts in Foods, Dry Rehydratable Film (Petrifilm Count Plate) Methods (3M Microbiology, 225-5S 3M Center, St. Paul, MN 55144, USA), 2002a.
ASSOCIATION of oficial analytical chemists-AOAC (986.33). Bacterial and Coliform Counts in Milk, Dry Rehydratable Film Method (3M Microbiology/3M, 225-5S 3M Center, St. Paul, MN 55144 USA), 2002b.
BERESFORD, T.P.; FITZSIMONS, N.A.; BRENNAN, N.L. et al. Recent advances in cheese microbiology. Int. Dairy J., v.11, p.259-274, 2001.
BRASIL. Ministério da Saúde. Agência Nacional de Vigilância Sanitária. Regulamento Técnico Sobre os Padrões Microbiológicos para Alimentos. Diário Oficial da República Federativa do Brasil, Brasília, Resolução RDC n.12, p.1-54, 02/01/2001.
CHENG, S.; MCCLESKEY, F.K.; GRESS, M.J. et al. PCR assay for identification of Enterococcus faecium. J. Clin. Microbiol., v.35, p.49-56, 1997.
CORROLER, D.; DESMASURES, N.; GUÉGUEN, M. Correlation between polymerase chain reaction analysis of the histidine biosynthesis operon, randomly amplified polymorphic DNA analysis and phenotypic characterization of dairy Lactococcus isolates. Appl. Microbiol. Biotechnol., v.51, p.91-99, 1999.
DOLCI, P.; ALESSANDRIA, V.; RANTSIOU, K. et al. Microbial dynamics of Castelmagno PDO, a traditional Italian cheese, with a focus on lactic acid bacteria ecology. Int. J. Food Microbiol., v.122, p.302-311, 2008a.
DOLCI, P.; ALESSANDRIA, V.; ZEPPA, G. et al. Microbiological characterization of artisanal Raschera PDO cheese: Analysis of its indigenous lactic acid bacteria. Food Microbiol., v.25, p.392-399, 2008b.
DUARTE, D.A.M.; SCHUCH, D.M.T.; SANTOS, S.B. et al. Pesquisa de Listeria monocytogenes e microrganismos indicadores higiênico-sanitários em queijo de coalho produzido e comercializado no estado do Pernambuco. Arq. Inst. Biol., v.72, p.297-302, 2005.
DUTKA-MALEN, S.; EVERS, S.; COURVALIN, S.P. Detection of glycopeptide resistance genotypes and identification to the species level of clinically relevant enterococci by PCR. J. Clin. Microbiol., v.33, p.24-27, 1995.
ERCOLINI, D.; FRISSO, G.; MAURIELLO, G. et al. Microbial diversity in Natural Whey Cultures used for the production of CaciocavalloSilano PDO cheese. Int. J. Food Microbiol., v.124, p.164-170, 2008.
FEITOSA, T.; BORGES, M.F.; NASSU, R.T. et al. Pesquisa de Salmonella sp., Listeria sp. e microrganismos indicadores higiênico- sanitários em queijos produzidos no estado do Rio Grande do Norte. Cienc. Tecnol. Aliment., v.23, p.162-165, 2003.
FUKA, M.M.; ENGEL, M.; SKELIN, A. et al. Bacterial communities associated with the production of artisanal Istrian cheese. Int. J. Food Microbiol., v.142, p.19-24, 2010.
FORTINA, M.G.; RICCI, G.; ACQUATI, A. et al. Genetic characterization of some lactic acid bacteria occurring in an artisanal protected denomination origin (PDO) Italian cheese, the Toma piemontese. Food Microbiol., v.20, p.397-404, 2003.
GIANNINO, M.L. Study of microbial diversity in raw milk and fresh curd used for Fontina cheese production by culture independent methods. Int. J. Food Microbiol., v.130, p.188-195, 2009.
KE, D.; PICARD, F.J.; MARTINEAU, F. et al. Development of a PCR assay for rapid detection of enterococci. J. Clin. Microbiol., v.37, p.3497-3503, 1999.
KONGO, J.M.; HO, A.J.; MALCATA, F.X. et al. Characterization of dominant lactic acid bacteria isolated from São Jorge cheese, using biochemical and ribotyping methods. J. Appl. Microbiol., v.103, p.1838-1844, 2007.
LICK, S.; KELLER, M.; BOCKELMANN, W. et al. Rapid identification of Streptococcus thermophilus by primer-specific PCR amplification based on its lacZ gene. Syst. Appl. Microbiol., v.19, p.74-77, 1996.
MARTÍN-PLATERO, A.M.; MAQUEDA, M.; VALDIVIA, E. et al. Polyphasic study of microbial communities of two Spanish farmhouse goats' milk cheeses from Sierra de Aracena. Food Microbiol, v.26, p.294-304, 2009.
OGIER, J.C.; LAFARGE, V.; GIRARD, V. et al. Molecular fingerprinting of dairy microbial ecosystems by use of temporal temperature and denaturing gradient gel electrophoresis. Appl. Environ. Microbiol., v.70, p.5628-5643, 2004.
OLIVEIRA, K.A.; EVÊNCIO NETO, J.; de PAIVA, J.E. et al. Qualidade microbiológica do queijo de coalho comercializado no Município do Cabo de Santo Agostinho, Pernambuco, Brasil. Arq. Inst. Biol., v.77, p.435-440, 2010.
PERRY, K.S.P. Queijos: aspectos químicos, bioquímicos e microbiológicos. Quim. Nova, v.27, p.293-300, 2004.
PU, Z.Y.; DOBOS, M.; LIMSOWTIN, G.K. et al. Integrated polymerase chain reaction-based procedures for the detection and identification of species and subspecies of the Gram-positive bacterial genus Lactococcus. J. Appl. Microbiol., v.93, p.353-361, 2002.
SERHAN, M.; CAILLIEZ-GRIMAL, C.; BORGES, F. et al. Bacterial diversity of Darfiyeh, a Lebanese artisanal raw goat's milk cheese. Food Microbiol., v.26, p.645-652, 2009.
WARD, L.J.H.; TIMMINS, M.J. Differentiation of Lactobacillus casei, Lactobacillus paracasei and Lactobacillus rhamnosus by polymerase chain reaction. Letters Appl. Microbiol., v.29, p.90-92, 1999.
Recebido em 1 de julho de 2011
Aceito em 7 de agosto de 2012
- ALMEIDA, S.L.; JÚNIOR, P.G.F.; GUERRA, J.R.F. A estratégia de internacionalização de negócios na perspectiva da tradução cultural: o caso da indicação geográfica no agronegócio. RIAE - Rev. Ibero-Americana de Estratégia, v.9, p.74-97, 2010.
- ASSOCIATION of oficial analytical chemists-AOAC (991.14). Coliforms and Escherichia coli Counts in Foods, Dry Rehydratable Film (Petrifilm Count Plate) Methods (3M Microbiology, 225-5S 3M Center, St. Paul, MN 55144, USA), 2002a.
- ASSOCIATION of oficial analytical chemists-AOAC (986.33). Bacterial and Coliform Counts in Milk, Dry Rehydratable Film Method (3M Microbiology/3M, 225-5S 3M Center, St. Paul, MN 55144 USA), 2002b.
- BERESFORD, T.P.; FITZSIMONS, N.A.; BRENNAN, N.L. et al. Recent advances in cheese microbiology. Int Dairy J., v.11, p.259-274, 2001.
- BRASIL. Ministério da Saúde. Agência Nacional de Vigilância Sanitária. Regulamento Técnico Sobre os Padrões Microbiológicos para Alimentos. Diário Oficial da República Federativa do Brasil, Brasília, Resolução RDC n.12, p.1-54, 02/01/2001.
- CHENG, S.; MCCLESKEY, F.K.; GRESS, M.J. et al PCR assay for identification of Enterococcus faecium J. Clin. Microbiol, v.35, p.49-56, 1997.
- CORROLER, D.; DESMASURES, N.; GUÉGUEN, M. Correlation between polymerase chain reaction analysis of the histidine biosynthesis operon, randomly amplified polymorphic DNA analysis and phenotypic characterization of dairy Lactococcus isolates. Appl. Microbiol. Biotechnol., v.51, p.91-99, 1999.
- DOLCI, P.; ALESSANDRIA, V.; RANTSIOU, K. et al Microbial dynamics of Castelmagno PDO, a traditional Italian cheese, with a focus on lactic acid bacteria ecology. Int. J. Food Microbiol., v.122, p.302-311, 2008a.
- DOLCI, P.; ALESSANDRIA, V.; ZEPPA, G. et al Microbiological characterization of artisanal Raschera PDO cheese: Analysis of its indigenous lactic acid bacteria. Food Microbiol., v.25, p.392-399, 2008b.
- DUARTE, D.A.M.; SCHUCH, D.M.T.; SANTOS, S.B. et al Pesquisa de Listeria monocytogenes e microrganismos indicadores higiênico-sanitários em queijo de coalho produzido e comercializado no estado do Pernambuco. Arq. Inst. Biol., v.72, p.297-302, 2005.
- DUTKA-MALEN, S.; EVERS, S.; COURVALIN, S.P. Detection of glycopeptide resistance genotypes and identification to the species level of clinically relevant enterococci by PCR. J. Clin. Microbiol., v.33, p.24-27, 1995.
- ERCOLINI, D.; FRISSO, G.; MAURIELLO, G. et al Microbial diversity in Natural Whey Cultures used for the production of CaciocavalloSilano PDO cheese. Int. J. Food Microbiol., v.124, p.164-170, 2008.
- FEITOSA, T.; BORGES, M.F.; NASSU, R.T. et al Pesquisa de Salmonella sp., Listeria sp. e microrganismos indicadores higiênico- sanitários em queijos produzidos no estado do Rio Grande do Norte. Cienc. Tecnol. Aliment, v.23, p.162-165, 2003.
- FUKA, M.M.; ENGEL, M.; SKELIN, A. et al Bacterial communities associated with the production of artisanal Istrian cheese. Int. J. Food Microbiol., v.142, p.19-24, 2010.
- FORTINA, M.G.; RICCI, G.; ACQUATI, A. et al Genetic characterization of some lactic acid bacteria occurring in an artisanal protected denomination origin (PDO) Italian cheese, the Toma piemontese. Food Microbiol., v.20, p.397-404, 2003.
- GIANNINO, M.L. Study of microbial diversity in raw milk and fresh curd used for Fontina cheese production by culture independent methods. Int. J. Food Microbiol., v.130, p.188-195, 2009.
- KE, D.; PICARD, F.J.; MARTINEAU, F. et al Development of a PCR assay for rapid detection of enterococci. J. Clin. Microbiol., v.37, p.3497-3503, 1999.
- KONGO, J.M.; HO, A.J.; MALCATA, F.X. et al Characterization of dominant lactic acid bacteria isolated from São Jorge cheese, using biochemical and ribotyping methods. J. Appl. Microbiol., v.103, p.1838-1844, 2007.
- LICK, S.; KELLER, M.; BOCKELMANN, W. et al Rapid identification of Streptococcus thermophilus by primer-specific PCR amplification based on its lacZ gene. Syst. Appl. Microbiol., v.19, p.74-77, 1996.
- MARTÍN-PLATERO, A.M.; MAQUEDA, M.; VALDIVIA, E. et al Polyphasic study of microbial communities of two Spanish farmhouse goats' milk cheeses from Sierra de Aracena. Food Microbiol, v.26, p.294-304, 2009.
- OGIER, J.C.; LAFARGE, V.; GIRARD, V. et al Molecular fingerprinting of dairy microbial ecosystems by use of temporal temperature and denaturing gradient gel electrophoresis. Appl. Environ. Microbiol., v.70, p.5628-5643, 2004.
- OLIVEIRA, K.A.; EVÊNCIO NETO, J.; de PAIVA, J.E. et al Qualidade microbiológica do queijo de coalho comercializado no Município do Cabo de Santo Agostinho, Pernambuco, Brasil. Arq. Inst. Biol., v.77, p.435-440, 2010.
- PERRY, K.S.P. Queijos: aspectos químicos, bioquímicos e microbiológicos. Quim. Nova, v.27, p.293-300, 2004.
- PU, Z.Y.; DOBOS, M.; LIMSOWTIN, G.K. et al Integrated polymerase chain reaction-based procedures for the detection and identification of species and subspecies of the Gram-positive bacterial genus Lactococcus. J. Appl. Microbiol., v.93, p.353-361, 2002.
- SERHAN, M.; CAILLIEZ-GRIMAL, C.; BORGES, F. et al Bacterial diversity of Darfiyeh, a Lebanese artisanal raw goat's milk cheese. Food Microbiol., v.26, p.645-652, 2009.
- WARD, L.J.H.; TIMMINS, M.J. Differentiation of Lactobacillus casei, Lactobacillus paracasei and Lactobacillus rhamnosus by polymerase chain reaction. Letters Appl. Microbiol., v.29, p.90-92, 1999.
Datas de Publicação
-
Publicação nesta coleção
10 Jan 2013 -
Data do Fascículo
Dez 2012
Histórico
-
Recebido
01 Jul 2011 -
Aceito
07 Ago 2012