Resumos
A utilização de marcadores moleculares pode servir para direcionar cruzamentos, confirmar novos híbridos e identificar genótipos para fins comerciais. Nesse contexto, o objetivo deste trabalho foi analisar a diversidade genética entre cultivares e híbridos de Brachiaria spp. e Panicum maximum usando marcadores moleculares do tipo RAPD (Polimorfismos de DNA amplificados ao acaso). Foram 22 genótipos analisados com 10 primers, os quais amplificaram 178 fragmentos polimórficos de DNA, que foram usados para estimar a similaridade genética por meio do coeficiente de Jaccard. Os valores de similaridade obtidos variaram de 0,066 a 0,841. A estrutura genética entre todos os genótipos estudados foi estimada pelo método UPGMA (Método de agrupamento com médias aritméticas não ponderadas), revelando três grupos distintos com altos valores de bootstrap (>89%). Os resultados demonstraram que a técnica RAPD oferece uma maneira rápida, relativamente barata e útil para a caracterização da diversidade genética entre as diferentes cultivares e híbridos de Brachiaria ssp. e P. maximum analisados.
Brachiaria spp.; forrageiras; marcadores moleculares; Panicum maximum; RAPD
The use of molecular markers may serve to direct crossings, confirm new hybrids and identify new genotypes for commercial purposes. In that context, this research aimed to analyze the genetic diversity among cultivars and hybrids of Brachiaria spp. and P. maximum using molecular markers of the type RAPD (Random amplified polymorphic DNA). It was analyzed 22 genotypes with 10 primers, which amplified 178 DNA polymorphic fragments, which were used to estimate the similarity using Jaccard coefficient. The values of similarity ranged from 0.066 to 0.841. The genetic structure among genotypes was estimated by UPGMA (Unweighted pair-group method with arithmetical average) and revealed three distinct groups with high bootstrap values (>89%). The results showed that the RAPD is a fast, relatively inexpensive and useful technique for genetic divergence characterization between different cultivars and hybrids of Brachiaria spp. and P. maximum.
Brachiaria spp.; forages; molecular markers; Panicum maximum; RAPD
ARTIGOS CIENTÍFICOS
PRODUÇÃO ANIMAL
Diversidade genética molecular entre cultivares e híbridos de Brachiaria spp. e Panicum maximum
Molecular genetic diversity among cultivars and hybrids of Brachiaria spp. and Panicum maximum
Mariana Castro da Costa AlmeidaI; Lucimara ChiariII,1 1 Autor para correspondência. ; Liana JankII; Cacilda Borges do ValleII
IUniversidade Católica Dom Bosco (UCDB), Campo Grande, MS, Brasil
IIEmbrapa Gado de Corte, BR 262 km 4, CP 154, 79002-970, Campo Grande, MS, Brasil. E-mail: lchiari@cnpgc.embrapa.br
RESUMO
A utilização de marcadores moleculares pode servir para direcionar cruzamentos, confirmar novos híbridos e identificar genótipos para fins comerciais. Nesse contexto, o objetivo deste trabalho foi analisar a diversidade genética entre cultivares e híbridos de Brachiaria spp. e Panicum maximum usando marcadores moleculares do tipo RAPD (Polimorfismos de DNA amplificados ao acaso). Foram 22 genótipos analisados com 10 primers, os quais amplificaram 178 fragmentos polimórficos de DNA, que foram usados para estimar a similaridade genética por meio do coeficiente de Jaccard. Os valores de similaridade obtidos variaram de 0,066 a 0,841. A estrutura genética entre todos os genótipos estudados foi estimada pelo método UPGMA (Método de agrupamento com médias aritméticas não ponderadas), revelando três grupos distintos com altos valores de bootstrap (>89%). Os resultados demonstraram que a técnica RAPD oferece uma maneira rápida, relativamente barata e útil para a caracterização da diversidade genética entre as diferentes cultivares e híbridos de Brachiaria ssp. e P. maximum analisados.
Palavras-chave:Brachiaria spp., forrageiras, marcadores moleculares, Panicum maximum, RAPD.
ABSTRACT
The use of molecular markers may serve to direct crossings, confirm new hybrids and identify new genotypes for commercial purposes. In that context, this research aimed to analyze the genetic diversity among cultivars and hybrids of Brachiaria spp. and P. maximum using molecular markers of the type RAPD (Random amplified polymorphic DNA). It was analyzed 22 genotypes with 10 primers, which amplified 178 DNA polymorphic fragments, which were used to estimate the similarity using Jaccard coefficient. The values of similarity ranged from 0.066 to 0.841. The genetic structure among genotypes was estimated by UPGMA (Unweighted pair-group method with arithmetical average) and revealed three distinct groups with high bootstrap values (>89%). The results showed that the RAPD is a fast, relatively inexpensive and useful technique for genetic divergence characterization between different cultivars and hybrids of Brachiaria spp. and P. maximum.
Key words:Brachiaria spp., forages, molecular markers, Panicum maximum, RAPD.
INTRODUÇÃO
O Brasil ocupa o primeiro lugar no ranking mundial de exportações de carne bovina e detém o maior rebanho comercial de bovinos do mundo (ANUALPEC, 2008). A criação animal em pasto é, sem dúvida, uma das maiores responsáveis pelo sucesso atingido no setor, pois promoveu um diferencial qualitativo para a carne brasileira no mercado internacional, principalmente em função de barreiras sanitárias (como, por exemplo, a Encefalopatia Espongiforme Bovina - BSE).
Estima-se que a área coberta por pastagens no Brasil alcance cerca de 170 milhões de hectares, dos quais 135 milhões são de pastagens cultivadas, principalmente gramíneas dos gêneros Brachiaria e Panicum (DIAS-FILHO & ANDRADE, 2005). Apesar da incontestável importância dessas gramíneas forrageiras tropicais para a sustentabilidade da pecuária bovina nacional, há poucas cultivares disponíveis no mercado, as quais cobrem extensas áreas e formam uma espécie de "monocultivo". Desse modo, problemas como o aparecimento de pragas ou doenças podem colocar em risco todo o sistema de produção, como ocorreu com a síndrome da morte do capim braquiarão (B. brizantha cv. 'Marandu') na Amazônia Legal, em 2006.
O programa de melhoramento genético de gramíneas forrageiras dos gêneros Brachiaria e Panicum da Embrapa Gado de Corte vem trabalhando no desenvolvimento de cultivares forrageiras, visando à redução da vulnerabilidade dos sistemas de produção de gado pela liberação de variedades de Brachiaria e Panicum maximum que apresentem boa produtividade e alto desempenho animal, minimizando também a necessidade de abertura de novas áreas de cultivo.
Os marcadores moleculares têm sido utilizados extensivamente como ferramenta para conservação de recursos genéticos vegetais e para fornecer informações para o melhoramento das plantas (ROUT & MOHAPATRA, 2006). Dentre essas técnicas, o RAPD (Polimorfismos de DNA amplificados ao acaso) (WILLIANS et al., 1990), que utiliza um primer de sequência arbitrária para amplificar simultaneamente fragmentos de DNA, tem sido uma das mais utilizadas para estudos de diversidade genética em genomas anônimos (DIAS et al., 2004).
Considerando a importância dessas gramíneas forrageiras tropicais e as poucas informações genético-moleculares disponíveis na literatura, objetivou-se neste trabalho estimar a diversidade genética nas principais cultivares e em acessos e híbridos elite do programa de melhoramento genético da Embrapa Gado de Corte, usando marcadores moleculares do tipo RAPD.
MATERIAL E MÉTODOS
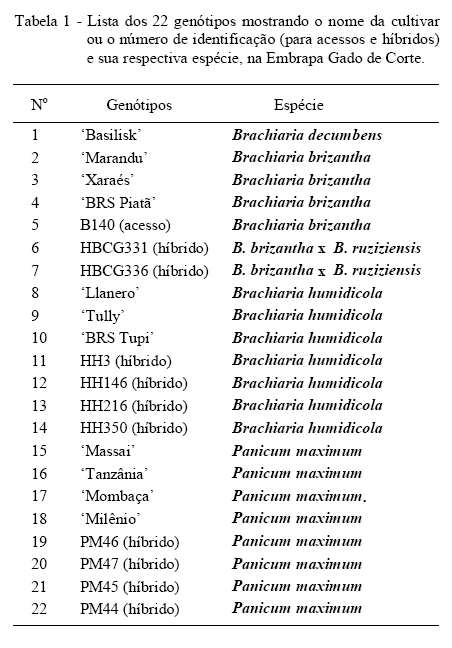
Foram avaliados neste trabalho 22 genótipos incluindo cultivares, acessos e híbridos de Brachiaria e P. maximum pertencentes ao banco de germoplasma e ao programa de melhoramento genético da Embrapa Gado de Corte, Campo Grande, MS (Tabela 1).
O DNA foi extraído de folhas jovens e frescas pelo método CTAB modificado para espécies de Brachiaria e Panicum (BONATO et. al., 2002). As reações de polimerase em cadeia (PCR) foram feitas em 20µL contendo 1x tampão de PCR, 3mM MgCl2, 100mM de cada dNTPs, 0,4mM de primer, 30ng de DNA genômico e uma unidade de Taq DNA polimerase (Invitrogen). Dez primers randômicos foram usados. Foi utilizado um termociclador PTC-100 (MJ Research) programado para 40 ciclos de 1min a 94°C, 1min a 35°C, 2min a 72°C, com uma desnaturação inicial de 5min a 94°C e uma extensão final de 7min a 72°C.
Os produtos amplificados foram analisados em gel de agarose 1,5% contendo 0,5mg mL-1 de brometo de etídio para coloração do DNA. A eletroforese foi realizada com tampão TBE 1x (90mM Tris-HBO3, 2mM EDTA, pH 8.0) a 100 Volts por três horas. O gel foi fotografado sob luz UV em sistema digital L.Pix Image (Loccus Biotechnology).
O escore dos fragmentos de RAPD foi feito considerando os dados como marcadores binários, presença "1" e ausência "0" do marcador. Cada fragmento foi considerado um alelo de um loco. A similaridade genética foi estimada através do coeficiente de Jaccard. A análise de agrupamento foi feita pelo método UPGMA (Unweighted Pair-group Method with Arithmetical Average) e, como suporte estatístico para os resultados, foi utilizada a análise de bootstrap com 10.000 interações. Essas análises foram realizadas pelo programa GENES (CRUZ, 2001), versão 2008.
RESULTADOS E DISCUSSÃO
Os 10 primers de RAPD usados amplificaram bem em todos os genótipos, independentemente da espécie. Nas espécies do gênero Brachiaria, foram amplificados 152 fragmentos de RAPD, enquanto em P. maximum foram amplificados 113. As análises foram feitas utilizando os dados de todos os genótipos, gerando um total de 182 fragmentos de DNA amplificados, dos quais apenas quatro (2,2%) foram não-informativos (monomórficos) e 178 foram informativos (polimórficos). Em média, cada primer gerou 17,8 fragmentos polimórficos. Os primers OPE03, OPAB01 e OP93 foram os que mais se destacaram, pois amplificaram mais de 20 fragmentos de DNA polimórficos cada, enquanto os primers OPP14, OPM05 e OPAL03 amplificaram 13, 14 e 14 fragmentos polimórficos, respectivamente (Tabela 2).
AMBIEL et al. (2008) obtiveram uma média de 10,2 fragmentos polimórficos por primer de RAPD estudando a variabilidade genética em acessos e cultivares comerciais de quatro espécies de Brachiaria (B. arrecta, B. brizantha, B. decumbens e B. ruziziensis) e duas cultivares de P. maximum (Mombaça e Tanzânia).
Pela análise do perfil eletroforético gerado pelo primer OP93, foi possível discriminar todas as cultivares estudadas, mostrando a eficiência dessa técnica na detecção de polimorfismos nessas espécies forrageiras (Figura 1). Entre os genótipos de Brachiaria, os perfis mais similares foram os da cultivar BRS Tupi de B. humidicola e seus híbridos HH3, HH146, HH216, HH350. Entre os genótipos de P. maximum analisados, a cultivar Massai foi a que apresentou o perfil mais distinto dos demais. Essa cultivar é um híbrido interespecífico espontâneo entre P. maximum e P. infestum (EMBRAPA GADO DE CORTE, 2001).
Entre os genótipos de Brachiaria avaliados, a similaridade genética variou de 0,07 a 0,84, denotando uma alta variabilidade genética. Os genótipos mais similares foram os híbridos HH3 e HH146, obtidos do cruzamento entre a cultivar 'BRS Tupi' (apomítica) e a planta H31 (sexual) do germoplasma da Embrapa Gado de Corte. Os genótipos menos similares foram o acesso B140 de B. brizantha e o híbrido HH216 de B. humidicola.
Para os genótipos de P. maximum, a amplitude de variação nos coeficientes de similaridade foi um pouco menor, 0,21 a 0,71. Os genótipos mais similares foram as cultivares Mombaça e Tanzânia e os menos similares foram o híbrido PM46 e a cultivar 'Massai'.
Alta variabilidade genética entre acessos e cultivares destes dois gêneros aqui estudados foi previamente reportada por outros autores. CHIARI et al. (2008) encontraram uma variação de 0,49 a 0,87 entre os coeficientes de similaridade genética obtidos para 14 genótipos de quatro espécies de Brachiaria, que incluíram as cultivares 'Marandu', 'BRS Piatã', 'Basilisk' e 'BRS Tupi'. Esse estudo foi realizado usando 396 marcadores RAPD obtidos pela análise de 47 primers.
BONATO et al. (2003) utilizaram 79 marcadores RAPD polimórficos para estimar a variabilidade genética em 24 genótipos de P. maximum, incluindo as cultivares 'Massai', 'Mombaça' e 'Tanzânia', e obtiveram valores de similaridade que variaram de 0,15 a 0,97.
AMBIEL et al. (2008) usaram 10 primers de RAPD, que amplificaram 102 marcadores, para estabelecer o agrupamento entre 40 genótipos de Brachiaria, incluindo as cultivares 'Marandu' e 'Xaraés'; e dois genótipos de P. maximum, cultivares 'Tanzânia' e 'Mombaça'. Os coeficientes de similaridade genética variaram de 0,24 a 0,83, sendo que 'Tanzânia' e 'Mombaça' foram os genótipos mais similares.
Com relação aos híbridos avaliados neste estudo, aqueles resultantes de cruzamentos interespecíficos, B. brizantha x B. ruziziensis, eles apresentaram menor coeficiente de similaridade genética (0,69) comparando aos híbridos intraespecíficos de B. humidicola, nos quais a similaridade variou de 0,70 a 0,84. Todos os híbridos de B. humidicola analisados foram obtidos do mesmo cruzamento entre a cultivar 'BRS Tupi' e a planta sexual H31, logo, são irmãos-completos, diferentemente dos híbridos interespecíficos.
Entre os híbridos intraespecíficos de P. maximum, o coeficiente de similaridade genética variou de 0,38 a 0,64, denotando maior variabilidade genética que entre os híbridos de Brachiaria. Os híbridos mais similares foram o PM45 e PM47, ambos originados do mesmo parental materno, portanto, meio-irmãos; e os mais divergentes foram PM45 e PM44, obtidos de diferentes parentais.
Em outras espécies do gênero Panicum, marcadores RAPD também foram utilizados para estudos de variabilidade genética. M'RIBU e HILU (1994) estimaram valores de similaridade que variaram de 0,60 a 1,0, utilizando o coeficiente de Dice. GUNTER et al. (1996) avaliaram 14 populações de P. virgatum e as similaridades entre elas, obtidas pelo coeficiente de Dice, variaram de 0,53 a 0,78. Nesses dois trabalhos, a variabilidade genética foi menor que a descrita aqui para P. maximum.
No dendrograma obtido pelo método UPGMA, três grupos podem ser destacados: Grupo I, que compreende todos os genótipos de B. humidicola; Grupo II, com todos os genótipos de P. maximum; Grupo III, contendo os genótipos de B. brizantha mais a cultivar B. decumbens cv. 'Basilisk'. A análise de bootstrap revelou uma consistência de 100%, 89% e 100%, respectivamente, para esses grupos (Figura 2).
Um subgrupo distinto pode ser encontrado dentro do Grupo I, que compreende a culvivar BRS Tupi e os híbridos HH3, HH146, HH216 e HH350, suportado por um alto valor de consistência (98%). Este resultado concorda com os dados de parentesco desses genótipos, pois todos os híbridos são irmãos completos originados do cruzamento entre a cultivar 'BRS Tupi' e a planta sexual H31.
Dentro do Grupo II, um subgrupo consistente a um nível de 82% se destaca, contendo os híbridos PM46, PM47 e PM45 e as cultivares 'Tanzânia', 'Mombaça' e 'Milênio'. Se o híbrido PM44 for incluído, esse valor de confiabilidade cai para 77%. Os híbridos PM46 e PM47 são irmãos-completos e têm o mesmo parental materno do PM45 sendo, portanto, meio-irmãos desse último. Já o híbrido PM44, originou-se de parentais diferentes dos demais, justificando este resultado. Como já foi mencionado, a cultivar 'Massai' que é a mais divergente dos demais genótipos é um híbrido interespecífico espontâneo.
No Grupo III, a cultivar 'Basilisk' de B. decumbens ficou agrupada com os genótipos de B. brizantha. No estudo de AMBIEL et al. (2008), a cultivar 'Basilisk' também foi agrupada com os acessos e cultivares de B. brizantha. Esses resultados corroboram as observações de REINVOIZE et al. (1996) de que a cultivar 'Basilisk', amplamente utilizada e comumente identificada como B. decumbens é na verdade uma B. brizantha. No entanto, um subgrupo contendo todos os genótipos de B. brizantha pode ser considerado com um valor de bootstrap de 73%, separando a cultivar 'Basilisk'.
CONCLUSÃO
Com os resultados obtidos neste trabalho, pode-se concluir que: 1) todos os genótipos estudados são molecularmente divergentes e que os marcadores RAPD utilizados podem auxiliar na identificação de cada genótipo; 2) existe diferença no grau de similaridade genética entre e dentro das quatro espécies estudadas, e esses dados podem orientar os melhoristas na tomada de decisão ao longo do programa de melhoramento genético dessas forrageiras; 3) o agrupamento dos genótipos corroborou os dados de pedigree existentes.
AGRADECIMENTOS
À assistente de laboratório Gisele Olivas de Campos Leguizamón, pelo auxílio prestado no desenvolvimento deste trabalho. Ao Conselho Nacional de Desenvolvimento Científico e Tecnológico (CNPq), pela bolsa concedida à primeira autora e à Associação para Fomento à Pesquisa de Melhoramento de Forrageiras Tropicais (UNIPASTO), pelo apoio financeiro.
Recebido para publicação 04.03.10
Aprovado em 01.09.11
Devolvido pelo autor 23.09.11
CR-3214
- AMBIEL, A.C. et al. Agrupamento de acessos e cultivares de três espécies de Brachiaria por RAPD. Acta Scientiarum Agronomy, v.30, n.4, p.457-464, 2008. Disponível em: <http://periodicos.uem.br/ojs/index.php/ActaSciAgron/article/view/5298/5298>. Acesso em: 26 fev. 2010. doi: 10.4025/actasciagron.v30i4.5298.
- ANUALPEC. Anuário da pecuária brasileira São Paulo: IFNP, 2008. 380p.
- BONATO, A.L.V. et al. Extração de DNA genômico de Brachiaria e Panicum maximum Campo Grande, MS: Embrapa Gado de Corte, 2002. 4p. (Embrapa Gado de Corte. Comunicado Técnico, 79). Disponível em: <http://www.cnpgc.embrapa.br/index.php?pagina=publicacoes/cot/index.html>. Acesso em: 26 fev. 2010.
- BONATO, A.L.V. et al. Similaridade genética entre acessos de Panicum maximum Jacq. determinada por marcadores RAPD. In: CONGRESSO BRASILEIRO DE MELHORAMENTO DE PLANTAS, 2., 2003, Porto Seguro, BA. Anais... Porto Seguro: SBMP, 2003. 6p. 1 CD-ROM.
- CHIARI, L. et al. Variabilidade genética em acessos e cultivares de quatro espécies de Brachiaria estimada por marcadores RAPD Campo Grande, MS: Embrapa Gado de Corte, 2008. 20p. (Embrapa Gado de Corte. Boletim de Pesquisa e Desenvolvimento, 24).
- CRUZ, C.D. Programa genes: aplicativos computacional em genética e estatística. Viçosa: UFV, 2001. 648p.
- DIAS, L.A.S. et al. A priori choice of hybrid parents in plants. Genetics Molecular Research, Ribeirão Preto, v.3, n.3, p.356-368, 2004. Disponível em: <http://www.funpecrp.com.br/gmr/year2004/vol3-3/gmr0087_full_text.htm>. Acesso em: 26 fev. 2010.
- DIAS-FILHO, M.B.; ANDRADE, C.M.S. Pastagens no ecossistema do Trópico Úmido. In: REUNIÃO ANUAL DA SOCIEDADE BRASILEIRA DE ZOOTECNIA, 2005, Goiânia, GO. Anais... Goiânia: SBZ, 2005. p.95-104.
- EMBRAPA GADO DE CORTE. Capim-massai (Panicum maximum cv. Massai): alternativa para diversificação de pastagens Campo Grande, MS: Embrapa Gado de Corte, 2001. 5p. (Embrapa Gado de Corte. Comunicado Técnico, 69). Disponível em: <http://www.cnpgc.embrapa.br/index.php?pagina=publicacoes/cot/index.html>. Acesso em: 26 fev. 2010.
- GUNTER, L.E. et al. Diversity of populations of switch grass based on RAPD markers. Crop Science, v.36, p.1017-1022, 1996.
- M'RIBU, H.K.; HILU, K.W. Detection of interspecific and intraspecific variation in Panicum millets through random amplified polymorphic DNA. Theoretical Applied of Genetics, v.88, p.412-416, 1994.
- RENVOIZE, S.A. et al. Morphology, taxonomy, and natural distribution of Brachiaria (Trin.) Griseb. In: MILES, J.W. et al. (Ed.). Brachiaria: biology, agronomy and improvement Cali: Embrapa/CIAT, 1996. p.1-15.
- ROUT, G.R.; MOHAPATRA, A. Use of molecular markers in ornamental plants: a critical reappraisal. European Journal of Horticultural Science, Stuttgart, v.71, n.2, p.53-68, 2006.
- WILLIANS, J.G.K. et al. DNA polymorphisms amplified by arbitrary primers useful as genetic markers. Nucleic Acids Research, v.18, n.22, p.6531-6535, 1990.
Datas de Publicação
-
Publicação nesta coleção
21 Nov 2011 -
Data do Fascículo
Nov 2011
Histórico
-
Aceito
01 Set 2011 -
Recebido
04 Mar 2010