Resumos
Taxonomicamente, Stylosanthes guianensis está dividida em quatro variedades botânicas: var. guianensis, var. pauciflora, var. canescens e var. microcephala. A cultivar 'BRS Bela' é uma das duas cultivares dessa leguminosa forrageira registradas no Brasil e é composta por uma mistura física de sementes de quatro acessos pertencentes a variedades botânicas ainda desconhecidas. O objetivo deste trabalho foi caracterizar a variabilidade genética entre os quatro acessos que compõem esta cultivar e determinar suas inter-relações com acessos de variedades botânicas conhecidas, usando a técnica de polimorfismos de DNA amplificados ao acaso (RAPD). Foram analisados 10 primers decâmeros em 36 acessos, do banco de germoplasma da Embrapa gado de Corte, de S. guianensis: 14 da variedade botânica pauciflora, 11 da var. guianensis, quatro da var. canescens, três da var. microcephala e os quatro acessos da cultivar 'BRS Bela'. As bandas amplificadas foram analisadas como dados binários e uma matriz de similaridade genética foi gerada, usando coeficiente de Jaccard. Com base na dissimilaridade genética, os acessos foram agrupados pelos métodos de ligação média entre grupos (UPGMA) e Tocher. A média de similaridade genética dentro das variedades botânicas foi de 0,72 para var. pauciflora, 0,63 para var. microcephala, 0,62 para var. canescens e 0,46 para var. guianensis. Entre os quatro acessos da cultivar 'BRS Bela', esta média foi de 0,62. Ambos os métodos de agrupamento geraram resultados similares e tenderam a agrupar os acessos da mesma variedade botânica. Os acessos da cultivar 'BRS Bela' foram agrupados com acessos var. guianensis. Os resultados mostram que há variabilidade genética dentro das variedades botânicas de S. guianensis e entre os acessos da cultivar 'BRS Bela' e que existe uma tendência ao agrupamento por variedade botânica.
agrupamento; marcadores moleculares; Stylosanthes; variabilidade genética
Taxonomically Stylosanthes guianensis is divided in four botanical varieties: var. guianensis, var. pauciflora, var. canescens and var. microcephala. The 'BRS Bela' cultivar is one of the two cultivars of this forage legume registered on Brazil, it is made up of a physical mixture of seeds of four accessions of unknown botanical varieties. The objective in this research was to characterize the genetic variability among four accessions of the cultivar 'BRS Bela' and to determine its genetic relationship with others accessions of known botanical varieties, using the random amplified polymorphic DNA (RAPD) technique. Ten decamer primers were evaluated in 36 accessions, from the germoplasm bank of Embrapa Beef Cattle, of S. guianensis: 14 of the botanical variety pauciflora, 11 of var. guianensis, four of var. canescens, three of var. microcephala and the four accessions of cultivar 'BRS Bela'. The amplified bands were analyzed as binary data and a matrix of genetic similarity was generated using the coefficient of Jaccard. Genetic dissimilarity data were utilized for clustering the accessions by not weighted pair-group method with arithmetical average (UPGMA) and Tocher methods. The mean genetic similarity within of the botanic varieties was of 0.72 for var. pauciflora, 0.63 for var. microcephala, 0.62 for var. canescens and 0.46 for var. guianensis. Among the four accessions of cultivar 'BRS Bela' this mean was 0.62. Both methods of clustering generated similar results and showed a tendency to cluster the accessions of the same botanical variety. The accessions of cultivar 'BRS Bela' were grouped with accessions of botanical variety guianensis. The results show that there is genetic variability within of the botanical varieties of S. guianensis and among the accessions of the cultivar 'BRS Bela' and that there is a tendency to the clustering by botanical variety.
clustering; genetic variability; molecular markers; Stylosanthes
ARTIGOS CIENTÍFICOS
PRODUÇÃO ANIMAL
Variabilidade genética de acessos da cultivar BRS Bela de Stylosanthes guianensis usando marcadores moleculares RAPD
Genetic variability of accessions of BRS Bela cultivar of Stylosanthes guianensis using RAPD markers
Elizangela Tieko MatidaI; Lucimara ChiariII,1; Rosangela Maria SimeãoII; Celso Dornelas FernandesII; Edihanne Gamarra ArguelhoIII; Diego Pazinato BothIV
IUniversidade Federal de Mato Grosso do Sul (UFMS), Campo Grande, MS, Brasil
IIEmbrapa Gado de Corte, Avenida Rádio Maia, 830, Vila Popular, CP 154, 79106-550, Campo Grande, MS, Brasil. E-mail: lchiari@cnpgc.embrapa.br
IIIUniversidade Estadual de Londrina (UEL), Londrina, PR, Brasil
IVUniversidade Católica Dom Bosco (UCDB), Campo Grande, MS, Brasil
RESUMO
Taxonomicamente, Stylosanthes guianensis está dividida em quatro variedades botânicas: var. guianensis, var. pauciflora, var. canescens e var. microcephala. A cultivar 'BRS Bela' é uma das duas cultivares dessa leguminosa forrageira registradas no Brasil e é composta por uma mistura física de sementes de quatro acessos pertencentes a variedades botânicas ainda desconhecidas. O objetivo deste trabalho foi caracterizar a variabilidade genética entre os quatro acessos que compõem esta cultivar e determinar suas inter-relações com acessos de variedades botânicas conhecidas, usando a técnica de polimorfismos de DNA amplificados ao acaso (RAPD). Foram analisados 10 primers decâmeros em 36 acessos, do banco de germoplasma da Embrapa gado de Corte, de S. guianensis: 14 da variedade botânica pauciflora, 11 da var. guianensis, quatro da var. canescens, três da var. microcephala e os quatro acessos da cultivar 'BRS Bela'. As bandas amplificadas foram analisadas como dados binários e uma matriz de similaridade genética foi gerada, usando coeficiente de Jaccard. Com base na dissimilaridade genética, os acessos foram agrupados pelos métodos de ligação média entre grupos (UPGMA) e Tocher. A média de similaridade genética dentro das variedades botânicas foi de 0,72 para var. pauciflora, 0,63 para var. microcephala, 0,62 para var. canescens e 0,46 para var. guianensis. Entre os quatro acessos da cultivar 'BRS Bela', esta média foi de 0,62. Ambos os métodos de agrupamento geraram resultados similares e tenderam a agrupar os acessos da mesma variedade botânica. Os acessos da cultivar 'BRS Bela' foram agrupados com acessos var. guianensis. Os resultados mostram que há variabilidade genética dentro das variedades botânicas de S. guianensis e entre os acessos da cultivar 'BRS Bela' e que existe uma tendência ao agrupamento por variedade botânica.
Palavras-chave: agrupamento, marcadores moleculares, Stylosanthes, variabilidade genética.
ABSTRACT
Taxonomically Stylosanthes guianensis is divided in four botanical varieties: var. guianensis, var. pauciflora, var. canescens and var. microcephala. The 'BRS Bela' cultivar is one of the two cultivars of this forage legume registered on Brazil, it is made up of a physical mixture of seeds of four accessions of unknown botanical varieties. The objective in this research was to characterize the genetic variability among four accessions of the cultivar 'BRS Bela' and to determine its genetic relationship with others accessions of known botanical varieties, using the random amplified polymorphic DNA (RAPD) technique. Ten decamer primers were evaluated in 36 accessions, from the germoplasm bank of Embrapa Beef Cattle, of S. guianensis: 14 of the botanical variety pauciflora, 11 of var. guianensis, four of var. canescens, three of var. microcephala and the four accessions of cultivar 'BRS Bela'. The amplified bands were analyzed as binary data and a matrix of genetic similarity was generated using the coefficient of Jaccard. Genetic dissimilarity data were utilized for clustering the accessions by not weighted pair-group method with arithmetical average (UPGMA) and Tocher methods. The mean genetic similarity within of the botanic varieties was of 0.72 for var. pauciflora, 0.63 for var. microcephala, 0.62 for var. canescens and 0.46 for var. guianensis. Among the four accessions of cultivar 'BRS Bela' this mean was 0.62. Both methods of clustering generated similar results and showed a tendency to cluster the accessions of the same botanical variety. The accessions of cultivar 'BRS Bela' were grouped with accessions of botanical variety guianensis. The results show that there is genetic variability within of the botanical varieties of S. guianensis and among the accessions of the cultivar 'BRS Bela' and that there is a tendency to the clustering by botanical variety.
Key words: clustering, genetic variability, molecular markers, Stylosanthes.
INTRODUÇÃO
O gênero Stylosanthes, pertencente à família Leguminosae, inclui cerca de 50 espécies descritas (LEWIS et al., 2005) e um grande número de subespécies e de variedades botânicas descritas (FERREIRA & COSTA, 1979). A maioria dessas espécies é perene, com vigoroso sistema radicular pivotante, tolerante à seca e de grande capacidade colonizadora por sua adaptação a solos de baixa fertilidade e por apresentar simbiose com bactérias fixadoras de nitrogênio (ANDRADE et al., 2004).
Dentre as diversas espécies desse gênero com potencial de uso no Cerrado, destaca-se S. guianensis, que pode ser usada tanto em pastagens consorciadas com gramíneas quanto em bancos de proteína (COELHO et al., 2006). Essa espécie divide-se em quatro variedades botânicas: var. guianensis, var. canescens, var. microcephala e var. pauciflora (FERREIRA & COSTA, 1979). As duas primeiras com maior produção de matéria seca e retenção de folhas verdes durante a estação seca em solos de baixa fertilidade. É a espécie desse gênero com a mais ampla distribuição geográfica, ocorrendo desde o México até a Argentina e, consequentemente, é a que apresenta maior diversidade fenotípica (ANDRADE et al., 2004).
No Brasil, somente duas cultivares de S. guianensis foram registradas: S. guianensis cv. 'Mineirão' (EMBRAPA CERRADOS, 1998) e S. guianensis cv. 'BRS Bela' (BRASIL, 2007). Essa última, ainda não lançada comercialmente, é formada por uma mistura física de sementes de quatro acessos (GC 1; GC 2; GC 3 e GC 4), sendo 25% de sementes de cada um. Os acessos GC 1 e GC 3 foram reintroduzidos das Filipinas e, possivelmente, são híbridos artificiais, entretanto, sem dados completos de passaporte. O GC 4 foi selecionado a partir do acesso GC 3 e o acesso GC 2 é originário da Colômbia (CIAT, 2007).
A cultivar 'BRS Bela' foi avaliada em ensaios regionais, em quatro locais: Chapadão do Sul, MS; Sete Lagoas, MG; Goiânia, GO; e Planaltina, DF, e destacou-se pela produtividade de matéria seca, pela elevada resistência à antracnose e, principalmente, pela maior produtividade de sementes quando comparada à cultivar 'Mineirão' (FERNANDES et al., 2004).
O emprego de chave para identificação das variedades botânicas de S. guianensis componentes da cultivar 'BRS Bela', com base em COSTA (2006), não foi conclusivo. Os acessos apresentaram características morfológicas intermediárias entre as variedades botânicas. Pela possibilidade de três dos acessos terem se originado por meio de hibridação artificial ou seleção intra-populacional e como não há informações sobre os genitores utilizados, outras formas de elucidação para a questão taxonômica precisam ser buscadas.
Os marcadores moleculares apresentam inúmeras possibilidades de estudo em espécies do gênero Stylosanthes, como a geração de informações sobre a variabilidade genética interespecífica (KAZAN et al., 1993) e intra-específica (CHIARI et al., 2006; KARIA, 2008), e estimativas de taxa de cruzamento (CHIARI et al., 2010) entre outras.
Dentre os marcadores moleculares, destacam-se os marcadores RAPD (Random Amplified Polymorphic DNA) pela facilidade de execução, número de polimorfismos gerados, baixo custo das análises e, principalmente, por permitirem a análise de genomas desconhecidos (FERREIRA & GRATAPAGLIA, 1996).
O objetivo deste trabalho foi analisar a variabilidade genética entre os quatro acessos que compõem esta cultivar e determinar suas inter-relações com acessos de variedades botânicas conhecidas, usando a técnica de RAPD.
MATERIAL E MÉTODOS
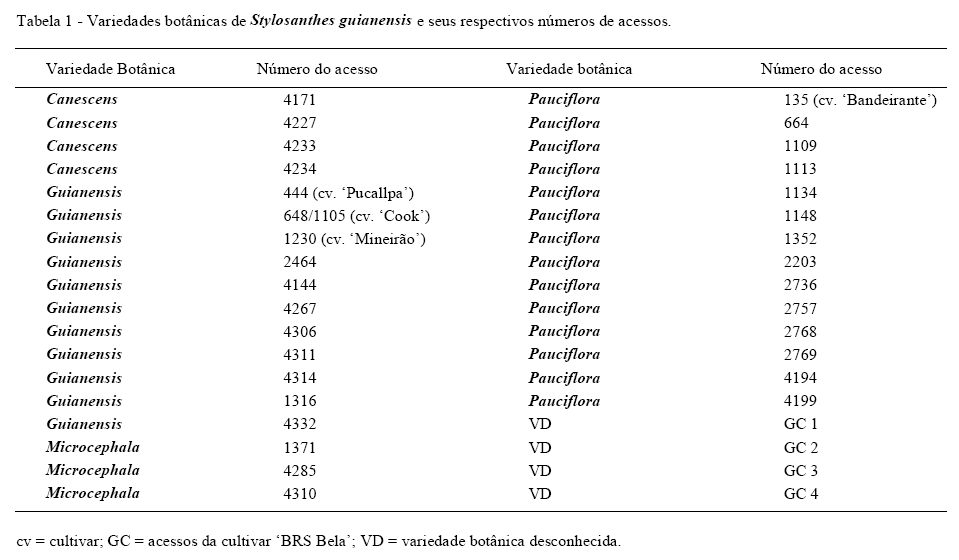
Foram analisados 36 acessos de S. guianensis, do germoplasma da Embrapa Gado de Corte, Campo Grande, MS, Brasil: 14 da variedade botânica (var.) pauciflora, 11 var. guianensis, quatro var. canescens, três var. microcephala e os quatro acessos que compõem a cultivar 'BRS Bela' (Tabela 1). Os acessos foram clonados por estaquia e mantidos em casa de vegetação na Embrapa Gado de Corte.
O DNA foi extraído de folhas jovens seguindo a metodologia de BONATO et al. (2002), ajustada para esta espécie por CHIARI et al. (2009). O DNA extraído foi quantificado por comparação com padrões do lambda (Invitrogen) de concentrações conhecidas em ng µL-1 (500, 300 e 100) em gel de agarose 0,8%, corado com brometo de etídio (0,5µg mL-1) e visualizado em transluminador sob luz Ultra-Violeta (UV). A fotodocumentação foi feita em sistema digital LPix (Loccus Biotecnologia). As amostras foram diluídas para a concentração 5ng.µL-1 e mantidas a 4oC para serem utilizadas nas reações de RAPD. Os estoques foram congelados a -20°C.
Para as análises moleculares, foram utilizados dez primers previamente selecionados em S. guianensis (CHIARI et al., 2006; CHIARI et al., 2010). As reações de RAPD foram preparadas em volume final de 25µL contendo: 1x tampão da Taq DNA polimerase (Invitrogen), 2mM de MgCl2 (Invitrogen), 1,5U de Taq DNA polimerase (Invitrogen), 2,5mM de cada dNTP (Invitrogen), 4% DMSO (Sigma), 1,2µM de primer e 20ng de DNA. Para todas as reações, foi feito um controle negativo, isto é, sem a adição de DNA, para fornecer maior confiabilidade às amplificações obtidas. As amplificações foram realizadas em termociclador PTC 100 (MJ Research), com desnaturação inicial a 94°C durante cinco minutos, seguida de 40 ciclos de 94°C por um minuto, 40°C por 1,5 minutos e 72°C por dois minutos e, para finalizar uma etapa de extensão, a 72°C por sete minutos.
Os produtos da PCR foram submetidos à eletroforese em gel de agarose 1,5% em tampão TBE 1x (0,89M Tris; 0,89M borato e 0,08M EDTA). Os géis foram corados com brometo de etídio (0,5µg mL-1) e visualizados em transluminador sob luz UV. A fotodocumentação foi feita em sistema digital LPix (Loccus Biotecnologia).
Os perfis de RAPD foram determinados pela observação direta dos fragmentos de DNA amplificados (bandas) nos acessos a partir de cada um dos primers. Os dados foram analisados como variáveis binárias ("1" para presença e "0" para ausência). Uma matriz de similaridade genética foi obtida usando o coeficiente de similaridade de Jaccard (S). O agrupamento dos acessos foi realizado a partir dos dados de dissimilaridade genética (D=1-S), usando o método de ligação média entre grupos (UPGMA). Para essas análises, foi utilizado o programa computacional GENES (CRUZ, 2001), versão 2008 para Windows.
RESULTADOS E DISCUSSÃO
Os dez primers analisados amplificaram 99 bandas, das quais apenas cinco foram monomórficas. O número de bandas analisadas por primer variou de três a 15, sendo que o primer G07 foi o que amplificou o menor número e o AN10 o maior número. A quantidade de bandas polimórficas amplificadas por primer também variou bastante, de duas a 15, também para os primers G07 e AN10 (Tabela 2). Essas bandas apresentaram nitidez e facilidade de análise. Todos os indivíduos foram analisados no mesmo gel. Além disso, uma reação de controle negativo (branco) foi utilizada para garantir a confiabilidade dos resultados.
Os valores de similaridade genética variaram de 0,28 a 0,90, considerando os acessos de todas as variedades botânicas e os quatro acessos da cultivar 'BRS Bela'. O menor valor foi obtido para os acessos 4144 e GC 1 e o maior para 135 e 648. A média de similaridade genética dentro da mesma variedade botânica foi 0,72 para os acessos da var. pauciflora, 0,63 para var. microcephala, 0,62 para var. canescens e 0,46 para var. guianensis. Entre os quatro acessos da cultivar 'BRS Bela', a média de similaridade foi igual a 0,62 (Tabela 3).
Um dendrograma foi gerado pelo método UPGMA, utilizando os dados de dissimilaridade. Nove grupos podem ser visualizados, nos quais o número de acessos variou de 1 a 12, mostrando uma tendência no agrupamento pela variedade botânica (Figura 1).
FALEIRO et al. (2007) também utilizaram a técnica de RAPD para estimar a variabilidade genética em S. guianensis, estudando outros acessos dessas mesmas variedades botânicas. Nesse trabalho, os valores de similaridade genética variaram de 0,46 a 0,96 e também observou-se uma tendência de agrupamento de acessos que pertencem à mesma variedade botânica, o que reforça a importância desses marcadores moleculares como ferramenta de auxílio na classificação taxonômica de acessos dessa espécie.
Usando sete marcadores microssatélites (SSR) desenvolvidos por VANDER STAPPEN et al. (1999), KARIA (2008) estudou a diversidade genética de acessos S. guianensis, pertencentes ao germoplasma da Embrapa Cerrados, e também observou uma tendência dos acessos a se agruparem de acordo com as variedades botânicas descritas por FERREIRA & COSTA (1979), além disso observou correlação significativa entre os dados moleculares e morfoagronômicos, porém de baixa magnitude.
LAZAROTTO-FORMAGINI et al. (2006) analisaram a variabilidade genética em alguns dos acessos analisados neste trabalho, utilizando caracteres morfoagronômicos. O dendrograma gerado pela distância genética de Mahalanobis não mostrou a tendência de agrupamento por variedades botânicas.
Pelo que foi apresentado, para S. guianensis, os marcadores moleculares mostraram-se mais eficientes que os morfoagronômicos na discriminação de grupos, de acordo com a classificação taxonômica.
CONCLUSÃO
Os resultados obtidos mostram considerável variabilidade genética entre os acessos das diferentes variedades botânicas de S. guianensis avaliados, assim como dentro dessas variedades e entre os acessos que formam a cultivar 'BRS Bela'. Também revelam uma tendência de agrupamento dos acessos de acordo com a variedade botânica e sugerem que os acessos da cultivar 'BRS Bela' sejam geneticamente mais próximos dos acessos da variedade botânica guianensis.
AGRADECIMENTOS
Os autores agradecem ao Conselho Nacional de Desenvolvimento Científico e Tecnológico (CNPq), pelas bolsas concedidas ao primeiro, quarto e quinto autores, e à Fundação de Apoio ao Desenvolvimento do Ensino, Ciência e Tecnologia do Estado de Mato Grosso do Sul (FUNDECT) e à Associação para o Fomento à Pesquisa de Melhoramento de Forrageiras (UNIPASTO), pelo apoio financeiro.
Recebido para publicação 01.02.11
Aprovado em 29.05.12
Devolvido pelo autor 05.10.12
CR-4679
- ANDRADE, R.P. et al. Stylosanthes as a forage legume at its centre of diversity. In: CHAKRABORTY, S. (Ed.). High-yielding anthracnose-resistant Stylosanthes for agricultural systems Canberra: Australian Centre for International Agricultural Research, 2004. Cap.3, p.37-50.
- BONATO, A.L.V. et al. Extração de DNA genômico de Stylosanthes spp Campo Grande: Embrapa Gado de Corte, 2002. 4p. (Comunicado técnico, 78).
- BRASIL. Ministério da Agricultura, Pecuária e Abastecimento. Portal do Ministério da Agricultura, Pecuária e do Abastecimento Disponível em: <http://extranet.agricultura.gov.br/php/proton/cultivarweb/detalhe_cultivar.php?codsr=20613> Online. Acesso em: 14 maio, 2010.
- CHIARI L. et al. Análise da diversidade genética em Stylosanthes guianensis utilizando marcadores RAPD Mato Grosso do Sul: Embrapa Gado de Corte, 2006. 25p. (Boletim de Pesquisa e Desenvolvimento, 20).
- CHIARI, L. et al. Comparação de três métodos de extração de DNA genômico para análises moleculares em Stylosanthes guianensis Mato Grosso do Sul: Embrapa Gado de Corte, 2009. 6p. (Circular Técnica, 36).
- CHIARI, L. et al. Mating system parameters in Stylosanthes guianensis (Aubl.) Sw. based on RAPD markers. African Journal of Biotechnology, v.9, n.36, p.5820-5822, 2010. Disponível em: <http://www.academicjournals.org/pdf/Pdf2010/6Sep/Chiari%20et%20al.pdf> Acesso em: 24 abr. 2012.
- CIAT (CENTRO INTERNACIONAL DE AGRICULTURA TROPICAL). Disponível em: <http://www.ciat.cgiar.org> Acesso em: 14 ago. 2007.
» link - COSTA, N.M.S. Revisão do género Stylosanthes Sw 2006. 470f. Tese (Doutorado em Engenharia Agronômica) - Instituto Superior de Agronômia, Universidade Técnica de Lisboa, Lisboa.
- COELHO, M.H. et al. Avaliação e caracterização de acessos de Stylosanthes guianensis quanto à produção de sementes. Pasturas Tropicales, v. 28, p.3-14, 2006. Disponível em: <http://webapp.ciat.cgiar.org/forrajes/pdf/pasturas_tropicales_2006/pt_28_3_3_14.pdf>. Acesso em: 24 abr. 2012.
- CRUZ, C.D. Programa genes - Aplicativo computacional em genética e estatística Versão Windows. Viçosa: UFV, 2001. 642p.
- EMBRAPA CERRADOS. Estabelecimento e utilização do estilosantes Mineirão Planaltina: Embrapa Cerrados, 1998. 6p. (Comunicado Técnico, 74).
- FALEIRO, F.G. et al. Utilização de marcadores moleculares na composição de amostra nuclear de Stylosanthes guianensis In: CONGRESSO BRASILEIRO DE MELHORAMENTO DE PLANTAS, 4., 2007, São Lourenço, MG. Anais... São Lourenço: SBMP, 2007. p.1-3. 1 CD-ROM.
- FERNANDES, C.D. et al. Regional evaluation of Stylosanthes germplasm in Brazil. In: CHAKRABORTY, S. High-yielding anthracnose-resistant Stylosanthes for agricultural systems Canberra, Austrália: ACIAR, 2004. Cap.10, p.127-134.
- FERREIRA, M.B.; COSTA, N.M.S. O gênero Stylosanthes Sw. no Brasil Belo Horizonte: EPAMIG, 1979. 108p.
- FERREIRA, M.E.; GRATTAPAGLIA, D. Introdução ao uso de marcadores moleculares em análise genética 2.ed. Brasília: EMBRAPA-CENARGEN, 1996. 220p.
- KARIA, C.T. Caracterização genética e morfoagronômica de germoplasma de Stylosanthes guianensis (Aubl.) SW 2008. 140f. Tese (Doutorado em Agronomia) - Curso de Pós-graduação em Agronomia, Universidade Federal de Goiás, GO.
- KAZAN, K. et al. Genetic variation in agronomically important species of Stylosanthes determined using random amplified polymorphic DNA mark. Theoretical and Applied Genetics, v.85, p.882-888, 1993.
- LAZAROTTO-FORMAGINI, E. Avaliação morfoagronômica de acessos da leguminosa forrageira Stylosanthes guianensis Aubl. (Sw.) em Mato Grosso do Sul 2006. 20f. Monografia (Curso de Ciências Biológicas) - Universidade para o Desenvolvimento do Estado e da Região do Pantanal-UNIDERP.
- LEWIS, G.L.et al. Legumes of the World Kew: Royal Botanic Gardens, 2005. 579p.
- VANDER STAPPEN, J. et al. Microsatellite markers in Stylosanthes guianensis Molecular Ecology, v.8, p.514-517, 1999.
Datas de Publicação
-
Publicação nesta coleção
09 Nov 2012 -
Data do Fascículo
Jan 2013
Histórico
-
Recebido
01 Fev 2011 -
Aceito
29 Maio 2012