Resumos
A hanseníase é doença infecciosa milenar que, apesar da existência de terapêutica eficaz, ainda persiste como problema de saúde pública em seis países, entre eles o Brasil, líder mundial em prevalência da doença. Ao longo das últimas décadas, a hanseníase vem sendo estudada por perspectiva talvez inesperada para uma doença infecciosa: modernos métodos de análise experimental têm sido empregados para evidenciar a importância do componente genético no controle da susceptibilidade do hospedeiro à hanseníase e seus fenótipos. Esses estudos indicam que constituição genética favorável do hospedeiro, somada a fatores propícios, ambientais e relativos ao agente patogênico, tem alto impacto na definição da susceptibilidade tanto à infecção propriamente dita quanto à evolução clínica da doença. Hoje, diversos genes e regiões genômicas já foram relacionados ao controle da susceptibilidade à hanseníase. Outros estudos estão em andamento, visando ao avanço no entendimento das bases moleculares de controle da susceptibilidade do hospedeiro à doença. O conjunto de resultados desses estudos pode levar a formas mais eficazes de diagnóstico, tratamento e prevenção da hanseníase e outras doenças infecciosas.
Genética; Hanseníase; Polimorfismo genético; Predisposição genética para doença
Leprosy is an ancient chronic infectious disease that, despite the existence of efficient therapy, still persists as a public health problem in six countries, including Brazil, the world leader in leprosy prevalence. During the past decades, leprosy has been studied from a somewhat unusual perspective for an infectious disease: modern methods for experimental analysis have been applied to demonstrate the existence of an important genetic effect controlling host susceptibility to leprosy and its phenotypes. These studies indicate that a favorable host genetic make-up, combined with environmental and pathogen-related variables, has a high impact on the definition of susceptibility to both infection and clinical manifestation of leprosy. To date, several genes and genomic regions have been implicated in the control of leprosy susceptibility. Additional ongoing studies aim at advancing further in the elucidation of the molecular basis of host susceptibility to the disease. This body of evidence may lead to new strategies for diagnosis, treatment and prevention of leprosy and other infectious diseases.
Genetics; Genetic predisposition to disease; Leprosy; Polymorphism, genetic
ARTIGO DE REVISÃO
Hanseníase: uma doença genética?* * Trabalho realizado na Pontifícia Universidade Católica do Paraná Curitiba (PR), Brasil. Conflito de interesse: Nenhum / Conflict of interest: None
Leprosy: a genetic disease?
Flávia Costa PrevedelloI; Marcelo Távora MiraII
IMD. Mestranda do Núcleo de Investigação Molecular Avançada (NIMA), Programa de Pós-Graduação em Ciências da Saúde da Pontifícia Universidade Católica do Paraná Curitiba (PR), Brasil
IIPhD. Professor adjunto e investigador principal do Núcleo de Investigação Molecular Avançada (NIMA), Programa Pós-Graduação em Ciências da Saúde da Pontifícia Universidade Católica do Paraná Curitiba (PR) e diretor científico da Fundação Pró-Hansen Curitiba (PR), Brasil
Endereço para correspondência Endereço para correspondência Marcelo Távora Mira Pontifícia Universidade Católica do Paraná,Centro de Ciências Biológicas e da Saúde Programa de Pós-Graduação em Ciências da Saúde Rua Imaculada Conceição 1115 80215 901 - Curitiba - PR Tel./Fax: : (41) 3271-2618 / (41) 3271-1657 E-mail: m.mira@pucpr.br
RESUMO
A hanseníase é doença infecciosa milenar que, apesar da existência de terapêutica eficaz, ainda persiste como problema de saúde pública em seis países, entre eles o Brasil, líder mundial em prevalência da doença. Ao longo das últimas décadas, a hanseníase vem sendo estudada por perspectiva talvez inesperada para uma doença infecciosa: modernos métodos de análise experimental têm sido empregados para evidenciar a importância do componente genético no controle da susceptibilidade do hospedeiro à hanseníase e seus fenótipos. Esses estudos indicam que constituição genética favorável do hospedeiro, somada a fatores propícios, ambientais e relativos ao agente patogênico, tem alto impacto na definição da susceptibilidade tanto à infecção propriamente dita quanto à evolução clínica da doença. Hoje, diversos genes e regiões genômicas já foram relacionados ao controle da susceptibilidade à hanseníase. Outros estudos estão em andamento, visando ao avanço no entendimento das bases moleculares de controle da susceptibilidade do hospedeiro à doença. O conjunto de resultados desses estudos pode levar a formas mais eficazes de diagnóstico, tratamento e prevenção da hanseníase e outras doenças infecciosas.
Palavras-chave: Genética; Hanseníase; Polimorfismo genético; Predisposição genética para doença
ABSTRACT
Leprosy is an ancient chronic infectious disease that, despite the existence of efficient therapy, still persists as a public health problem in six countries, including Brazil, the world leader in leprosy prevalence. During the past decades, leprosy has been studied from a somewhat unusual perspective for an infectious disease: modern methods for experimental analysis have been applied to demonstrate the existence of an important genetic effect controlling host susceptibility to leprosy and its phenotypes. These studies indicate that a favorable host genetic make-up, combined with environmental and pathogen-related variables, has a high impact on the definition of susceptibility to both infection and clinical manifestation of leprosy. To date, several genes and genomic regions have been implicated in the control of leprosy susceptibility. Additional ongoing studies aim at advancing further in the elucidation of the molecular basis of host susceptibility to the disease. This body of evidence may lead to new strategies for diagnosis, treatment and prevention of leprosy and other infectious diseases
Keywords: Genetics; Genetic predisposition to disease; Leprosy; Polymorphism, genetic
INTRODUÇÃO
Na história da humanidade, provavelmente nenhuma doença gerou estigma social tão intenso quanto a hanseníase, sempre associada com conceitos tais como pecado, impureza e punição. Na era pré-microbiológica, contribuía para o fortalecimento do estigma a noção de que a hanseníase era hereditária, já que agregação familiar de casos era observada com freqüência. Com a identificação do bacilo causador da doença no século XVII por Armauer Hansen, confirmou-se a natureza infecciosa da enfermidade, e muito da investigação científica passou a se concentrar em aspectos relativos ao agente patogênico e suas propriedades, tais como virulência, capacidade de indução de resposta imune e resposta a tratamento. Contudo, observações clínicas sempre indicaram que não bastava o contato com o bacilo para se desenvolver a doença, que a hanseníase ocorria com maior ou menor freqüência em diferentes populações e que a doença se manifestava de diferentes maneiras em diferentes indivíduos. Por exemplo, já em 1937, Rotberg, estudando a reação de Mitsuda, havia sugerido a possibilidade de um componente genético de controle da susceptibilidade à doença, em que pacientes que manifestassem positividade à reação apresentariam um fator natural de resistência, denominado pelo autor Fator N, de natureza desconhecida.1 Hoje, pode-se especular que o Fator N é na verdade a manifestação de um conjunto de genes, os quais, interagindo de forma complexa, resultam em diferentes formas de resposta do indivíduo ao antígeno de Mitsuda.
Hanseníase pode ser brevemente definida como doença infecciosa crônica causada pelo Mycobacterium leprae que afeta primariamente a pele e o sistema nervoso periférico. A doença ocorre naturalmente apenas em duas espécies conhecidas, os seres humanos e os tatus. O seqüenciamento completo do genoma do M. leprae revelou extensa redução evolutiva, com perda de grande número de genes em comparação com o M. tuberculosis, desde que essas espécies divergiram de um ancestral comum.2 A manutenção de um conjunto mínimo de genes pode ser a explicação para algumas características únicas do M. leprae, tais como a incapacidade de ser cultivado em meio artificial, o longo tempo de duplicação e a altíssima especificidade por seus alvos celulares, os macrófagos e as células de Schwann do sistema nervoso periférico. Recentemente, ferramentas de genômica comparativa foram aplicadas para dar suporte à hipótese de que a doença tenha tido origem na África oriental, de onde se espalhou para o resto do mundo seguindo as rotas migratórias das primeiras populações humanas.3
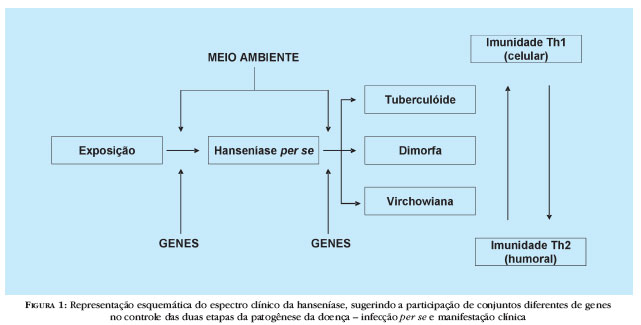
A hanseníase é doença de manifestação clínica espectral. Segundo Ridley e Jopling,4 os pólos desse espectro são ocupados de um lado pela forma mais localizada denominada tuberculóide, associada a resposta imunológica do tipo Th1 (celular), e do outro pela forma virchowiana (assim denominada no Brasil em substituição ao termo "lepromatosa" da classificação original), sistêmica, e associada a resposta imunológica do tipo Th2 (humoral), com três formas clínicas intermediárias ou borderline. Os pólos tuberculóide e virchowiano correspondem aproximadamente às formas paucibacilar e multibacilar criadas pela Organização Mundial da Saúde (OMS) para fins de orientação terapêutica.
Desde meados da década de 1980, esforço global tem sido empregado, sob orientação da OMS, com o objetivo de eliminar a hanseníase como problema de saúde pública. Originalmente, essa meta deveria ter sido atingida em 2000. No entanto, em 2007, cerca de 300.000 novos casos/ano ainda são diagnosticados, a maioria no Brasil e na Índia. Considerando-se que (i) não existe reservatório natural de importância biológica conhecido para o M. leprae; (ii) casos novos da doença são tratados por poliquimioterapia através de protocolos 100% eficazes; e (iii) os medicamentos são fornecidos gratuitamente pela OMS, a origem desses novos casos permanece desconhecida.
BREVE INTRODUÇÃO À ANÁLISE GENÉTICA DAS DOENÇAS COMPLEXAS
Em genética, doenças complexas são aquelas que não apresentam padrão de herança mendeliana clássica, em que há perfeita correlação entre genótipo e fenótipo. A ocorrência dessas doenças é controlada por componente genético, resultado da ação integrada de um número variado de genes, associado a fatores ambientais, socioeconômicos, culturais, entre outros. Tal definição conduz à conclusão de que todas as doenças comuns, entre elas as infecções, pelo menos em parte, são genéticas.
Em termos gerais, a dissecção do componente genético de doenças complexas inicia-se por meio de estudos observacionais, que buscam evidências de participação de genes contribuindo para a característica que se está estudando. Entre as estratégias adotadas estão as análises de agregação familial de casos, os estudos comparativos de taxas de concordância da ocorrência da doença entre gêmeos mono e dizigóticos, e as análises de segregação complexa, que visam descrever o modo de herança genética que melhor explica as observações em um conjunto de pedigrees. Esses estudos, entretanto, não fornecem informações sobre a exata natureza do componente genético em questão, tais como localização, número e identidade dos genes envolvidos. Para se avançar, portanto, é necessária a adoção de outros modelos de estudos, agora envolvendo marcadores genéticos moleculares, tal como é feito nas análises de ligação e associação.
A. Estudos de ligação
Estudos de ligação são estudos de mapeamento genético, cujo objetivo é localizar genes responsáveis por determinado efeito no intervalo cromossômico mais estreito possível. Esses estudos, embora poderosos no mapeamento de genes exercendo efeito genético moderado a intenso, não têm poder de resolução para implicar um único gene tipicamente, estudos de ligação terminam localizando o gene em questão em um intervalo genômico contendo dezenas de genes. O resultado desse tipo de estudo normalmente é expresso em LOD escores; um LOD escore igual ou superior a 3,0 indica significância estatística, e a região genômica encontrada passa a ser tratada como região candidata a abrigar o gene que está se procurando identificar.
B. Estudos de associação
Os estudos de associação, em seu formato mais simples, baseiam-se na comparação das freqüências alélicas de um marcador genético entre os indivíduos afetados e não afetados. Determinado alelo é considerado associado com o fenótipo estudado quando ocorre com freqüência diferente entre indivíduos afetados em comparação com um grupo controle de não afetados. Estudos de associação têm alto poder de detecção de efeitos genéticos moderados a fracos e geralmente são utilizados ou na análise de genes candidatos, ou como seqüência complementar de estudos de ligação, com o objetivo de se identificar com precisão, em região genômica candidata, o gene responsável pelo efeito detectado.
GENÉTICA DA HANSENÍASE
Na hanseníase, estratégias como as descritas acima têm sido empregadas com o objetivo de serem elucidadas as bases moleculares do controle da susceptibilidade do hospedeiro ao M. leprae. Hoje, é amplamente aceita a noção de que conjuntos diferentes de genes modificam a susceptibilidade à doença em pelo menos dois momentos distintos, a saber: (i) no controle da infecção per se, isto é , a doença independentemente de sua forma de manifestação clínica; e (ii), uma vez o indivíduo infectado, na definição da diferentes formas clínicas da doença, conforme ilustrado na figura 1.
A Primeiros estudos: estudos de gêmeos e análises de segregação complexa
A hipótese de que a hanseníase teria componente hereditário vem sendo investigada desde a Idade Média. Observações do monge franciscano Bartholomaeus Angelicus em 1246 já o levaram a postular que a transmissão da doença ocorria de pais para filhos.5 Bem mais tarde, estudo clássico de gêmeos realizado em larga escala na Índia evidenciou taxas significativamente maiores de concordância da ocorrência de hanseníase entre gêmeos monozigóticos em comparação com gêmeos dizigóticos, não só para a doença per se, mas também para suas formas clínicas.6
Em 1988, foi realizada análise de segregação complexa em uma população da ilha caribenha Desirade, envolvendo 953 indivíduos, entre eles 82 pacientes hansenianos (28 virchowianos e 54 não virchowianos). A prevalência de hanseníase na ilha era de aproximadamente 30/1.000 habitantes, uma das mais elevadas do mundo na ocasião. Os resultados do estudo rejeitaram um modelo esporádico (não familial), indicando um modelo de herança mendeliana com gene principal co-dominante ou recessivo controlando a susceptibilidade à doença.7 Mais recentemente, esse achado foi em parte confirmado em estudo envolvendo amostra de 76 pedigrees brasileiros e que também resultou na rejeição de modelo predominantemente ambiental de controle da susceptibilidade à hanseníase.8
B Genes candidatos
A escolha de genes candidatos para análise genética molecular é tarefa difícil para o geneticista, em particular frente à constatação de que o genoma humano é formado por estimados 30 a 40 mil genes. Assim, genes candidatos são tipicamente selecionados com base em três critérios: (i) possível papel crítico na patogênese da doença (candidatos funcionais); (ii) sua localização em região genômica previamente envolvida no controle da doença (candidatos posicionais); ou (iii) combinação dos dois anteriores (candidatos funcionais/posicionais). Breve descrição dos genes candidatos mais intensamente estudados em hanseníase é apresentada a seguir.
B.1 Genes ligados ao complexo do antígeno leucocitário humano (HLA)
B.1.1 HLA II
O complexo HLA é conjunto de genes localizados na região cromossômica 6q21 (Figura 2).9 Estudos relacionando genes HLA com hanseníase são suportados pela observação de que as formas clínicas da doença estão associadas ao perfil de resposta Th1/Th2 apresentado pelo hospedeiro; essa resposta seria parcialmente controlada por mecanismos de apresentação de antígenos e interação célula/célula, por sua vez dependentes da expressão de genes HLA. De fato, um grande número de estudos de ligação e associação demonstrou envolvimento de alelos e haplótipos do complexo HLA, em particular, HLA-DR2 e HLA-DR3, ambos da classe HLA II, como importantes fatores de risco genético para susceptibilidade a subtipos da hanseníase. 5,8,10 Esse efeito não é tão evidente para susceptibilidade à hanseníase per se.
B.1.2 MICA e MICB
MICA e MICB são dois genes da classe I do complexo HLA que expressam proteínas na superfície de células quando sujeitas a estresse, fazendo então que sejam reconhecidas por células T CD8+ e natural killer. Estudo recente envolvendo pares de irmãos afetados por hanseníase paucibacilar recrutados no sul da Índia identificou evidência positiva para associação entre a doença e uma variação funcional do gene MICA, bem como com polimorfismo do tipo microssatélite próximo ao gene vizinho MICB. Esta associação parece ser independente daquela observada no mesmo estudo para variantes do gene TNFA, apesar da sua proximidade genômica (Figura 2). O mesmo não pôde ser confirmado para alelos do complexo HLA classe II, especificamente HLA-DRB1, devido à falta de poder de resolução estatístico da amostra.11 Infelizmente, os autores não testaram nenhum marcador para o gene LTA, associado de forma independente com hanseníase em diferentes populações.12 As proteínas codificadas pelos genes MIC parecem ter papel na resposta imune inata à infecção; resta agora esclarecer melhor a relevância dos polimorfismos dos genes MICA e MICB na patogênese da hanseníase.
B.1.3 TNFA
O TNFA é o gene que codifica o fator de necrose tumoral alfa, citocina pró-inflamatória que participa de vários processos biológicos, incluindo a modulação da resposta imune inata e adaptativa. Diversos estudos genéticos têm produzido evidências de impacto de variações genéticas no gene TNFA sobre fenótipos da hanseníase em populações de origem étnica distinta, como a brasileira13 e a indiana.14 Porém, a exata natureza desse efeito ainda é motivo de controvérsia, uma vez que a direção da associação de variantes do gene TNFA tem-se mostrado inconsistente em diferentes estudos, com um mesmo alelo ora associado com fenótipos de resistência, ora com fenótipos de susceptibilidade nas populações brasileira e indiana.
B.1.4 LTA
A linfotoxina alfa, codificada pelo gene LTA, é citocina importante na modulação da resposta inflamatória. Recentemente, estudo em larga escala envolvendo mais de 400 marcadores genéticos da região HLA encontrou forte evidência de associação entre alelos do gene LTA e susceptibilidade à hanseníase per se em duas amostras populacionais independentes do Vietnam do Sul. No mesmo estudo, o achado foi replicado em amostra populacional da Í ndia.12 Interessantemente, evidência para associação, em todas as populações analisadas, mostrou-se mais forte à medida que diminuía a idade de diagnóstico dos casos, sugerindo efeito genético mais pronunciado em pacientes precocemente afetados por hanseníase. Além disso, a proximidade física entre os genes TNFA e LTA (Figura 2) permite especulações quanto a qual dos dois genes senão os dois é o verdadeiro responsável pelos efeitos genéticos observados para esse segmento genômico em diversas doenças complexas.
B.2 Genes não-HLA
Os achados, amplamente replicados, apontando para a região HLA como lócus crucial na definição do risco genético de susceptibilidade a fenótipos da hanseníase têm levado a uma série de perguntas não respondidas. Por exemplo, associação de alelos HLA com hanseníase per se é menos evidente do que com determinadas formas clínicas da doença. É, portanto, razoável pressupor a existência de genes não ligados à região HLA envolvidos na definição da susceptibilidade à hanseníase. Assim, vários genes candidatos não-HLA têm sido testados para ligação e/ou associação com fenótipos da doença. Além disso, até o momento, três scans genômicos completos já foram realizados em coleções de famílias afetadas por hanseníase em diferentes populações. Todos eles apontam para a existência de loci não-HLA envolvidos no controle de fenótipos da doença. O quadro 1 resume alguns dos principais achados envolvendo genes e regiões genômicas não ligadas ao complexo HLA. Mais detalhes desses estudos estão apresentados a seguir.
B.2.1 IL-10
Evidência experimental acumulada sugere que a interleucina-10 é citocina importante na modulação da resposta imune do hospedeiro contra o M. leprae. Estudo de associação baseado em populações realizado por Moraes e colaboradores, do Laboratório de Hanseníase da Fundação Oswaldo Cruz, detectou efeito protetor de haplótipo específico formado por três marcadores intragênicos ao gene IL-10, tanto contra hanseníase per se quanto contra sua forma multibacilar.15,16 No mesmo estudo, haplótipo alternativo composto por alelos dos mesmos três marcadores foi encontrado em associação com susceptibilidade à hanseníase per se, mas não com determinada forma clínica da doença, reforçando a hipótese da participação de variantes do gene IL-10 no controle de fenótipos da hanseníase.
B.2.2 NRAMP1 (ou SCL11A1)
Em importante série de artigos, Gros e colaboradores acumularam sólida evidência de que a resistência natural a parasitas intracelulares no camundongo é controlada pelo gene Natural resistance associated macrophage protein 1 (Nramp1), como foi batizado pelos autores.17 O gene homólogo no ser humano localiza-se na região cromossômica 2q25 e é expresso no baço, fígado, nos pulmões e mais intensamente nos leucócitos do sangue periférico. Hoje, numerosos estudos têm sugerido impacto de variações genéticas do gene NRAMP1 sobre o controle de várias doenças inflamatórias crônicas, auto-imunes e, em particular, infecciosas, tais como Aids, leishmaniose e tuberculose.
As primeiras evidências de que polimorfismos nesse gene seriam fatores de risco para susceptibilidade a doenças em humanos surgiram de estudos com hanseníase. Estudos baseados em famílias encontraram evidência positiva de ligação entre haplótipos do NRAMP1 e susceptibilidade a hanseníase per se18 e à resposta granulomatosa da reação de Mitsuda, analisada como fenótipo tanto quantitativo quanto qualitativo.19 Entretanto, estudos de associação baseados em populações têm encontrado resultados discordantes, deixando ainda incerta a magnitude e a natureza da participação desse gene no controle da manifestação de fenótipos da hanseníase em populações humanas.20-22
B.2.3 Os genes PARK2/PACRG
Um scan genômico para genes de susceptibilidade à hanseníase per se em população vietnamita resultou no primeiro estudo bem-sucedido de clonagem de posição de variantes genéticas exercendo papel no controle da susceptibilidade do ser humano a uma doença infecciosa. Análise de ligação não paramétrica foi aplicada no estudo de 86 famílias multiplex afetadas por hanseníase, recrutadas na cidade de Ho Chi Min, Vietnam do Sul. Foi encontrada forte evidência de ligação entre hanseníase per se e a região cromossômica 6q25-q27, com LOD escore máximo multiponto de 4,31 (P = 2.5x10-14).23 Em estudo complementar, 197 famílias vietnamitas simplex independentes foram utilizadas na construção de um mapa de associação de alta resolução da região candidata 6q25-q27. Esse estudo levou à identificação de 17 SNPs (polimorfismos de nucleotídeo ú nico) associados com hanseníase per se na população vietnamita. Análise de desequilíbrio de ligação da região candidata mostrou que 15 dos 17 SNPs associados estavam localizados em um único bloco com 80 quilobases de extensão, cobrindo região reguladora compartilhada por dois genes: o gene do parkinsonismo juvenil autossômico recessivo (AR-JP), denominado Parquina (PARK2) e o gene PACRG (gene co-regulado com parquina). Análise de regressão logística multivariada detectou dois tag SNPs que capturavam completamente a informação de associação observada na região. Haplótipos definidos por estes dois tag SNPs foram associados com aumento de até 5,28 vezes do risco de contrair hanseníase (P = 0,0005). Ainda no mesmo estudo, esses achados foram integralmente replicados em população caso-controle independente e etnicamente distinta recrutada na cidade do Rio de Janeiro, Brasil.24 Mais recentemente, associação entre um dos tag SNPs associados no Vietnam e Brasil foi detectada em população caso-controle da Índia.25 Embora significância estatística não tenha resistido à correção para testes múltiplos, em um contexto de replicação, esse achado independente reforça a hipótese da participação universal de variantes regulatórias dos genes PARK2 e PACRG no controle da susceptibilidade à hanseníase.
Interessantemente, as mesmas variações do sistema PARK2/PACRG associadas com hanseníase foram relacionadas com susceptibilidade à febre tifóide e paratifóide em população da Indonésia,26 sugerindo papel desses genes candidatos sobre o controle da susceptibilidade do hospedeiro a outros patógenos intracelulares.
B.2.4 VDR
As primeiras evidências de um papel imunológico importante para a vitamina D datam do século XVIII, quanto propriedades terapêuticas do óleo de fígado de peixe eram aplicadas no tratamento da tuberculose. Em meados do século XX, a vitamina D era usada com sucesso no tratamento da tuberculose cutânea. A forma ativa da vitamina D é produzida pelos macrófagos e é capaz de ativar monócitos, estimular a imunidade celular e suprimir a imunidade humoral através da regulação da produção de citocinas. 27 Para exercer seus efeitos, a vitamina D precisa ligar-se a um receptor polipeptídico intracelular específico, codificado pelo gene do receptor da vitamina D (VDR). Essas observações levaram à hipótese de que variantes do gene VDR pudessem estar envolvidas no controle da resposta imune do hospedeiro contra micobactérias. Em hanseníase, Roy e colaboradores 20 conduziram um estudo envolvendo 170 casos de indivíduos com hanseníase tuberculóide, 124 com a forma virchowiana da doença, e 166 doadores de sangue saudáveis. Os autores encontraram associação estatisticamente significativa entre alelos de marcador do gene VDR e susceptibilidade a formas clínicas da hanseníase, com homozigotos "tt" e " TT" enriquecidos nos grupos tuberculóide e virchowiano, respectivamente. Esse achado foi confirmado em estudo mais recente envolvendo população de 270 indivíduos hansenianos e 452 controles saudáveis recrutados em Malawi.28
C Regiões candidatas posicionais
C.1 Regiões cromossômicas 10p13 e 20p12
O primeiro scan genômico para susceptibilidade à hanseníase foi realizado em coleção de famílias indianas das províncias de Tamil Nadu e Andhra Pradesh apresentando predominantemente a forma paucibacilar da doença. Uma análise em dois passos resultou em evidência positiva para ligação entre hanseníase e a região cromossômica 10p13, com LOD escore máximo multiponto de 3,52. A adição de oito marcadores na região candidata resultou em aumento do conteúdo de informação e aumento do LOD escore para 4,09 (P < 2x10-5).29 Esse achado foi confirmado no scan genômico realizado na população vietnamita, quando incluída na análise apenas uma subpopulação de famílias com hanseníase paucibacilar.23 Esse resultado sugere o lócus 10p13 como modificador para a forma clínica paucibacilar da hanseníase.
Em desdobramento do estudo original, análise envolvendo apenas famílias recrutadas na província de Tamil Nadu identificou ligação entre a região cromossômica 20p12 com hanseníase, com LOD escore máximo de dois pontos de 3,48 (P=0,00003).30 Curiosamente, a análise multiponto resultou em LOD escore máximo menor, de 3,16. Quando as famílias de Andhra Pradesh foram incluídas, não foi encontrada ligação estatisticamente significativa na análise multiponto. Embora encorajadores, esses achados devem ser interpretados com cautela, uma vez que a forma paucibacilar da hanseníase não pode ser considerada representativa de todas as formas clínicas da doença. Até o momento, nenhum gene candidato emergiu de estudos envolvendo as regiões 10p13 e 20p12.
C.2 Região cromossômica 6q25-q27
O forte efeito genético de ligação detectado para a região cromossômica 6q25-q27, associado à observação de associação positiva entre hanseníase e alelos de marcadores do tipo microssatélites e SNPs localizados adjacentes à região regulatória dos genes PARK2/PACRG, sugere a presença de genes candidatos adicionais para a susceptibilidade à hanseníase nessa região cromossômica.23,24 Estudos adicionais estão em andamento, com o objetivo de se avançar na dissecção completa do papel deste lócus no controle da susceptibilidade do hospedeiro à doença.
C.3 Região cromossômica 17q11-q21
Mais recentemente, um terceiro scan genômico em hanseníase foi realizado em coleção de 71 famílias brasileiras multiplex multigeracionais. Os autores reportaram evidências sugestivas para ligação entre a região 17q11-q21 e susceptibilidade à hanseníase. O indício de ligação manifesta-se em dois picos com LOD escore máximo de 2,67 (P = 0,004). Infelizmente, o número de trios era insuficiente para análise de associação baseada em famílias. O mesmo estudo reportou fraca evidência para ligação entre a região 20p13 e hanseníase (LOD escore 1,51, P = 0,004), região localizada 3,5 megabases distais do reportado no estudo indiano.31
CONSIDERAÇÕES FINAIS
A participação de fatores genéticos contribuindo para a infecção e fenótipos clínicos da hanseníase, de mera suposição, é hoje uma certeza, graças em grande parte à aplicação das poderosas ferramentas de análise genética modernas atualmente disponíveis. Em conjunto, dados acumulados a partir de estudos observacionais e estudos moleculares de ligação e/ou associação têmnos levado mais próximo da elucidação das bases moleculares que influenciam a susceptibilidade do hospedeiro a essa doença milenar. No entanto, a complexidade da característica, claramente resultado de efeito poligênico associado a ampla gama de variáveis ambientais, ainda faz de sua completa dissecção enorme desafio à ciência. Hoje, uma importante questão diz respeito à viabilidade de aplicação de estudos de associação, mais sensíveis que os de ligação, na investigação de genomas completos, os chamados "estudos pangenômicos de associação". Esses estudos, geradores de hipóteses, embora potencialmente muito poderosos, ainda dependem da aplicação de tecnologia avançada, hoje só disponível em poucos grandes centros de pesquisa, concentrados na América do Norte e Europa. Além disso, a imensa quantidade de dados primários produzida por essas plataformas tecnológicas gera dificuldades consideráveis para sua análise e interpretação, tornando necessário o desenvolvimento de métodos e de estratégias inovadoras de análise estatística. Finalmente, esses estudos pangenômicos ainda são de altíssimo custo. A superação dessas dificuldades é ponto crucial na popularização dessas estratégias.
Na hanseníase, pouco se questiona quando à relevância da participação de genes HLA e não-HLA no controle da manifestação de fenótipos da doença. Alguns dos resultados já obtidos têm levado à implicação de vias metabólicas antes insuspeitas na patogênese da doenca, por exemplo: o gene PARK2 codifica uma E3 proteína-ubiquitina ligase, enzima que participa da via da ubiquitina de degradação de proteínas no proteossomo. Mutações nesse gene têm sido associadas à ocorrência de doença de Parkinson de início precoce. Como a via da ubiquitina participa da patogênese da hanseníase e qual a relação entre hanseníase e doença de Parkinson duas patologias neurodegenerativas crônicas de etiologias distintas são perguntas científicas intrigantes que podem ser exploradas intensamente.
Apesar desses avanços, a exata natureza deste componente genético, em particular o número exato de genes envolvidos, suas funções biológicas e as variações genéticas desses genes responsáveis pelos efeitos observados ainda é amplamente desconhecida. O melhor entendimento do impacto da genética em doenças infecciosas como a hanseníase poderá levar ao desenvolvimento de novas estratégias de diagnóstico, prevenção e terapêutica dessas doenças no futuro.
Aprovado pelo Conselho editorial para publicação em 04.10.2007
Suporte financeiro / Finacial funding: Unicef/UNDP/World Bank/WHO Special Programme for Research and Training in Tropical Diseases (TDR), e Conselho Nacional de Desenvolvimento Científico e Tecnológico (CNPq), Edital MCT/CNPq/MS-SCTIE-DECIT 35/2005 Estudo da Hanseníase
- 1. Beiguelman B. Genética e Hanseníase. Ciência & Saúde. 2002;7:117-28.
- 2. Cole ST, Eiglmeier K, Parkhill J, James KD, Thomson NR, Wheeler PR, et al.Massive gene decay in the leprosy bacillus. Nature. 2001;409:1007-11.
- 3. Monot M, Honore N, Garnier T, Araoz R, Coppee JY, Lacroix C, et al.On the origin of leprosy. Science. 2005;308:1040-2.
- 4. Ridley DS, Jopling WH. Classification of leprosy according to immunity. A five-group system. Int J Lepr Other Mycobact Dis. 1966;34:255-73.
- 5. Mira MT. Genetic host resistance and susceptibility to leprosy. Microbes Infect. 2006;8:1124-31.
- 6. Chakravartii MR. A twin study on leprosy. Stuttgart: Georg Thieme; 1973.
- 7. Abel L, Demenais F. Detection of major genes for susceptibility to leprosy and its subtypes in a Caribbean island: Desirade island. Am J Hum Genet. 1988;42:256-66.
- 8. Shaw MA, Donaldson IJ, Collins A, Peacock CS, Lins Lainson Z, Shaw JJ, et al.Association and linkage of leprosy phenotypes with HLA class II and tumour necrosis factor genes. Genes Immun. 2001;2:196-204.
-
9Wellcome Trust Sanger Institute [homepage on the internet]. Cambridge: Human Chromosome 6 Project Overview; [updated 2007 August 3; cited 2007 October 8]. Available from: http://www.sanger.ac.uk/HGP/Chr6/
- 10. Mira MT, Alcais A, di Pietrantonio T, Thuc NV, Phuong MC, Abel L, et al.Segregation of HLA/TNF region is linked to leprosy clinical spectrum in families displaying mixed leprosy subtypes. Genes Immun. 2003;4:67-73.
- 11. Tosh K, Ravikumar M, Bell JT, Meisner S, Hill AV, Pitchappan R. Variation in MICA and MICB genes and enhanced susceptibility to paucibacillary leprosy in South India. Hum Mol Genet. 2006;15:2880-7.
- 12. Alcais A, Alter A, Antoni G, Orlova M, Nguyen VT, Singh M, et al.Stepwise replication identifies a lowproducing lymphotoxin-alpha allele as a major risk factor for early-onset leprosy. Nat Genet. 2007;39:517-22.
- 13. Santos AR, Suffys PN, Vanderborght PR, Moraes MO, Vieira LM, Cabello PH, et al.Role of tumor necrosis factor-alpha and interleukin-10 promoter gene polymorphisms in leprosy. J Infect Dis. 2002;186:1687-91.
- 14. Roy S, McGuire W, Mascie-Taylor CG, Saha B, Hazra SK, Hill AV, et al.Tumor necrosis factor promoter polymorphism and susceptibility to lepromatous leprosy. J Infect Dis. 1997;176:530-2.
- 15. Moraes MO, Cardoso CC, Vanderborght PR, Pacheco AG. Genetics of host response in leprosy. Lepr Rev. 2006;77:189-202.
- 16. Moraes MO, Pacheco AG, Schonkeren JJ, Vanderborght PR, Nery JA, Santos AR, et al.Interleukin-10 promoter single-nucleotide polymorphisms as markers for disease susceptibility and disease severity in leprosy. Genes Immun. 2004;5:592-5.
- 17. Skamene E, Schurr E, Gros P. Infection genomics: Nramp1 as a major determinant of natural resistance to intracellular infections. Annu Rev Med. 1998;49:275-87.
- 18. Abel L, Sanchez FO, Oberti J, Thuc NV, Hoa LV, Lap VD, et al.Susceptibility to leprosy is linked to the human NRAMP1 gene. J Infect Dis. 1998;177:133-45.
- 19. Alcais A, Sanchez FO, Thuc NV, Lap VD, Oberti J, Lagrange PH, et al.Granulomatous reaction to intradermal injection of lepromin (Mitsuda reaction) is linked to the human NRAMP1 gene in Vietnamese leprosy sibships. J Infect Dis. 2000;181:302-8.
- 20. Roy S, Frodsham A, Saha B, Hazra SK, Mascie-Taylor CG, Hill AV. Association of vitamin D receptor geno type with leprosy type. J Infect Dis. 1999;179:187-91.
- 21. Meisner SJ, Mucklow S, Warner G, Sow SO, Lienhardt C, Hill AV. Association of NRAMP1 polymorphism with leprosy type but not susceptibility to leprosy per se in west Africans. Am J Trop Med Hyg. 2001;65:733-5.
- 22. Ferreira FR, Goulart LR, Silva HD, Goulart IM. Susceptibility to leprosy may be conditioned by an interaction between the NRAMP1 promoter polymorphisms and the lepromin response. Int J Lepr Other Mycobact Dis. 2004;72:457-67.
- 23. Mira MT, Alcais A, Van Thuc N, Thai VH, Huong NT, Ba NN, et al.Chromosome 6q25 is linked to susceptibility to leprosy in a Vietnamese population. Nat Genet. 2003;33:412-5.
- 24. Mira MT, Alcais A, Nguyen VT, Moraes MO, Di Flumeri C, Vu HT, et al.Susceptibility to leprosy is associated with PARK2 and PACRG. Nature. 2004;427:636-40.
- 25. Malhotra D, Darvishi K, Lohra M, Kumar H, Grover C, Sood S, et al.Association study of major risk single nucleotide polymorphisms in the common regulatory region of PARK2 and PACRG genes with leprosy in an Indian population. Eur J Hum Genet. 2006;14:438-42.
- 26. Ali S, Vollaard AM, Widjaja S, Surjadi C, van de Vosse E, van Dissel JT. PARK2/PACRG polymorphisms and susceptibility to typhoid and paratyphoid fever. Clin Exp Immunol. 2006;144:425-31.
- 27. Hayes CE, Nashold FE, Spach KM, Pedersen LB. The immunological functions of the vitamin D endocrine system. Cell Mol Biol (Noisy-le-grand). 2003;49:277-300.
- 28. Fitness J, Floyd S, Warndorff DK, Sichali L, Mwaungulu L, Crampin AC, et al.Large-scale candi date gene study of leprosy susceptibility in the Karonga district of northern Malawi. Am J Trop Med Hyg. 2004;71:330-40.
- 29. Siddiqui MR, Meisner S, Tosh K, Balakrishnan K, Ghei S, Fisher SE, et al.A major susceptibility locus for leprosy in India maps to chromosome 10p13. Nat Genet. 2001;27:439-41.
- 30. Tosh K, Meisner S, Siddiqui MR, Balakrishnan K, Ghei S, Golding M, et al.A region of chromosome 20 is linked to leprosy susceptibility in a South Indian population. J Infect Dis. 2002;186:1190-3.
- 31. Miller EN, Jamieson SE, Joberty C, Fakiola M, Hudson D, Peacock CS, et al.Genome-wide scans for leprosy and tuberculosis susceptibility genes in Brazilians. Genes Immun. 2004;5:63-7.
Datas de Publicação
-
Publicação nesta coleção
01 Abr 2008 -
Data do Fascículo
Out 2007