Resumos
Este estudo foi realizado com o objetivo de aplicar a técnica de REP-PCR no monitoramento da qualidade do leite de cabra por meio da detecção de Staphylococcus aureus em amostras de mãos de ordenhador, tetos das cabras, leite, ordenhadeira e água para estabelecimento e implantação do sistema de Análise de Perigos e Pontos Críticos de Controle (APPCC). Verificaram-se vários fingerprints de todos os isolados coletados das diferentes fontes estudadas (mãos de ordenhador, tetos das cabras, leite, ordenhadeira e água). Observaram-se comportamentos muito similares das bandas, o que indica que os isolados podem ser relatados como clones epidemiológicos. As mãos do ordenhador caracterizaram-se como ponto crítico de controle, pois se destacaram como iniciador de contaminação nas amostras Staphylococcus aureus. A técnica demonstrou ser eficiente para a análise da similaridade entre indivíduos da espécie Staphylococcus aureus e, portanto, constitui ferramenta útil para investigação de falhas no manejo e na busca de controle mais eficiente para evitar ou reduzir a disseminação de microrganismos patogênicos causadores de sérias enfermidades em humanos e animais, que muitas vezes podem ser transmitidas por produtos como o leite e seus derivados.
epidemiologia molecular; identidades genômicas; manejo
The objective of the present study was to apply REP-PCR sequences in the monitoring goat milk quality, by detecting Staphylococcus aureus, in samples of from milker hands, goat teats, milk, milking machine and water, for the future establishment and implantation of the system of Hazard Analysis Critical Control Points (HACCP). Several fingerprints were verified of all the isolates collected from the different sources studied (milker hands, goat teats, milk, milking machine and water). Very similar band behaviors were observed that indicated that the isolates can be reported as epidemic clones. Milker´s hands were was characterized as a critical control point (CCP), because it stands out as an initial point of contaminationin the Staphylococcus aureus samples. The technique was shown to be efficient for the similarity analysis among individuals of the Staphylococcus aureus species, and is therefore a useful tool for investigation of management faults and consequently, in the search for more efficient control to prevent or minimize the spread of pathogenic microorganisms that cause serious illnesses in humans and animals that can be transmitted through products such as milk and its products.
fingerprints; management; molecular epidemiology
PRODUÇÃO ANIMAL
Detecção de Staphylococcus aureus utilizando a técnica de REP-PCR no monitoramento da qualidade do leite de cabra1 1 Pesquisa financiada pela EMBRAPA.
Detection of Staphylococcus aureus using the REP-PCR technique to monitor goat milk quality
Cellyneude de Souza OlivindoI; Lea ChapavalII; Arturo Bernardo Selaive VillarroelIII; Francisco Selmo Fernandes AlvesII; Francisca Geovania Canafístula de SousaIV; Francisco Eden Paiva FernandesI
IDoutorando em Zootecnia - Universidade Federal de Viçosa, Viçosa, MG
IIEmbrapa Caprinos, Sobral, CE
IIIDepartamento de Zootecnia - Universidade Federal do Ceará, Fortaleza, CE
IVGraduanda em Biologia - Universidade Estadual Vale do Acaraú, Sobral, CE
Resumo
Este estudo foi realizado com o objetivo de aplicar a técnica de REP-PCR no monitoramento da qualidade do leite de cabra por meio da detecção de Staphylococcus aureus em amostras de mãos de ordenhador, tetos das cabras, leite, ordenhadeira e água para estabelecimento e implantação do sistema de Análise de Perigos e Pontos Críticos de Controle (APPCC). Verificaram-se vários fingerprints de todos os isolados coletados das diferentes fontes estudadas (mãos de ordenhador, tetos das cabras, leite, ordenhadeira e água). Observaram-se comportamentos muito similares das bandas, o que indica que os isolados podem ser relatados como clones epidemiológicos. As mãos do ordenhador caracterizaram-se como ponto crítico de controle, pois se destacaram como iniciador de contaminação nas amostras Staphylococcus aureus. A técnica demonstrou ser eficiente para a análise da similaridade entre indivíduos da espécie Staphylococcus aureus e, portanto, constitui ferramenta útil para investigação de falhas no manejo e na busca de controle mais eficiente para evitar ou reduzir a disseminação de microrganismos patogênicos causadores de sérias enfermidades em humanos e animais, que muitas vezes podem ser transmitidas por produtos como o leite e seus derivados.
Palavras-chave: epidemiologia molecular, identidades genômicas, manejo
ABSTRACT
The objective of the present study was to apply REP-PCR sequences in the monitoring goat milk quality, by detecting Staphylococcus aureus, in samples of from milker hands, goat teats, milk, milking machine and water, for the future establishment and implantation of the system of Hazard Analysis Critical Control Points (HACCP). Several fingerprints were verified of all the isolates collected from the different sources studied (milker hands, goat teats, milk, milking machine and water). Very similar band behaviors were observed that indicated that the isolates can be reported as epidemic clones. Milker´s hands were was characterized as a critical control point (CCP), because it stands out as an initial point of contaminationin the Staphylococcus aureus samples. The technique was shown to be efficient for the similarity analysis among individuals of the Staphylococcus aureus species, and is therefore a useful tool for investigation of management faults and consequently, in the search for more efficient control to prevent or minimize the spread of pathogenic microorganisms that cause serious illnesses in humans and animals that can be transmitted through products such as milk and its products.
Key Words: fingerprints, management, molecular epidemiology
Introdução
O sistema APPCC (análise de perigos e pontos críticos de controle) tem sido útil para avaliar os riscos de perigos e estabelecer mecanismos ou medidas de controle visando reduzir os riscos de contaminação física, química ou microbiológica (Heeschen et al., 1997). A implementação do processo APPCC é uma alternativa no ciclo de produção do leite de cabra, no entanto, faltam informações e dados e existem dificuldades de seleção dos Pontos Críticos de Controle (PCC) para um número de perigos biológicos encontrados nas fazendas, onde há a impossibilidade de erradicar ou controlar a maior parte dos patógenos, especialmente aqueles que causam problemas clínicos em animais e que também podem causar danos à saúde humana, como o Staphylococcus aureus. Assim, métodos baseados em DNA estão surgindo como vias confiáveis, simples e acessíveis, para identificar e classificar microrganismos (Rademarker & De Bruijn, 2003). O método referido como REP-PCR, "impressão digital" do genoma, uma técnica baseada na amplificação do DNA, é considerada extremamente confiável, reprodutível, rápida e altamente discriminatória (Versalovic et al., 1994; Louws et al., 1996).
Este estudo foi realizado com o objetivo de aplicar a técnica de PCR em sequências palindrômicas extragênicas repetidas (REP-PCR) no monitoramento da qualidade do leite de cabra, por meio da detecção de Staphylococcus aureus em amostras de mãos de ordenhador, tetos das cabras, leite, ordenhadeira e água, para futuro estabelecimento e implantação do sistema APPCC.
Material e Métodos
O estudo foi conduzido na Embrapa Caprinos, em Sobral, região norte do estado do Ceará, no período de agosto de 2005 a janeiro de 2006. Para obtenção das cepas bacterianas de Staphylococcus aureus utilizadas neste estudo, utilizaram-se 15 fêmeas caprinas da raça Saanen, em início de lactação, as quais foram ordenhadas duas vezes ao dia, utilizando-se ordenha mecânica, e submetidas à higienização do úbere, pré e pós-ordenha, com solução iodada. Foram coletadas 40 amostras na primeira ordenha às 7h30, divididas da seguinte forma: amostras de leite (n = 15), swab das teteiras (n = 3), swabs das mãos do ordenhador (n = 3) e swabs dos tetos dos animais (n = 15) coletadas no início, no meio e no final da ordenha. As amostras da água utilizada para a lavagem das mãos dos ordenhadores (n = 4) foram coletadas antes e após o tratamento da água com água sanitária, no início, meio e final da ordenha. A coleta foi realizada utilizando-se técnicas assépticas e conforme protocolos padrões (NMC, 1990). As amostras foram encaminhadas em caixa térmicas para o Laboratório de Bacteriologia da Embrapa Caprinos.
O material coletado foi semeado e as culturas que se mostraram positivas (presença de colônias características) em meios de cultura seletivos (ágar Baird Paker OXOID®, England) foram inoculadas em tubos tipo Falcon de 15 mL, em 5 mL de caldo cérebro coração (BHI) durante 18 horas a 37°C (dez identificadas como características para cada amostra semeada), priorizando a extração do DNA da população a ser estudada.
Para extração de DNA das cepas bacterianas, utilizou-se o protocolo proposto por Chapaval et al. (2006), no qual uma quantidade de 2,5 mL de cultura da população bacteriana, para cada amostra semeada, crescida em meio BHI por 18 horas a 37°C foram precipitados em tubos de microcentrífuga com 1,7 mL por meio de um pulso de centrifugação a 14.000 rpm. O sobrenadante foi eliminado e este processo foi repetido até que todo o volume (2,5 mL) fosse precipitado. Ao pélete obtido foram adicionados 700 mL de tampão de extração (1,4 M NaCl; 100 mM Tris-HCl, pH 8,0; 20mM EDTA, pH8,0; PVP-40 1%; CTAB 2%; Proteinase K, 20mg/mL; δ-Mercaptoetanol 0,2%). A solução foi misturada em vórtex e incubada por 30 minutos a 65ºC em banho-maria, sendo misturada com o cuidado de não se fazer movimentos bruscos a cada 10 minutos. Foram acrescentados 650 μL de clorofórmio:álcool-isoamílico (24:1), a solução foi homogeneizada até formar uma emulsão e centrifugada a 14.000 rpm durante 7 minutos. A fase aquosa foi transferida para um novo tubo e adicionada de 200 mL de tampão de extração sem Proteinase K, homogeneizado, e adicionados então 650 μL de clorofórmio:álcool-isoamílico (24:1), homogeneizada novamente e centrifugada a 14.000 rpm por 7 minutos. O processo com 650 μL de clorofórmio:álcool-isoamílico (24:1) foi repetido por mais duas vezes até que a fase aquosa adquirisse aparência translúcida. O DNA foi precipitado com 1 volume de Isopropanol conservado em temperatura ambiente, homogeneizado e centrifugado a 14.000 rpm durante 7 minutos. O sobrenadante foi então removido e a superfície do precipitado foi lavada por duas vezes com 70 mL de etanol 70%, preparado um pouco antes do uso. A cada lavagem o precipitado foi centrifugado por 2 minutos a 14.000 rpm. Em seguida o pélete foi seco em temperatura ambiente por 30 minutos e ressuspendido em 40 μL de TE (10 mM Tris-HCl, pH 8,0; 1 mM EDTA, pH 8,0 + 10 mg/μL de RNAse) e deixado em banho-maria a 37°C por 30 minutos.
A síntese de oligonucleotídeos foi elaborada pela Bio Global Com. Ltda., Curitiba, Paraná, Brasil. As sequências utilizadas foram as seguintes: REP-PCR (REP 1: 5' NNN NCG NCG NAC TCC NGG C 3' ; REP 2: 5' NCG NCT TAT CNG GCC TAC TAC 3'descritas porDe Bruijn(1992).
Sequencialmente, o PCR foi realizado com 25 μL do volume total, incluindo 5 μL do DNA alvo (20 a 90 ng/μL). Os componentes do master mix (GIBCO BRL - Life Technologies, Inc., MD, U.S.A.) foram utilizados conforme as instruções do fabricante. As amostras de DNA alvo (máximo de 5 μL) foram amplificadas em termociclador Gene Amp® PCR System 9700 (Perkim Elmer) com os seguintes ciclos: para REP, desnaturação inicial 95°C por 6 minutos seguida por 30 ciclos de amplificação (desnaturação a 94°C por 1 minuto, anelamento a 40°C por 1 minuto, polimerização a 65°C por 8 minutos), terminando com a polimerização final a 65°C por 7 minutos. Com o intuito de realizar detecção dos produtos de PCR, 8 μL do produto amplificado pela reação de PCR e observado por eletroforese em gel de agarose 2% (GIBCO BRL - Life Technologies, Inc., MD, U.S.A.) em TAE 1x (para 50 X, 242 g Tris; 37,2 g EDTA [Na2], 800 mL de água MilliQ autoclavada, 57% de ácido acético, pH 8,1) e um padrão molecular 1kb DNA (GIBCO BRL - Life Technologies, Inc., MD, U.S.A.) foi usado como marcador molecular. A eletroforese foi realizada em cuba eletroforética Pharmacia Biotech (max submarine unit HE 99 X), com fonte Pharmacia Biotech (EPS 300) nas condições de 70 V por 2 horas.
A análise das similaridades entre as cepas foi baseada na presença ou ausência de bandas específicas na análise da reação de PCR, de modo quediferenças e similaridades entre as cepas foram analisadas visualmente de acordo com o comportamento de migração das bandas dos produtos da reação de PCR. Os perfis das 11 primeiras bandas foram considerados altamente similares quando todas as bandas visíveis dos isolados possuíam a mesma distância aparente de migração. Quando não houve possibilidade de comparação entre migração de bandas semelhantes, os isolados foram considerados diferentes. Uma matriz de similaridade foi obtida por meio de comparações usando um coeficiente simples de similaridade (coeficiente de Jaccard). Para esta análise, o programa utilizado foi o NTSYS Numerical Taxonomy and Multivariate Analysis System, versão NTSYSpc 2.0 (Rohlf, 1997). Um dendograma foi construído pela obtenção das médias aritméticas de grupos em pares de dados combinados (UP-GMA).
Após a análise computacional dos dados de similaridade genética, o estudo da filogenia foi realizado. Organismos considerados geneticamente iguais, ou semelhantes, foram considerados clones epidemiológicos e, portanto, de mesma ancestralidade e origem.
Resultados e Discussão
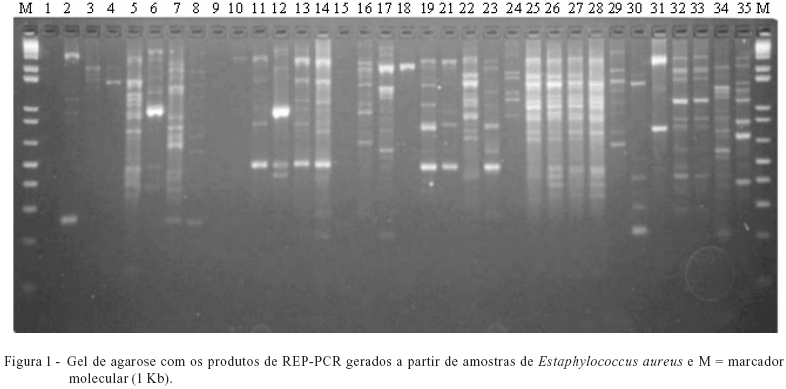
Em geral, os modelos das bandas dos isolados das fontes (leite, teto, teteira, mão do ordenhador e água) foram similares (Figura 1) e indicam que os isolados são estreitamente relacionados. Foram similares, porém nem sempre idênticos.
O comportamento das bandas maiores que 300pb até 10.000 pb obtidas pelo uso de primer REP (Figura 1) para Staphylococcus aureus comprovou similaridade em aproximadamente um quarto das bandas que foram comuns a 80% dos isolados, podendo diferenciar as cepas dentro de cinco agrupamentos (clusters) distintos de acordo com o dendograma gerado (Figura 2).
Verificaram-se no grupo 1 similaridades que variavam entre 75 e 100%. A amostra R1 que corresponde mão ordenhador no início do procedimento de ordenha mostrou-se, em aproximadamente 80%, similar às amostras R21 (teto final), R24 (leite início) e R27 (leite meio), que, por sua vez, são idênticas entre si (100% de similaridade) e 90% similares às amostras R25 (leite início), R26 (leite início) e R29 (leite meio), também 100% similares entre si. Nesta situação, observou-se a presença de vários clones (100% de similaridade), o que permite inferir, considerando a similaridade genética, que provavelmente o leite inicialmente contaminado passa para mão do ordenhador e torna-se fonte de contaminação para as amostras presentes no mesmo grupo.
Verificou-se no grupo 2 similaridade de aproximada-mente 85% entre as amostras de swab de teto coletada no meio da ordenha (R12) e amostra de leite coletada no final da ordenha (R30).
No grupo 3, observaram-se similaridades entre as amostras de 75 a 100%, a amostra R2 (swab da mão do ordenhador coletado no meio da ordenha) possui similaridade de aproximadamente 95% para com as amostras R15 (teto do meio) e R23 (leite do início da ordenha), que, por sua vez, são 100% similares entre si. Ainda no grupo 3, as amostras R9 (swab do teto do início), R31 e R32 (amostras de leite do final da ordenha, respectivamente) se mostraram 100% similares entre si, o que indica contaminação no momento inicial da ordenha, transmitida até o final da mesma, conforme situação relatada anteriormente no grupo 1. Entre as amostras R8 (swab de teto coletadas no início da ordenha e R33 (de leite coletada no final da ordenha), o índice de similaridade foi de aproximadamente 90%.
Observou-se no grupo 4 que as amostras R10 (swab de tetos do início da ordenha) e R18 (swab de tetos final da ordenha) possuem aproximadamente 85% de similaridade entre si. No entanto, o grupo 5 apresentou índice de similaridade entre 60 e 75% e reúne amostras de tetos do início, meio e final da ordenha e uma amostra de swab da teteira. Com exceção do grupo 5, os demais grupos não demonstraram grandes variações nos índices de similaridade.
Os índices de similaridade observados neste estudo podem ser justificados pelo fato de que o Staphylococcus aureus têm sido apontada como bactéria contagiosa, ou seja, que pode ser transmitida durante as ordenhas e que fossas nasais, garganta, equipamento de ordenha (principalmente teteiras), pele dos tetos e humana são considerados importantes reservatórios destes organismos.
A REP-PCR foi uma ferramenta importante para a rápida identificação de cepas de Staphylococcusaureus e pode ser recomendada para detectar esta espécie em outros estudos. Esta técnica foi também utilizada por Wieser & Busse (2000) para rápida identificação de cepas de Staphylococcus epidermidis em mesma espécie. Além disso, Nascimento et al. (2005) afirmaram que esta técnica gera perfis moleculares com alto grau de similaridade e alta resolução e que podem ser obtidos em torno de 48 horas a partir de isolados de cultura microbiológica.
Segundo Struelens (1996) e Maslow & Mulligan (1996) a análise de similaridade molecular contribui para vigilância epidemiológica, pois possibilita a documentação dos clones epidêmicos pelo tempo e sua circulação em populações infectadas. Del Vecchio et al. (1995) utilizaram a técnica de REP-PCR para identificar cepas epidêmicas entre isolados de Staphylococcus aureus resistentes a meticilina (MRSA) e afirmaram que é importante o desenvolvimento de metodologias de tipagem moleculares rápidas e precisas na identificação da fonte de contaminação e disseminação de doenças infecciosas.
Conclusões
As mãos do ordenhador caracterizam-se como ponto crítico de controle, pois se destacam como iniciador de contaminação nas amostras Staphylococcus aureus. A técnica de REP-PCR é eficiente para a análise da similaridade entre indivíduos da espécie Staphylococcus aureus e também apresenta-se como ferramenta útil na produção animal para a rápida identificação de pontos de contaminação bacteriana horizontal durante o manejo de ordenha, o que permite a reavaliação de procedimentos de higienização de equipamentos, higiene pessoal do trabalhador da sala de ordenha, bem como da rotina de limpeza de tetos e eficácia dos desinfetantes utilizados para que, em tempo hábil, falhas no manejo sejam detectadas e corrigidas.
Literatura Citada
Este artigo foi recebido em 3/8/2007 e aprovado em 14/10/2008.
Correspondências devem ser enviadas para: cellyneudeolivindo@yahoo.com.br
- CHAPAVAL, L.; MOON, D. H.; GOMES, J.E. et al. Aplicação da técnica de Rep-PCR no rastreamento de Staphylococcus aureus em sala de ordenha para o monitoramento da qualidade do leite. Brazilian Journal of Veterinary Research and Animal Science, v.43, n.3, p.309-320, 2006.
- De BRUIJN, F.J. Use of repetitive (repetitive extragenic palindromic and enterocaterial repetitive intergenic consensus) sequences and the polymerase chain reaction to fingerprint the genomos of Rizobium meliloti isolates and other soil bacteria. AppliedEnvironmental Microbiology, v.58, p.2180-2187, 1992.
- DELVECCHIO, V.G.J.M.; PETROZIELLO, M.J.; GRESS, F.K. et al. Molecular genotyping of methicillin-resistant Staphylococcus aureus via fluorophore-enhanced repetitive- sequence PCR. Journal Clinical Microbiology, v.33, n.8, p.21412144. 1995.
- HEESCHEN, W.; REICHMUTH, J.; SUHREN, G. Quality milk production - potential hazards, critical control points and the application of risk analysis. In: NATIONAL MASTITIS COUNCIL ANNUAL MEETING, 36., 1997, s.l.. Proceedings... s.l., 1997. v.36, p.4-18.
- LOUWS, F.J.; SCHNEIDER, M.; De BRUJIN, F.J. In: TORANZOS, G. (Ed.) Nucleic acid amplification methods for the analisys of environmental samples Lancaste: Technomic Publication, p.63-94, 1996.
- MASLOW, J.; MULLIGAN, M.E. Epidemiologic typing systems. infection control and hospital epidemiology. The official Journal of the Society of Hospital Epidemiologistsof America, v.17, p.595-604,1996.
- National Mastitis Council - NMC. Current concepts of bovine mastitis 4.ed. Madison: National Mastitis Coucil, 1996. 64p.
- NASCIMENTO, S.B.; SOUSA, R.B.; MARTINS, M.J. et al. Glutamine depletion potentiates leucocyte-dependent inflammatory events induced by carrageenan or Clostridium difficile toxin A in rats. Journal of Immunology, v.116, n.3, p.328-36, 2005.
- RADEMAKER, J.L.W.; DE BRUIJN, F.J. Characterizarion and classification of microbes by Rep-PCR genomic fingerprint and computer-assisted pattern analysis Disponível em: < http://www.msu.edu/asci/debrujin/dna.htm>. Acesso em: 28/2/2003.
- ROHLF, F.J. NTSYSpc: numerical taxonomy and multivariate analyses system. Version 2.0. New York: Exeter Publications, 1997. (CD-ROM).
- STRUELENS, M.J. Consensus guidelines for appropriate use and evaluation of microbial epidemiologic typing systems. Clinical Microbiology and Infection, v.2, p.2-11, 1996.
- VERSALOVICK, J.; SCHNEIDER, M.; De BRUIJN, F.J. et al. Genomic fingerprinting of bacteria using repetitive sequence based PCR (rep-PCR). Methods of Cellular and Molecular Biology, v.5, p.25-40, 1994.
- WIESER, M.; BUSSE, H.J. Rapid identification of Staphylococcus epidermidis International Journal of Systematic and Evolutionary Microbiology,v.50, p.1087-1093, 2000.
Datas de Publicação
-
Publicação nesta coleção
15 Set 2009 -
Data do Fascículo
Jul 2009
Histórico
-
Recebido
03 Ago 2007 -
Aceito
14 Out 2008