Resumos
A preservação adequada do DNA de um determinado organismo é fundamental para o sucesso no uso de técnicas moleculares como RAPD-PCR. O objetivo deste trabalho foi comparar métodos simples de preservação de espécimes de Dalbulus maidis (DeLong & Wolcott) (Hemiptera: Cicadellidae) quanto à quantidade e qualidade do DNA para uso em RAPD-PCR, após períodos sucessivos de armazenamento. Avaliaram-se oito métodos: congelamento (-20ºC); álcool etílico 70% (-20ºC e temperatura ambiente); álcool etílico absoluto (-20ºC e temperatura ambiente); secagem ao ar; e preservação em tampão de extração (inseto inteiro e macerado). A intervalos de 10-30 dias, o DNA foi extraído, quantificado e amplificado via RAPD-PCR pelo oligonucleotídeo OPA-04. A qualidade do DNA extraído foi observada em gel de agarose 0,8%. Após 210 dias de armazenamento, o congelamento (-20ºC) mostrou ser a melhor técnica. Quantidades satisfatórias de DNA também foram obtidas dos insetos conservados em álcool etílico absoluto (-20ºC), álcool etílico 70% (-20ºC) e tampão de extração (inseto inteiro e macerado). Insetos conservados em álcool etílico absoluto (temperatura ambiente), em álcool etílico 70% (temperatura ambiente) e secos ao ar mostraram-se impróprios para estudos com RAPD-PCR após 120, 60 e 10 dias de armazenamento, respectivamente.
Insecta; cigarrinha-do-milho; marcador molecular; conservação de DNA; técnica de armazenamento
The appropriate preservation of the DNA of a certain organism is important for successful application of molecular techniques like RAPD-PCR. This study was designed to compare simple methods of preservation of specimens of Dalbulus maidis (DeLong & Wolcott) (Hemiptera: Cicadellidae) with respect to quantity and quality of DNA for use in RAPD-PCR, after different storage periods. Eight methods were tested: freezing (-20ºC); ethyl alcohol 70% (-20ºC and room temperature); absolute ethyl alcohol (-20ºC and room temperature); air-dry; and preservation in extraction buffer (whole and homogenized insect). At intervals of 10 to 30 days, insect DNA was extracted, quantified and amplified through RAPD-PCR using primer OPA-04. The quality of extracted DNA was observed on 0.8% agarose gel. After 210 days of preservation, freezing (-20ºC) showed to be the best method. Satisfactory quantities of DNA were also obtained from insects conserved in absolute ethyl alcohol (-20ºC), ethyl alcohol 70% (-20ºC) and extraction buffer (whole and homogenized insect). Insects conserved in absolute ethyl alcohol (room temperature), ethyl alcohol 70% (room temperature) and air-dry conditions were inappropriate for RAPD-PCR studies after 120, 60 and 10 days of storage, respectively.
Insecta; corn leafhopper; molecular marker; DNA conservation; storage technique
SYSTEMATICS, MORPHOLOGY AND PHYSIOLOGY
Análise Comparativa da Estabilidade do DNA de Dalbulus maidis (DeLong & Wolcott) (Hemiptera: Cicadellidae) sob Diferentes Métodos de Preservação Para Uso em RAPD-PCR
CHARLES M. OLIVEIRA1, MARIA H.P. FUNGARO2, LUÍS E.A. CAMARGO1 E JOÃO R.S. LOPES1
1Depto. Entomologia, Fitopatologia e Zoologia Agrícola, ESALQ/USP, C. postal 9, 13418-900, Piracicaba, SP
2Depto. Biologia Geral, Universidade Estadual de Londrina, C. postal 6001, 86051-970, Londrina, PR
Comparative Analysis of the Stability of DNA of Dalbulus maidis (DeLong & Wolcott) (Hemiptera: Cicadellidae) Under Different Methods of Preservation for Use in RAPD-PCR
ABSTRACT- The appropriate preservation of the DNA of a certain organism is important for successful application of molecular techniques like RAPD-PCR. This study was designed to compare simple methods of preservation of specimens of Dalbulus maidis (DeLong & Wolcott) (Hemiptera: Cicadellidae) with respect to quantity and quality of DNA for use in RAPD-PCR, after different storage periods. Eight methods were tested: freezing (-20oC); ethyl alcohol 70% (-20oC and room temperature); absolute ethyl alcohol (-20oC and room temperature); air-dry; and preservation in extraction buffer (whole and homogenized insect). At intervals of 10 to 30 days, insect DNA was extracted, quantified and amplified through RAPD-PCR using primer OPA-04. The quality of extracted DNA was observed on 0.8% agarose gel. After 210 days of preservation, freezing (-20oC) showed to be the best method. Satisfactory quantities of DNA were also obtained from insects conserved in absolute ethyl alcohol (-20oC), ethyl alcohol 70% (-20oC) and extraction buffer (whole and homogenized insect). Insects conserved in absolute ethyl alcohol (room temperature), ethyl alcohol 70% (room temperature) and air-dry conditions were inappropriate for RAPD-PCR studies after 120, 60 and 10 days of storage, respectively.
KEY WORDS: Insecta, corn leafhopper, molecular marker, DNA conservation, storage technique.
RESUMO - A preservação adequada do DNA de um determinado organismo é fundamental para o sucesso no uso de técnicas moleculares como RAPD-PCR. O objetivo deste trabalho foi comparar métodos simples de preservação de espécimes de Dalbulus maidis (DeLong & Wolcott) (Hemiptera: Cicadellidae) quanto à quantidade e qualidade do DNA para uso em RAPD-PCR, após períodos sucessivos de armazenamento. Avaliaram-se oito métodos: congelamento (-20oC); álcool etílico 70% (-20oC e temperatura ambiente); álcool etílico absoluto (-20oC e temperatura ambiente); secagem ao ar; e preservação em tampão de extração (inseto inteiro e macerado). A intervalos de 10-30 dias, o DNA foi extraído, quantificado e amplificado via RAPD-PCR pelo oligonucleotídeo OPA-04. A qualidade do DNA extraído foi observada em gel de agarose 0,8%. Após 210 dias de armazenamento, o congelamento (-20oC) mostrou ser a melhor técnica. Quantidades satisfatórias de DNA também foram obtidas dos insetos conservados em álcool etílico absoluto (-20oC), álcool etílico 70% (-20oC) e tampão de extração (inseto inteiro e macerado). Insetos conservados em álcool etílico absoluto (temperatura ambiente), em álcool etílico 70% (temperatura ambiente) e secos ao ar mostraram-se impróprios para estudos com RAPD-PCR após 120, 60 e 10 dias de armazenamento, respectivamente.
PALAVRAS-CHAVE: Insecta, cigarrinha-do-milho, marcador molecular, conservação de DNA, técnica de armazenamento.
Técnicas moleculares tornaram-se ferramentas importantes no estudo genético de populações naturais de vários organismos. Entre elas está a técnica de DNA polimórfico amplificado ao acaso ("Random Amplified Polymorphic DNA" - RAPD), desenvolvida de forma independente por dois grupos de pesquisadores nos EUA (Welsh & McClelland 1990, Willians et al. 1990). Embora algumas técnicas baseadas na reação da polimerase em cadeia ("Polymerase Chain Reaction" - PCR) (Saiki et al. 1988) produzam resultados satisfatórios utilizando-se DNA em pequenas quantidades e com algum grau de degradação (Reiss et al. 1995), outras, como RAPD, exigem um DNA íntegro, sendo que espécimes frescos constituem a melhor fonte de material genético para esse tipo de estudo. É comum em entomologia o intercâmbio de materiais entre pesquisadores e também a coleta de espécimes em regiões distantes. No entanto, a manutenção de espécimes vivos até o momento do processamento visando análise genética é inviável na maioria dos casos. Assim, torna-se necessário que estes insetos sejam mortos e convenientemente conservados até o momento de sua manipulação em laboratório.
Alguns estudos têm sido realizados para determinar o efeito de diferentes métodos de preservação na quantidade e qualidade do DNA (Goelz et al. 1985, Smith et al. 1987, Bramwell & Burns 1988); entretanto, a maioria deles foi realizada com tecidos de vertebrados, sendo que os resultados não são necessariamente aplicáveis a insetos (Post et al. 1993, Logan 1999). Alguns métodos de preservação têm sido testados para insetos, como solução de Carnoy, silica gel, etanol, metanol, propanol, etil acetato, etileno glicol, formalina, secagem ao ar, amil acetato, xileno, congelamento, acetona, tampão de extração, entre outros (Post et al. 1993, Reis et al. 1995, Dillon et al. 1996, Austin & Dillon 1997, Logan 1999), mas os resultados mostraram certas diferenças dependendo do grupo de insetos ou mesmo do estádio de desenvolvimento estudado, o que dificulta e torna questionável uma extrapolação de resultados entre diferentes grupos taxonômicos (Dillon et al. 1996).
O presente estudo teve por objetivo comparar alguns métodos simples de preservação de espécimes da cigarrinha-do-milho, Dalbulus maidis (DeLong & Wolcott), visando estudos de variabilidade genética em populações desse inseto, que é um importante vetor de patógenos associados ao enfezamento do milho na América Latina. Avaliou-se a eficiência quantitativa e qualitativa de preservação do DNA dos espécimes por cada um dos métodos, após sucessivos períodos de armazenamento, bem como a performance das amostras em relação à técnica de RAPD-PCR.
Material e Métodos
Os espécimes de D. maidis utilizados neste estudo foram obtidos de uma colônia formada a partir de indivíduos coletados em Piracicaba (SP) e mantida sobre plântulas de milho, em laboratório, por mais de 15 gerações. Todos os espécimes foram avaliados previamente quanto à possível ocorrência de parasitóides, através da observação da presença de manchas protuberantes de coloração negra ou vermelha no abdome ou tórax, ou de dilatação do abdome (Moya-Raygoza & Trujillo-Arriaga 1993).
Métodos de Preservação. Oito métodos de preservação foram utilizados neste estudo. Espécimes de D. maidis foram coletados vivos, mortos com CO2 e submetidos, simultaneamente, a cada um dos seguintes métodos de preservação: (A) congelamento em freezer (-20oC); (B) álcool etílico 70% (temperatura ambiente); (C) álcool etílico 70% (-20oC); (D) álcool etílico absoluto (temperatura ambiente); (E) álcool etílico absoluto (-20oC); (F) insetos mantidos a seco em placa de Petri (temperatura ambiente); (G) insetos inteiros mantidos individualmente em tubos de microcentrífuga com 300 ml de tampão de extração (200 mM Tris-HCl, pH 8,0; 250 mM NaCl; 25 mM EDTA e 1% SDS) (temperatura ambiente); e (H) insetos macerados mantidos individualmente em tubos de microcentrífuga com 300 ml de tampão de extração (temperatura ambiente). Nos tratamentos de (A) a (E), os insetos foram acondicionados em um tubo de microcentrífuga.
Extração do DNA. A intervalos de tempo de 10 a 30 dias, três espécimes de D. maidis de cada um dos oito métodos de preservação foram retirados e submetidos à extração do DNA genômico total, seguindo-se o protocolo modificado de Raeder & Broda (1985). Os espécimes foram colocados em tubos de microcentrífuga (1,5 ml) e macerados manualmente em presença de 300 ml de tampão de extração, utilizando-se pistilos plásticos, cuja extremidade tinha o formato exato do fundo dos tubos de microcentrífuga de 1,5 ml. Após incubação a 65oC por 30 min, foram feitas três extrações orgânicas com fenol, fenol:clorofórmio:álcool isoamílico (25:24:1) e clorofórmio:álcool isoamílico (24:1). Nas três extrações, acrescentou-se 300 ml de cada solvente, centrifugou-se a 14.800 g por 10 min e recolheu-se o sobrenadante. O DNA foi precipitado da fase aquosa ao final da extração, pela adição de igual volume de álcool isopropílico e de NaCl para uma concentração final de 0,3 M. As amostras foram mantidas em freezer (-20oC) por, aproximadamente, 12h. A sedimentação foi feita por centrifugação a 14.800 g por 10 min. O DNA foi lavado com álcool etílico 70%, seco ao ar, ressuspendido em 20 ml de TE (10 mM de Tris e 1 mM de EDTA; pH 8,0) e armazenado em freezer (-20oC) até a quantificação. No caso dos métodos de preservação que envolveram o uso de álcool como fixador, os espécimes foram submetidos a um pré-tratamento para reidratação antes da extração, que consistiu na lavagem dos espécimes em água destilada por 5 min e em TE por, aproximadamente, 1,5h sob agitação.
O DNA total de cada espécime foi quantificado pela fluorescência de uma alíquota de 2 ml de DNA extraído após mistura com o pigmento Hoescht 33258 Dye (Cesarone et al. 1979), utilizando-se fluorímetro Versa Fluor (Bio-Rad Laboratories, Hercules, CA 94547, USA). Após a quantificação, as amostras de DNA foram diluídas em TE para 3 ng/ml. Quantidades muito pequenas de DNA nas amostras (geralmente <1 ng) não puderam ser medidas com precisão pelo fluorímetro e ocasionalmente foram obtidas leituras negativas. Nestes casos, a quantidade de DNA foi corrigida para zero. Para a observação da integridade do DNA extraído, uma alíquota de 1 ml do DNA total das amostras (antes da diluição), foi misturada a 6 ml de tampão de corrida e analisada por eletroforese em tampão 1X TBE (100 mM Tris; 83 mM H3Bo3 e 1 mM EDTA). A eletroforese foi conduzida a 3,5 V/cm por 1 h, em gel de agarose 0,8% corado com brometo de etídio (1 mg/ml de gel) e fotografado sob luz U.V. com filme branco e preto NeoPan asa 100 (Fuji Photo Film da Amazônia Ltda., Manaus, AM 69075-000, Brasil). Nesse gel, além de amostras de DNA referentes aos tratamentos, foi incluída uma amostra contendo DNA extraído de espécime fresco.
Amplificação via RAPD-PCR. Nas reações RAPD utilizou-se o oligonucleotídeo OPA-04 (5'-AATCGGGCTG-3') (Operon Technologies, Alameda, CA 94501, USA). A amplificação foi realizada em um volume final de 20 ml, contendo 6 ng de DNA, 4 mM de MgCl2, 0,25 mM de dNTP, 0,3 mM do primer e 1 unidade de Taq DNA polimerase, adicionando-se ao final 30 ml de óleo mineral. Como controle negativo foi utilizada uma amostra que continha todos os reagentes, exceto DNA de cigarrinha, e como controle positivo utilizou-se DNA de espécime fresco. As reações foram realizadas em termociclador PTC-100 (MJ Research, Inc., Watertown, MA 02172, USA) nas seguintes condições de amplificação: 92oC por 5 min (1 ciclo), 92oC por 40 s, 40oC por 1,5 min e 72oC por 2 min (40 ciclos) e 72oC por 5 min (1 ciclo).
Após a adição de 3 ml do tampão de corrida, 20 ml das amostras amplificadas foram aplicados em gel de agarose 1,4%, procedendo-se a eletroforese em tampão 1X TBE por 3,5h. A eletroforese e a captura fotográfica foram efetuadas como descrito anteriormente. Realizou-se também a captura pelo sistema de documentação de gel "Eagle Eye II" (Stratagene, La Jolla, CA 92037, USA), para estimativa dos pesos moleculares dos locos amplificados. Foram realizadas três repetições para cada amplificação RAPD-PCR.
Análise dos Dados. Com os dados de quantidade de DNA total de cada espécime, para cada uma das datas de extração, realizou-se análise de variância. As médias foram comparadas pelo teste de Tukey (P£ 0,05), através do programa "Statistical Analysis System" V. 6.08 (SAS Institute 1993). Devido à heterogeneidade de variância, os dados de cada data de extração foram submetidos a um tipo de transformação que melhor os ajustassem ao modelo normal. As transformações utilizadas em cada data de extração foram: log (x+1) para 1, 20, 30, 40, 50, 60, 90, 100, 140 e 160 dias; (x+1)-2 para 80 e 210 dias;  para 120 e 180 dias;
para 120 e 180 dias; para 10 dias e
para 10 dias e  para 70 dias (SAS Institute 1993).
para 70 dias (SAS Institute 1993).
Resultados
Baseando-se nas leituras obtidas pelo fluorímetro (Tabelas 1 e 2), o método de extração utilizado neste estudo mostrou-se eficiente para D. maidis, já que as quantidades de DNA obtidas foram suficientes para realização de análises por RAPD-PCR.
A análise de variância, seguida do teste de Tukey, mostrou que com um dia de armazenamento todos os tratamentos forneceram quantidades semelhantes de DNA (Tabela 1). A partir dos 10 dias, a quantidade de DNA extraído de material seco ao ar mostrou-se estatisticamente inferior a dos demais métodos de preservação, não diferindo apenas de álcool etílico 70% e absoluto (temperatura ambiente) a partir dos 90 e 140 dias, respectivamente (Tabela 2). Os métodos de congelamento, tampão de extração (inseto macerado e inteiro), álcool etílico 70% e absoluto (ambos a -20oC) propiciaram quantidades de DNA consistentemente superiores a dos demais tratamentos a partir de 120 dias de armazenamento. Tais métodos não diferiram estatisticamente entre si na maioria das datas de avaliação, havendo poucas exceções. Por exemplo, o método de congelamento foi estatisticamente superior ao tampão de extração (inseto inteiro) aos 60, 90 e 100 dias, ao tampão de extração (inseto macerado) aos 100 dias, e ao álcool etílico absoluto (-20oC) aos 60 dias (Tabelas 1 e 2). Em geral, o congelamento dos insetos propiciou quantidades médias de DNA numericamente maiores do que a dos demais métodos, o que não ocorreu apenas em quatro datas de extração (1, 20, 30 e 50 dias).
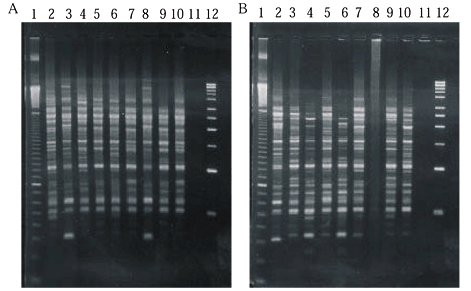
Um exemplo representativo da qualidade do DNA extraído pode ser observado na Fig. 1. Com um dia após o início do estudo, observou-se uma boa qualidade do DNA extraído em todos os tratamentos (colunas 7-14). Entretanto, nenhuma banda pôde ser visualizada no gel para os tratamentos álcool etílico 70% (colunas 16, 24 e 32) e álcool etílico absoluto (colunas 18, 26 e 34) sob temperatura ambiente, bem como para espécimes secos ao ar (colunas 20, 28 e 36), após 70, 120 e 210 dias, devido à baixa quantidade de DNA obtida ou à degradação total do ácido nucléico nesses tratamentos.
Com exceção do tratamento em que os espécimes foram conservados a seco, onde a partir dos 10 dias não foram observados produtos de amplificação (Fig. 2B, 2C e 2D, coluna 8), todos os tratamentos mostraram amplificação via RAPD-PCR bastante satisfatória e semelhantes à do DNA extraído de espécime fresco (Fig. 2). Um número razoável de locos gênicos foi amplificado de forma consistente, com pesos moleculares estimados entre 200 e 2.600 pares de bases.
Discussão
A técnica de RAPD-PCR é uma importante ferramenta na análise da estrutura e diversidade genética em populações naturais (Ferreira & Grattapaglia 1996). Diversos estudos baseados em marcadores RAPD-PCR com populações de insetos oriundos do campo têm sido realizados, como por exemplo, análise da diversidade genética entre e dentro de populações do pulgão Diuraphis noxia (Mordivilko) (Puterka et al. 1993), dentro de populações da cigarrinha Empoasca fabae (Harris) (Skinner & Camacho 1995), entre as espécies Myzus persicae (Sulzer) e Myzus nicotianae Blackman (Margaritopoulos et al. 1998) e detecção de polimorfismo genético em diversas espécies de pulgões (Black et al. 1992). Mas nem sempre é possível obter material fresco para tais estudos, como por exemplo quando se coleta materiais de regiões distantes (Reiss et al. 1995), ou mesmo quando a análise do material não pode ser realizada imediatamente. Muitas técnicas de preservação comumente utilizadas podem não conservar a integridade do DNA, que é um ponto crítico em muitas técnicas de análise genética.
Para ensaios RAPD-PCR utilizando-se D. maidis, quantidades mínimas de DNA, da ordem de 3 ng/ml, são suficientes para uma amplificação consistente (Oliveira et al. dados não publicados). No entanto, sabendo que esses ensaios requerem a utilização de vários "oligonucleotídeos" e computando-se as perdas durante a manipulação das amostras de DNA, neste estudo só foram consideradas satisfatórias as extrações que forneceram no mínimo 160 ng/20 ml de TE. A variação na quantidade de DNA obtida de espécime para espécime dentro dos tratamentos em cada uma das datas de extração pôde, em parte, ser explicada pelo fato de que os indivíduos foram tomados ao acaso. Como as fêmeas de D. maidis são significativamente maiores que os machos (Oliveira 1996), maiores quantidades de DNA são esperadas para as mesmas, como demonstraram Post et al. (1993) estudando o díptero Simulium damnosum Theobald.
A exemplo de resultados negativos obtidos da extração de DNA de insetos de museu (Post et al. 1993, Dillon et al. 1996), a manutenção de espécimes secos ao ar mostrou-se pouco apropriada como método de conservação de D. maidis, ocorrendo uma drástica degradação do DNA genômico total nos primeiros 10 dias após a morte dos espécimes, com degradação total após esse período (Fig. 1, colunas 20, 28 e 36).
A preservação em álcool etílico 70% sob temperatura ambiente propiciou quantidades satisfatórias de DNA até os 60 dias, enquanto que o armazenamento em álcool etílico absoluto, também à temperatura ambiente, mostrou-se adequado até os 120 dias (Tabelas 1 e 2). Para tecido de mamíferos, Smith et al. (1987) relataram a conservação do DNA em álcool etílico por mais de 5 anos, mas este não parece ser o comportamento do DNA de insetos preservados em álcool (Post et al. 1993). Reiss et al. (1995) constataram para o coleóptero Amara glacialis (Mannerheim), fixado em álcool etílico 95%, um período de preservação do DNA de 73 dias. Para os espécimes de D. maidis preservados em álcool etílico à temperatura ambiente, observou-se um gradiente de degradação ao longo do tempo, função da atividade contínua de nucleases (Dessauer et al. 1996), sendo este mais acentuado para álcool etílico 70%. Algumas variáveis têm se mostrado importantes na quantidade e qualidade de DNA extraído de materiais preservados em álcool etílico, tais como a concentração do álcool, temperatura e tempo de armazenamento (Dillon et al. 1996).
Os métodos álcool etílico absoluto (-20oC), álcool etílico 70% (-20oC) e espécimes conservados em tampão de extração (macerados ou inteiros) permitiram a extração de quantidades satisfatórias de DNA (>160 ng/20 ml) de alta qualidade em todas as datas de avaliação. A preservação de material em álcool etílico a baixas temperaturas tem sido apontada por alguns autores como um método eficiente de preservação do DNA de diversos grupos de insetos por longos períodos de tempo (Post et al. 1993, Dillon et al. 1996). Reiss et al. (1995) afirmaram que tampão de extração é um método adequado de preservação de DNA, desde que o inseto seja bem homogeneizado. Neste estudo, não houve diferença estatística entre as quantidades de DNA extraídas de inseto armazenado inteiro e de inseto homogeneizado em tampão de extração, sendo ambos os métodos satisfatórios para a preservação de DNA de D. maidis (Tabelas 1 e 2; Fig. 1).
O método de congelamento (-20oC) foi a forma mais eficiente de armazenamento de espécimes de D. maidis, preservando a integridade e fornecendo maiores quantidades de DNA do que os demais tratamentos na maioria das datas de avaliação (Tabelas 1 e 2; Fig. 1). Resultados semelhantes foram obtidos em outros trabalhos envolvendo os mais diferentes grupos de insetos, como Diptera (Post et al. 1993), Coleoptera (Reiss et al. 1995) e Hymenoptera (Dillon et al. 1996).
As amostras de DNA extraídas nas diferentes datas permitiram, com exceção do material seco ao ar a partir dos 10 dias de armazenamento, a amplificação via RAPD-PCR (Fig. 2). Para os tratamentos álcool etílico 70% e absoluto (temperatura ambiente), a não observação de banda correspondente ao DNA genômico total no gel de agarose 0,8% aos 70, 120 e 210 dias (Fig. 1, colunas 16, 18, 24, 26, 32 e 34) se deveu a baixas quantidades de DNA extraído, que a despeito disto foram detectadas pelo fluorímetro (Tabelas 1 e 2) e permitiram amplificação (Fig. 2B, 2C e 2D). Porém, para álcool etílico 70% após 60 dias e álcool etílico absoluto a partir dos 120 dias, a quantidade de DNA extraído (<160 ng/20 ml), apesar de permitir amplificação, mostrou-se inviável para estudos de RAPD-PCR pelo baixo volume de DNA ressuspendido.
De forma prática, para o intercâmbio de espécimes de D. maidis visando análise genética ou mesmo quando da coleta de espécimes em locais remotos, pode-se recomendar a preservação em álcool etílico absoluto por até 120 dias, que mostrou ser um método bastante simples, de baixo custo, podendo ser utilizado diretamente no campo (Reiss et al. 1995). Outra possibilidade é a preservação dos espécimes em tampão de extração (inseto macerado ou inteiro), que permitiu a conservação do DNA por até 210 dias sem a necessidade de refrigeração. A desvantagem, porém, é que materiais assim preservados não permitiriam estudos dos aspectos morfológicos ou a verificação da presença de parasitóides (Post et al. 1993), uma vez que o material é homogeneizado. Mesmo com o inseto inteiro, devido à ocorrência de lise das células, os espécimes tornariam-se inadequados para tais estudos, que via de regra são auxiliares à análise genética e permitem a correta identificação taxonômica do inseto.
No caso de necessidade de conservação dos espécimes em laboratório, sem dúvida, a prática mais recomendada é o congelamento (-20oC ou menos) (Reiss et al. 1995, Dillon et al. 1996), por manter a integridade e propiciar maiores quantidades de DNA. Entretanto, outros métodos como álcool etílico absoluto e 70% (-20oC), ou a conservação em tampão de extração também podem ser utilizados.
Agradecimentos
Os autores agradecem a Silvia Monteil Gomes, Américo Iorio Ciociolla e Marta Rocio Bressan Smith (ESALQ/USP) pelas informações preliminares sobre esse assunto e à Coordenação de Aperfeiçoamento de Pessoal de Ensino Superior (CAPES) pela concessão da bolsa de estudo ao primeiro autor.
Literatura Citada
Received 22/09/00. Accepted 12/04/02.
O arquivo disponível sofreu correções conforme ERRATA publicada no Volume 31 Número 3 da revista.
- Austin, A.D. & N. Dillon. 1997. Extraction and PCR of DNA from parasitoid wasps that have been chemically dried. Aust. J. Entomol. 36: 241-244.
- Black, W.C.I.V., N.M. DuTeau, G.J. Puterka, J.R. Nechols & J.M. Pettorini. 1992. Use of the random amplified polymorphic DNA polymerase chain reaction (RAPD-PCR) to detect DNA polymorphisms in aphids (Homoptera: Aphididae). Bull. Entomol. Res. 82: 151-159.
- Bramwell, N.H. & B.F. Burns. 1988. The effects of fixative type and fixation time on the quantity and quality of extractable DNA for hybridisation studies on lynphoid tissue. Expl. Hematol. 16: 730-732.
- Cesarone, C.F., C. Bolognesi & L. Santi. 1979. Improved microfluorometric DNA determination in biological material using Hoescht 33258. Analyt. Biochem. 100: 188-197.
- Dessauer, H.C., C.J. Cole & M.S. Hafner. 1996. Collection and storage of tissues, p. 25-41. In D. M. Hillis, C. & Mortis B. K. Mable (eds.), Molecular systematics, Sunderland: Sinauer, 655p.
- Dillon, N., A.D. Austin & E. Bartowsky. 1996. Comparison of preservation techniques for DNA extraction from hymenopterous insects. Insect Molec. Biol. 5: 21-24.
- Ferreira, E.F. & D.Grattapaglia. 1996. Introdução ao uso de marcadores moleculares em análise genética. 2 ed. Brasília: Embrapa-Cenargen, 220
- Goelz, S.E., S.R. Hamilton & B. Vogelstein. 1985. Purification of DNA from formaldehyde fixed and paraffin embedded human tissue. Biochem. Biophys. Res. Commun. 130: 118-126.
- Logan, J.A. 1999. Extraction, polymerase chain reaction, and sequencing of a 440 base pair region of the mitocondrial cytochrome oxidase I gene from two species of acetone-preserved damselflies (Odonata: Coenagrionidae, Agrionidae). Environ. Entomol. 28: 143-147.
- Margaritopoulos, J.T., Z. Mamuris & J.A. Tsitsipis. 1998. Attempted discrimination of Myzus persicae and Myzus nicotianae (Homoptera: Aphididae) by random amplified polymorphic DNA polimerase chain reaction technique. Ann. Entomol. Soc. Am. 91: 602-607.
- Moya-Raygoza, G. & J. Trujillo-Arriaga. 1993. Dryinid (Hym.: Dryinidae) parasitoids of Dalbulus leafhopper (Hom.: Cicadellidae) in México. Entomophaga 38: 41-49.
- Oliveira, C.M 1996. Variação morfológica entre populações de Dalbulus maidis (DeLong & Wolcott, 1923) (Hemiptera: Cicadellidae) de algumas localidades do Brasil. Dissertação de mestrado. ESALQ, USP, Piracicaba, 69p.
- Post, R.J., P.K. Flook & A.L. Millest. 1993. Methods for the preservation of insects for DNA studies. Biochem. Syst. Ecol. 21: 85-92.
- Puterka, G.J., W. C. IV. Black, W M. Steiner & R. L. Burton. 1993. Genetic variation and phylogenetic relationships among worldwide collections of the Russian wheat aphid, Diuraphis noxia (Mordvilko), inferred from allozyme and RAPD-PCR markers. Heredity 70: 604-618.
- Raeder, U. & P. Broda. 1985. Rapid preparation of DNA from filamentous fungi. Let. Appl. Microbiol. 1: 17-20.
- Reiss, R.A., D.P. Schwert & A.C. Ashworth. 1995. Field preservation of Coleoptera for molecular genetic analyses. Environ. Entomol. 24: 716-719.
- Saiki, R.K., D.H. Gelfand, S. Stoffel, S. Scharf, R.H. Higushi, G.T. Horn, K.B. Mullis & H.A. Erlich. 1988. Primer-directed enzymatic amplification of DNA with a thermostable DNA polymerase. Science 239: 487-491.
- SAS institute. 1993. SAS/STAT user's guide: release 6.08 8 ed. Cary.
- Skinner, D.Z. & R.F. Camacho. 1995. Genetic diversity within a potato leafhopper (Homoptera: Cicadellidae) population infesting alfalfa. J. Kansas Entomol. Soc. 68: 35-42.
- Smith, L.J., R.C. Brayland, J.E. Nutkis, K.B. Edmundson, J.R. Downing & E.K. Wakeland. 1987. Extraction of cellular DNA from human cells and tissues fixed in ethanol. Analyt. Biochem. 160: 135-138.
- Welsh, J. & M. McClelland. 1990. Fingerprinting genomes using PCR with arbitrary primers. Nucleic Acids Res. 18: 7213-7218.
- Williams, J.G.K, A.R. Kubelik, K.J. Livak, J.A. Rafalski & S.V. Tingey. 1990. DNA polymorphism amplified by arbitrary primers are useful as genetic markers. Nucleic Acids Res. 18: 6531-6535.
Datas de Publicação
-
Publicação nesta coleção
20 Jan 2003 -
Data do Fascículo
Jun 2002
Histórico
-
Aceito
12 Abr 2002 -
Recebido
22 Set 2000