Resumos
A grande diversidade genética dos agentes causadores da mancha-bacteriana dificulta sobremaneira o desenvolvimento de variedades de pimentão e tomate com resistência durável. Setenta e dois isolados de Xanthomonas spp. provenientes de campos comerciais de tomate para processamento industrial dos estados de Goiás, Minas Gerais, Pernambuco e Bahia foram classificados em raças com base nas reações de genótipos diferenciais de tomateiro (Walter, Hawaii 7998 e NIL 216) e de Capsicum (ECW [Early Calwonder], ECW-10R, ECW-20R, ECW-30R e PI235047). As plantas foram inoculadas no estádio de três a cinco folhas verdadeiras por infiltração de suspensão bacteriana (5 ´ 10(8) UFC/ml) na superfície abaxial da folha. Em seguida, foram mantidas em câmara de crescimento em fotoperíodo de 12 h/12 h (luz/escuro) a 28ºC. A reação de hipersensibilidade foi observada até 36 horas após a inoculação, dependendo do genótipo da hospedeira. Foram identificadas as raças T1P2, T1P8 e T3 em X. axonopodis pv. vesicatoria; a raça T2 em X. vesicatoria; e as raças T2P7 e T2P8 em X. gardneri. A presença dos genes avrRxv e avrXv3 nos isolados que causaram reação de hipersensibilidade em 'Hawaii 7998' (raça T1) e 'NIL 216' (raça T3), respectivamente, foi confirmada por reação em cadeia da polimerase (PCR) usando iniciadores específicos. Este é o primeiro relato da ocorrência no Brasil das raças T3, T1P8, T2P7 e T2P8.
Lycopersicon esculentum; Xanthomonas campestris pv. vesicatoria; X. axonopodis pv. vesicatoria; X. vesicatoria; X. gardneri; resistência genética
The great genetic diversity of the causal agents of bacterial spot is the main problem to the development of tomato and pepper varieties with durable resistance. Seventy two strains of Xanthomonas spp. collected from commercial fields of processing tomatoes in the states of Goiás, Minas Gerais, Pernambuco, and Bahia were classified in races according to their reactions on differential genotypes of tomato (Walter, Hawaii 7998 and NIL 216) and Capsicum [ECW (Early Calwonder), ECW-10R, ECW-20R, ECW-30R and PI 235047]. Bacterial suspensions (5 ´ 10(8) UFC/ml) were infiltrated in the abaxial leaf face of the plants at the three to five true-leaf stage. The plants were then kept in a growth chamber at 28ºC and a 12-h light/dark photoperiod. The response reactions were observed up to 36 hours after inoculation, depending on the genotype. Races T1P2, T1P8 and T3 were identified in X. axonopodis pv. vesicatoria; race T2 in X. vesicatoria; and races T2P7 e T2P8 in 'X. gardneri'. The presence of genes avrRxv and avrXv3 was confirmed by polymerase chain reaction (PCR) with specific primers in strains that produced hypersensitive reaction on 'Hawaii 7998' (races T1) and 'NIL 216' (race T3), respectively. This is the first report of races T3, T1P8, T2P7 and T2P8 in Brazil.
Lycopersicon esculentum; Xanthomonas campestris pv. vesicatoria; X. axonopodis pv. vesicatoria; X. vesicatoria; X. gardneri; genetic resistance
PESQUISA
Raças de Xanthomonas spp. associadas à mancha-bacteriana em tomate para processamento industrial no Brasil
Races of Xanthomonas spp. associated to bacterial spot in processing tomatoes in Brazil
Alice Maria Quezado-DuvalI; Luis Eduardo A. CamargoII
IEmbrapa Hortaliças, C. Postal 218, 70359-970 Brasília-DF; E-mail: alice@cnph.embrapa.br
IIUSP-ESALQ, C. Postal 09, 13418-900 Piracicaba-SP; E-mail: leacamar@carpa.ciagri.usp.br
RESUMO
A grande diversidade genética dos agentes causadores da mancha-bacteriana dificulta sobremaneira o desenvolvimento de variedades de pimentão e tomate com resistência durável. Setenta e dois isolados de Xanthomonas spp. provenientes de campos comerciais de tomate para processamento industrial dos estados de Goiás, Minas Gerais, Pernambuco e Bahia foram classificados em raças com base nas reações de genótipos diferenciais de tomateiro (Walter, Hawaii 7998 e NIL 216) e de Capsicum (ECW [Early Calwonder], ECW-10R, ECW-20R, ECW-30R e PI235047). As plantas foram inoculadas no estádio de três a cinco folhas verdadeiras por infiltração de suspensão bacteriana (5 ´ 108 UFC/ml) na superfície abaxial da folha. Em seguida, foram mantidas em câmara de crescimento em fotoperíodo de 12 h/12 h (luz/escuro) a 28ºC. A reação de hipersensibilidade foi observada até 36 horas após a inoculação, dependendo do genótipo da hospedeira. Foram identificadas as raças T1P2, T1P8 e T3 em X. axonopodis pv. vesicatoria; a raça T2 em X. vesicatoria; e as raças T2P7 e T2P8 em X. gardneri. A presença dos genes avrRxv e avrXv3 nos isolados que causaram reação de hipersensibilidade em 'Hawaii 7998' (raça T1) e 'NIL 216' (raça T3), respectivamente, foi confirmada por reação em cadeia da polimerase (PCR) usando iniciadores específicos. Este é o primeiro relato da ocorrência no Brasil das raças T3, T1P8, T2P7 e T2P8.
Palavras-chave:Lycopersicon esculentum, Xanthomonas campestris pv. vesicatoria, X. axonopodis pv. vesicatoria, X. vesicatoria, X. gardneri, resistência genética.
ABSTRACT
The great genetic diversity of the causal agents of bacterial spot is the main problem to the development of tomato and pepper varieties with durable resistance. Seventy two strains of Xanthomonas spp. collected from commercial fields of processing tomatoes in the states of Goiás, Minas Gerais, Pernambuco, and Bahia were classified in races according to their reactions on differential genotypes of tomato (Walter, Hawaii 7998 and NIL 216) and Capsicum [ECW (Early Calwonder), ECW-10R, ECW-20R, ECW-30R and PI 235047]. Bacterial suspensions (5 ´ 108 UFC/ml) were infiltrated in the abaxial leaf face of the plants at the three to five true-leaf stage. The plants were then kept in a growth chamber at 28ºC and a 12-h light/dark photoperiod. The response reactions were observed up to 36 hours after inoculation, depending on the genotype. Races T1P2, T1P8 and T3 were identified in X. axonopodis pv. vesicatoria; race T2 in X. vesicatoria; and races T2P7 e T2P8 in 'X. gardneri'. The presence of genes avrRxv and avrXv3 was confirmed by polymerase chain reaction (PCR) with specific primers in strains that produced hypersensitive reaction on 'Hawaii 7998' (races T1) and 'NIL 216' (race T3), respectively. This is the first report of races T3, T1P8, T2P7 and T2P8 in Brazil.
Keywords:Lycopersicon esculentum, Xanthomonas campestris pv. vesicatoria, X. axonopodis pv. vesicatoria, X. vesicatoria, X. gardneri, genetic resistance.
A resistência varietal é uma importante ferramenta para o controle de doenças de plantas, notadamente as causadas por bactérias fitopatogênicas onde o controle químico é muitas vezes ineficaz (Lopes e Quezado-Soares, 1997). A eficiência e durabilidade da resistência depende, entre outros fatores, da variabilidade genética do patógeno. Variações em patogenicidade expressas em termos de raças fisiológicas ocorrem em diversos patógenos, evoluindo freqüentemente nas populações em resposta à introdução de genes de resistência dominantes de efeito qualitativo (Crute e Pink, 1996).
No complexo etiológico da mancha-bacteriana das solanáceas causado por espécies de Xanthomonas (Jones et al., 2000), já foram identificadas três raças em relação ao gênero Lycopersicon (raças T) (Wang et al., 1990; Jones et al., 1995) e onze em relação a Capsicum (raças P) (Kousik e Ritchie, 1995; Ritchie et al., 1998; Sahin e Miller, 1995). Diferentes combinações entre as raças T e P também foram observadas em isolados que infectam as duas hospedeiras (Bouzar et al., 1994; Jones et al., 1995; Jones et al., 1998).
As raças são definidas com base em reações de um grupo definido de genótipos das hospedeiras (Leach e White, 1996). Algumas das interações observadas entre as espécies de Xanthomonas associadas à mancha-bacteriana e as hospedeiras Lycopersicon e Capsicum seguem o modelo gene-a-gene proposto por Flor na década de 40 (Astua-Monge et al., 2000a; Romero et al., 2002). No modelo, genes de avirulência (avr) do patógeno interagem com genes de resistência da hospedeira, resultando em uma reação de incompatibilidade, ou seja, de resistência (Leach e White, 1996).
Como exemplos que seguem o modelo no patossistema em questão cita-se: a) a interação entre os genes Bs1, Bs2 e Bs3, presentes em genótipos de Capsicum, e os respectivos genes avrBs1, avrBs2 e avrBs3 (Stall, 1997); b) entre o gene Xv3 encontrado nos genótipos Hawaii 7981 (L. esculentum), PI 128216 e PI 126932 (ambos L. pimpinellifolium) e o gene avrXv3; c) entre o gene Xv4 de 'LA716' (L. pennellii) e o gene avrXv4, ambos presentes na raça T3 (Astua-Monge et al., 2000a; Astua-Monge et al., 2000b) e d) avrBs4 (=avrBsP sensu Canteros et al., 1991 e avrBs3-2 sensu Bonas et al., 1993), que induz incompatibilidade em acessos de tomate portando o gene Bs4 (Ballvora, et al., 2001).
Existem ainda algumas interações cujos genes ainda não foram identificados (gene de avirulência e/ou de resistência) e outras que envolvem mais de um gene de resistência. No primeiro caso tem-se o gene avrBsT, que induz incompatibilidade em Capsicum (Minsavage et al., 1990) e os genes envolvidos na interação entre C. pubescens e certas raças P (Sahin e Miller, 1998). No segundo caso, cita-se a interação entre o gene avrRxv da raça T1 (Whalen et al., 1988; Whalen et al., 1993) com pelo menos três genes de resistência não-dominantes (rx1, rx2 e rx3) do genótipo 'Hawaii 7998' (Wang et al., 1994; Yu et al., 1995) que resulta em reação de hipersensibilidade (Jones e Scott, 1986). Por outro lado, para a raça T2 não existem fontes conhecidas de resistência qualitativa (Scott et al., 1997; Wang et al., 1990).
Isolados da raça T1 têm sido agrupados no grupo fenotípico "A" (= X. axonopodis pv. vesicatoria sensu Vauterin et al., 1995); da raça T2 no grupo "B" (= X. vesicatoria sensu Vauterin et al., 1995) ou no grupo "D" (= X. gardneri de acordo com Jones et al., 2000) e da raça T3, no grupo "C" (= subgrupo de X. axonopodis pv. vesicatoria conforme Jones et al., 2000). No relato de Bouzar et al. (1994), isolados brasileiros foram identificados como raças T1P0, T1P2, T2, T2P1 e T2P3.
Este trabalho teve como objetivo determinar as raças de Xanthomonas spp. associadas à mancha-bacteriana em relação a genótipos diferenciais de Lycopersicon esculentum e de Capsicum spp. presentes em amostras de isolados provenientes de campos de tomateiro destinados ao processamento industrial no Brasil.
MATERIAL E MÉTODOS
Obtenção, manutenção e identificação dos isolados
Foram analisados 72 isolados de Xanthomonas spp. coletados a partir de 1995 em 31 campos comerciais de tomate para processamento industrial localizados nos estados de Goiás, Minas Gerais, Pernambuco e Bahia (Tabela 1). Os isolados foram previamente classificados em X. axonopodis pv. vesicatoria (37 do grupo PFGE "A" e 15 do grupo PFGE "C" sensu Jones et al., 2000), X. vesicatoria (9 do grupo PFGE "B") ou X. gardneri (11 do grupo PFGE "D") por meio de análise de perfis genômicos através da eletroforese de campo pulsado, testes de patogenicidade, atividades amidolíticas e pectolíticas e utilização de fontes de carbono (Quezado-Duval, 2003).
Identificação das raças
Foram utilizados os genótipos diferenciais de tomateiro Walter, Hawaii 7998 e NIL 216 (linha quase isogênica ao genótipo suscetível Fla. 7060 com gene de resistência derivado do genótipo PI 128216 [Astua-Monge et al., 2000a]), e de Capsicum annum ECW (Early Calwonder), ECW-10R, ECW-20R, ECW-30R e C. pubescens PI 235047. As sementes foram obtidas dos bancos de germoplasma de tomateiro e de pimentas/pimentão da Embrapa Hortaliças, Brasília. O semeio foi em bandejas de poliestireno de 128 células preenchidas com substrato agrícola Plantmax® (Eucatex). O transplante para sacos de plástico preto com capacidade de 1,5 L, preenchidos com solo esterilizado, foi feito quando as plantas apresentavam de duas a três folhas verdadeiras.
Para a obtenção das culturas bacterianas, os isolados armazenados em tampão fosfato pH 7,0 foram recuperados em meio Ágar-Nutriente após incubação no escuro em câmara com temperatura controlada a 28ºC por 5 dias. Uma massa bacteriana foi então transferida com o auxílio de uma alça de platina para tubos de plástico de fundo cônico com capacidade para 50 ml contendo 15 ml de meio Caldo-Nutriente (Sigma Chemical Co., St. Louis, MO, EUA) e homogeneizada em agitador de tubos. O meio foi colocado em incubadora refrigerada MA 830 com agitador orbital (Marconi, Piracicaba, SP) a 148 rotações por minuto a 28ºC por cerca de 16 horas. As suspensões obtidas após o período de incubação foram então centrifugadas em centrífuga Beckman GS-6 (Palo Alto, CA, EUA), rotor GH3.8, a 2.800 × g por 20 minutos e o sedimentado resultante re-suspendido em água destilada a uma concentração de 5×108 UFC/ml segundo, leitura (A600=0,3), em espectrofotômetro Ultrospec® 3100 pro (Amersham Pharmacia Biotech, Uppsala, Sweden). As plantas foram inoculadas no estádio de seis a sete folhas verdadeiras (cerca de 7 a 8 semanas após o semeio de pimentão e de tomate, respectivamente), através da infiltração de suspensão bacteriana (5 ´ 108 UFC/ml) na superfície abaxial da folha. Durante esse período, as plantas foram mantidas em câmara de crescimento em fotoperíodo de 12h/12h (luz/escuro) a 28ºC. A reação de hipersensibilidade foi caracterizada pela necrose dos tecidos em até 36 horas após a inoculação, dependendo do genótipo da hospedeira (6-12 horas para ECW-10R; 12-24 horas para ECW-20R; 24-36 horas para ECW-30R e aproximadamente 24 horas para PI 235047, 'Hawaii 7998' e 'NIL 216' [Jones et al., 1995; Sahin e Miller, 1998; Stall, 1997]).
Presença dos genes avrRxv e avrXv3
A presença dos genes avrRxv e avrXv3foi confirmada pela reação em cadeia da polimerase (PCR) em todos os isolados que provocaram resposta de hipersensibilidade nos genótipos diferenciais Hawaii 7998 e NIL 216, respectivamente. DNA genômico total foi isolado pelo método de purificação CTAB (brometo de amônio-hexadeciltrimetil) (Wilson, 1999). Para a amplificação do fragmento de 680-pb contendo o gene avrRxv , a reação da PCR, em um volume final de 50 mL, foi composta por 100 ng do DNA molde, 5 mL de tampão de amplificação (Promega Life Science, Madison, WI, EUA), 1,5 mM de MgCl2, 1,25 U de Taq DNA polimerase (Promega), 100 mM de cada deoxinucleosídeo trifosfato (dNTPs) e 0,5 mM dos iniciadores RST27 (5'-AGTCGCGCGGACATTTAGCCCCGC C) e RST28 (5'-CGTCGATGGTGCGCCTGGAATGCGC) (Bouzar et al., 1993). O programa de amplificação constou de 30 ciclos, realizados em um termociclador PTC-100 MJ Research Inc. (Watertown, MA, EUA), nas seguintes condições: desnaturação a 95ºC por 30 segundos, hibridização a 64ºC por 30 segundos e extensão a 72ºC por 45 segundos. O último passo de extensão a 72ºC foi de 5 minutos. Já para a amplificação do fragmento de 654-pb contendo o gene avrXv3, a reação da PCR foi composta por 100 ng do DNA molde, 5 mL de tampão de amplificação (Promega Life Science, Madison, WI, EUA), 1,5 mM de MgCl2, 1,25 U de Taq DNA polimerase (Promega), 100 mM de cada deoxinucleosídeo trifosfato (dNTPs) e 1,0 mM dos iniciadores RST88 (5'-CCGCTCGAGATGACAAGTAGTATCA ATC) e RST89 (5'-CCGCTCGAGCTACTTAACGAGATTTGTTAC) (Astua-Monge et al., 2000a), em um volume final de 50 ml. Para esse par de iniciadores, empregou-se um ciclo inicial de desnaturação a 95ºC por 30 segundos, hibridização a 48ºC por 30 segundos e extensão a 72ºC por 1 minuto, seguido de 30 ciclos onde a temperatura de hibridização foi elevada a 56ºC e as demais mantidas. O último passo de extensão foi de 5 minutos a 72ºC. Para ambos os genes, os produtos da PCR foram detectados por eletroforese em gel de 1% de agarose em tampão de corrida TBE 0,5X a 5 V/cm de gel. Os géis foram corados com brometo de etídio (0,5 mg/ml) e fotodocumentados com aparelho Image Master® VDS (Pharmacia Biotech, San Francisco, EUA).
RESULTADOS E DISCUSSÃO
Respostas de hipersensibilidade foram observadas nos genótipos 'Hawaii 7998' e 'NIL 216', bem como nas linhas quase isogênicas de pimentão ECW portadoras dos genes de resistência Bs1, Bs2 e Bs3. A partir de 48 horas foi possível visualizar o colapso dos tecidos (reação de suscetibilidade) nas variedades suscetíveis Walter e Early Calwonder, bem como nas demais variedades desafiadas com isolados compatíveis.
Em relação à localidade, constatou-se a ocorrência das raças T1P8 e T3 apenas no nordeste, ao passo que as raças T2, T2P7 e T2P8 foram detectadas apenas no centro-sudeste do país (Tabela 2). A raça T1P2 ocorreu em ambas regiões. Nos quatro campos em Abaré e em um campo em Petrolina, em 1996, foi possível observar a ocorrência concomitante de duas raças distintas (Tabela 2). Nos demais campos, apenas uma raça foi detectada. Os prováveis genes de avirulência presentes nos isolados analisados foram compilados de acordo com as reações de resposta de cada hospedeira diferencial (Tabela 3).
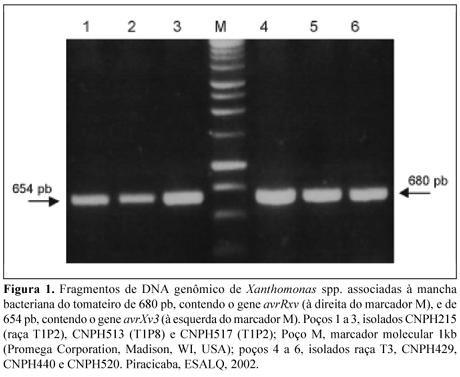
A amplificação do fragmento de 680-pb contendo o gene avrRxv ocorreu nos 38 isolados que provocaram resposta de hipersensibilidade no genótipo 'Hawaii 7998' (Figura 1). DNA de isolados representantes das raças T2, T2P7/8 e T3 foram utilizados com os mesmos iniciadores (RST27 e RST28) mas nenhum produto foi amplificado. Já o fragmento de 654-pb contendo o gene avrXv3 foi amplificado apenas nos 15 isolados que causaram reação de hipersensibilidade em 'NIL 216' (Figura 1).
O presente trabalho relata a ocorrência, pela primeira vez, de algumas raças de Xanthomonas spp. associadas à mancha-bacteriana do tomate para processamento industrial no Brasil. Maior destaque deve ser dado à detecção da raça T3 na Região Nordeste, uma vez que a ocorrência dessa raça havia sido descrita somente no estado da Flórida nos EUA, na Tailândia e no México (Bouzar et al., 1996; Jones et al., 1998). No nordeste, onde a gama de variedades de tomate plantadas é bem mais restrita comparativamente ao centro-sudeste do país, a utilização de variedades contendo o gene de resistência Xv3 poderia resultar em um controle da doença (Astua-Monge et al., 2000a). Alternativamente, o gene Xv4, de L. pennelli poderia ser utilizado, caso fosse constatado que os isolados da raça T3 no Brasil, a exemplo dos isolados da Flórida, também portam o gene avrRv4 (Astua-Monge et al., 2000b). A estabilidade da resistência conferida por esses genes, no entanto, ainda está por ser avaliada. Outro fator interessante em relação a raça T3 é que ela tem se mostrado mais agressiva do que a raça T1, e hoje predomina no estado da Flórida (Jones et al., 1998).
Em relação às espécies/grupos PFGE, isolados de X. axonopodis pv. vesicatoria (grupo PFGE "A" sensu Jones et al., 2000) foram classificados em raças T1P2 e T1P8; de X. vesicatoria ("B") em raça T2; de X. gardneri ( "D") e raças T2P7 e T2P8 e os 15 isolados pertencentes ao grupo PFGE 3, em raça T3. A distribuição das raças de acordo com as espécies apontadas neste estudo concorda com a existência de uma correlação entre raças T e grupos fenotípicos/espécies (Bouzar et al., 1994; Bouzar et al., 1996; Bouzar et al., 1999; Louws et al., 1995). Dessa maneira, isolados T1 apresentam certas características fenotípicas e genotípicas que os classificam como pertencentes à espécie X. axonopodis pv. vesicatoria (= grupo fenotípico "A", sensu Stall et al., 1994; PFGE "A" sensu Jones et al., 2000); isolados T3 têm sido classificados como um subgrupo dentro dessa espécie (Jones et al., 2000); e isolados T2 estavam associados a X. vesicatoria (grupo "B" sensu Stall et al., 1994; PFGE "B" sensu Jones et al., 2000) (Bouzar et al., 1994).
Já para o gênero Capsicum, assim como relatado por Bouzar et al. (1999) e Louws et al. (1995) que utilizaram perfis gerados por rep-PCR para caracterizar isolados de Xanthomonas associadas à mancha-bacteriana, perfis de PFGE não se correlacionaram com as características de patogenicidade. Esse resultado não é surpreendente, uma vez que essas características estão relacionadas com a presença de certos genes de avirulência na bactéria, em geral localizados em plasmídeos, não detectados pela técnica e passíveis de transferência lateral.
Como também analisado por Sahin (1997), a raça T1 parece ter maior habilidade em adaptar-se às plantas de Capsicum uma vez que também é patógeno dessa hospedeira. Essa adaptação talvez possa ter ocorrido pela perda do gene avrBsT que resulta em hipersensibilidade em genótipos de Capsicum (Minsavage et al., 1990). No entanto, os fatores evolutivos envolvidos não são conhecidos. A raça T2, por sua vez, era considerada mais especializada para infectar Lycopersicon (Bouzar et al., 1994; Sahin, 1997); porém, em X. gardneri, tem se mostrado patogênica às duas hospedeiras. Já a raça T3 ainda é aparentemente mais adaptada ao tomateiro.
A presença do gene avrBsT nas raças T2 de X. vesicatoria e T3 pode ser confirmada através de detecção por PCR, já que sua seqüência é conhecida (Ciesiolka et al., 1999), a exemplo do que foi feito neste trabalho para os genes avrRxv e avrXv3. Para isso, no entanto, seria necessário o desenvolvimento de iniciadores específicos. Da mesma forma, seria interessante confirmar a presença do gene avrBs2. Sua presença foi inferida com base nos testes de hipersensibilidade aqui apresentados em todos os isolados que também foram patogênicos a Capsicum. De fato, uma alta freqüência desse gene tem sido observada, inclusive de seus homólogos em Xanthomonas de outras hospedeiras (Kearney e Staskawicz, 1990). Um papel importante na patogenicidade tem sido apontado como a causa dessa ampla distribuição (Kearney e Staskawicz, 1990). Em virtude disso, a possibilidade do uso do gene de resistência Bs2 do pimentão em plantas transgênicas de tomate está sendo analisada (Tai et al., 1999).
Em relação a X. gardneri, por ser aparentemente de ocorrência pouco freqüente, apenas o isolado-tipo XG101 e isolados da Costa Rica haviam sido classificados quanto à raça, sendo agrupados, respectivamente, em T2P1 e P1 (Bouzar et al., 1994; Bouzar et al., 1999). Os isolados de X. gardneri analisados neste estudo, por sua vez, foram classificados em raças distintas (T2P7 ou T2P8). No entanto, convém ressaltar que, com a inclusão do genótipo de C. pubescens PI 235047, raças anteriormente classificadas como P1 e P3 podem passar a ser designadas P7 e P8, respectivamente, de acordo com a reação de resposta desse genótipo (Ritchie et al., 1998; Sahin e Miller, 1998).
O número de isolados analisados por campo em cada município foi baixo para que se viabilizasse uma análise apropriada do grau de diversidade de raças por campo. Porém, vale notar que, dos 31 campos avaliados, em apenas três campos em Abaré e um em Petrolina, foi possível detectar duas raças distintas em relação a Capsicum (T1P2 e T1P8). A conversão da raça P2 em raça P8 (ex-P3) poderia ser possível no próprio campo através da perda do gene avrBs1, localizado em plasmídeo (Minsavage et al., 1990) ou inserção de um elemento genético móvel do tipo IS476 que inativa o gene avrBs1 (Kearney e Staskawicz, 1990). Mudanças de raças P têm sido observadas tanto in vitro, como em campo (Dahlbeck e Stall, 1979; Pohoronezny et al., 1992). Kousik e Ritchie (1996) registraram a conversão de populações de raças P1 (avrBs3) e P2 (avrBs1), em populações P3, em dois campos, respectivamente, cultivados com pimentão resistentes ECW-30R (Bs3) e ECW-10R (Bs1) no mesmo ciclo de cultivo. Para o primeiro caso, confirmou-se a perda do plasmídeo contendo o gene avrBs3 e para o segundo, a mutação do gene avrBs1 pela inserção do elemento IS476. Outra hipótese para a presença das raças P2 e P8, mencionadas anteriormente no presente trabalho, seria a de que as duas raças foram introduzidas simultaneamente ou em eventos distintos via sementes infectadas.
Considerando que, no Brasil, tanto as variedades de tomate para indústria como as de mesa e de pimentão não possuem nenhum gene de resistência caracterizado, sugere-se que a composição racial das populações aqui relatadas não seja um resultado de pressão de seleção exercida pela hospedeira e sim de eventos de introdução ocorridos ao acaso nos campos (efeito fundador) seguidos de adaptação ao ambiente.
O conhecimento da diversidade patogênica das populações bacterianas é de extrema importância para direcionar os programas de melhoramento e utilização de genes de resistência. À medida que o conhecimento sobre os genes de avirulência se acumula, torna-se possível avaliar as populações bacterianas locais e, desse modo, lançar mão daqueles genes de resistência que reconheçam os genes complementares de avirulência predominantes. O emprego da PCR pode auxiliar no processo de designação das raças e monitoramento das populações quanto à presença dos genes de avirulência.
AGRADECIMENTOS
Os autores agradecem a Luiz Cláudio Rodrigues (Olé Alimentos), Rafael Sant'ana (Arisco) e Leonardo Costa da Fonte (Vanden Bergh Alimentos) pelo auxílio nas coletas dos isolados do centro-sudeste, a pesquisadora Mirtes Freitas Lima (Embrapa Semi-Árido) pela coleta das amostras de plantas do nordeste do país. Este trabalho foi em parte financiado pelo CNPq e FAPESP.
LITERATURA CITADA
Recebido para publicação em 23 de dezembro e aceito em 8 de outubro de 2003
- ASTUA-MONGE, G.; MINSAVAGE, G.V.; STALL, R.E.; DAVIS, M.J.; BONAS, U.; JONES, J.B. Resistance of tomato and pepper to T3 strains of Xanthomonas campestris pv. vesicatoria is specified by a plant-inducible avirulence gene. Molecular Plant-Microbe Interactions, v.13, n.9, p.911-921, 2000a.
- ASTUA-MONGE, G.; MINSAVAGE, G.V.; STALL, R.E.; VALLEJOS, C.E.; DAVIS, M.J.; JONES, J.B. Xv4-vrxv4: a new gene-for-gene interaction identified between Xanthomonas campestris pv. vesicatoria race T3 and the wild tomato relative Lycopersicon pennellii. Molecular Plant-Microbe Interactions, v.13, n.12, p.1346-1355, 2000b.
- BALLVORA, A.; PIERRE, M.; ACKERVEKEN, G.; SEBASTIAN, S.; ROSSIER, O.; GANAL, M.; LAHYE, T.; BONAS, U. Genetic mapping and functinal analysis of the tomato Bs4 locus governing recognition of the Xanthomonas campestris pv. vesicatoria AvrBs4 protein. Molecular Plant-Microbe Interactions, v.14, n.14, p.629-638, 2001.
- BONAS, U.; CONRADS-STRAUCH, J.; BALBO, I. Resistance in tomato to Xanthomonas campestris pv. vesicatoria is determined by alleles of the pepper-specific avirulence gene avrBs3 Molecular General Genetics, v.238, p.261-269, 1993.
- BOUZAR, H.; JONES, J.B.; MINSAVAGE, G.V.; STALL, R.E.; SCOTT, J.W. Proteins unique to phenotypically distinct groups of Xanthomonas campestris pv. vesicatoria revealed by silver staining. Phytopathology, v.84, n.1, p.39-44, 1993.
- BOUZAR, H.; JONES, J.B.; SOMODI, G.C.; STALL, R.E.; DAOUZLI, N.; LAMBE, R.C.; FELIX-GASTELUM, R.; TRINIDAD-CORREA, R. Diversity of Xanthomonas campestris pv. vesicatoria in tomato and pepper fields of Mexico. Canadian Journal of Plant Pathology, v.18, n.1, p.75-77, 1996.
- BOUZAR, H.; JONES, J.B.; STALL, R.E.; HODGE, N.C.; MINSAVAGE, G.V.; BENEDICT, A.A.; ALVAREZ, A.M. Physiological, chemical, serological, and pathogenic analysis of a worldwide collection of Xanthomonas campestris pv. vesicatoria. Phytopathology, v.84, n.7, p.663-671, 1994.
- BOUZAR, H.; JONES, J.B.; STALL, R.E.; LOUWS, F.J.; SCHNEIDER, M.; RADEMAKER, J.L.W.; BRUIJN, F.J.; JACKSON, L.E. Multiphasic analysis of xanthomonnads causing bacterial spot disease on tomato and pepper in the Caribbean an Central America: evidence for common lineages within and between countries. Phytopathology, v.89, n.4, p.328-335, 1999.
- CANTEROS, B.; MINSAVAGE, G.; BONAS, U.; PRING, D.; STALL, R.E. A gene from Xanthomonas campestris pv. vesicatoria that determines avirulence in tomato is related to avrBs3 Molecular Plant-Microbe Interactions, v.4, n.6, p.628-632, 1991.
- CIESIOLKA, L.D.; HWIN, T.; GEARLDS, J.D.; MINSAVAGE, G.V.; SAENZ, R.; BRAVO, M.; HANDLEY, V.; CONOVER, S.M.; ZHANG, H.; CAPORGNO, J.; PHENGRASAMY, N.B.; TOMS, A.O.; STALL, R.E.; WHALEN, M.C. Regulation of expression of avirulence gene avrRxv and identification of a family of host interaction factors by sequence analysis of avrBsT Molecular Plant-Microbe Interactions, v.12, n.1, p.35-44, 1999.
- CRUTE, I.A.; PINK, A.C. Genetics and utilization of pathogen resistance in plants. The Plant Cell, v.8, n.10, p.1747-1755, 1996.
- DAHLBECK, D.; STALL, R.E. Mutations for change of race in cultures of Xanthomonas vesicatoria Phytopathology, v.69, n.6, p.634-636, 1979.
- JONES, J.B.; STALL, R.E.; BOUZAR, H. Diversity among xanthomonads pathogenic on pepper and tomato. Annual Review of Phytopathology, v.36, p.41-58, 1998.
- JONES, J.B.; BOUZAR, H.; STALL, R.E.; ALMIRA, E.C.; ROBERTS, P.D.; BOWEN, B.W.; SUDBERRY, J.; STRICKLER, P.M.; CHUN, J. Systematic analysis of xanthomonads (Xanthomonas spp.) associated with pepper and tomato lesions. International Journal of Systematic and Evolutionary Microbiology, v.50, p.1211-1219, 2000.
- JONES, J.B.; SCOTT, J.W. Hypersensitive response in tomato to Xanthomonas campestris pv. vesicatoria . Plant Disease, v.70, n.4, p.337-339, 1986.
- JONES, J.B.; STALL, R.E.; SCOTT, J.W.; SOMODI, G.C.; BOUZAR, H. A third tomato race of Xanthomonas campestris pv. vesicatoria Plant Disease, v.79, n.4, p.395-398, 1995.
- KEARNEY, B.; STASKAWICZ, B.J. Widespread distribution and fitness contribution of Xanthomonas campestris avirulence gene avrBs2 Nature, v.346, n.7, 1990.
- KOUSIK, C.S.; RITCHIE, D.F. Isolation of pepper races 4 and 5 of Xanthomonas campestris pv. vesicatoria from diseased peppers in southeastern U.S. fields. Plant Disease, v.79, n.5, p.540, 1995. (Resumo)
- KOUSIK, C.S.; RITCHIE, D.F. Race shift in Xanthomonas campestris pv. vesicatoria within a season in field-grown pepper. Phytopathology, v.86, n.9, 1996.
- LEACH, J.E.; WHITE, F.F. Bacterial avirulence genes. Annual Review of Phytopathology, v.34, p.153-179, 1996.
- LOPES, C.A.; QUEZADO-SOARES, A.M. Controle químico da mancha-bacteriana em tomateiro para processamento industrial. Summa Phytopathologica, v. 25, n.1, p.21-22, 1999.
- LOUWS, F.J.; FULBRIGHT, D.W.; STEPHENS, C.T.; BRUIJN, F.J. Differentiation of genomic structure by rep-PCR fingerprinting to rapidly classify Xanthomonas campestris pv. vesicatoria Phytopathology, v.85, n.5, p.528-536, 1995.
- MINSAVAGE, G.V.; DAHLBECK, D.; WHALEN, M.C.; KEARNEY, B.; BONAS, U.; STASKAWICZ, B.J.; STALL, R.E. Gene-for-gene relationships specifying disease resistance in Xanthomonas campestris pv. vesicatoria pepper interactions. Molecular Plant-Microbe Interactions, v.3, n.1, p.41-47, 1990.
- POHRONEZNY, K.; STALL, R.E.; CANTEROS, B.I.; KEGLEY, M.; DATNOFF, L.E.; SUBRAMANYA, R. Sudden shift in the prevalent race of Xanthomonas campestris pv. vesicatoria in pepper fields in Southern Florida. Plant Disease, v.76, n.2, 1992.
- QUEZADO-DUVAL, A.M. Diversidade de Xanthomonas spp. associadas à mancha-bacteriana em tomateiro para processamento industrial no Brasil Piracicaba; ESALQ, 2003. 111 p. (Tese doutorado)
- RITCHIE, D.F.; KOUSIK, C.S.; PAXTON, T.C. Response of bacterial spot pathogen strains to four major resistance genes in pepper. In: Villalón, B.; Brandenberger, L. (eds.). PROCEEDINGS OF A NATIONAL PEPPER CONFERENCE. Tampa, Flórida. Citrus and Vegetable Magazine, p.14, 1998.
- ROMERO, A.M.; KOUSIK, C.S.; RITCHIE, D.F. Temperature sensitivity of the hypersensitive response of bell pepper to Xanthomonas axonopodis pv. vesicatoria, Phytopathology, v.92, n.2, 2002.
- SAHIN, F. Detection, identification and characterization of strains of Xanthomonas campestris pv. vesicatoria by traditional and molecular methods, and resistance in Capsicum species to Xanthomonas campestris pv. vesicatoria pepper race 6. Columbus: The Ohio State University, 1997. 182 p. (Tese doutorado)
- SAHIN, F.; MILLER, S.A. First report of pepper race 6 of Xanthomonas campestris pv. vesicatoria, causal agent of bacterial spot of pepper. Plant Disease, v.79, n.11, p.1188, 1995. (Resumo)
- SAHIN, F.; MILLER, S.A. Resistance in Capsicum pubescens to of Xanthomonas campestris pv. vesicatoria pepper race 6. Plant Disease, v.82, n.7, p.794-799, 1998.
- SCOTT, J.W.; MILLER, S.A.; STALL, R.E.; JONES, J.B.; SOMODI, G.C.; BARBOSA, V.; FRANCIS, D.L.; SAHIN, F. Resistance to race T2 of the bacterial spot pathogen in tomato. HortScience, v.32, n.4, p.724-727, 1997.
- STALL, R.E.; BEAULIEU, C.; EGEL, D.; HODGE, N.C.; LEITE, R.P.; MINSAVAGE, G.V.; BOUZAR, H.; JONES, J.B.; ALVAREZ, A.M.; BENEDICT, A.A. Two genetically diverse groups of strains are included in Xanthomonas campestris pv. vesicatoria International Journal of Systematic Bacteriology, v.44, n.1, p.47-53, 1994.
- STALL, R.E. Breeding for resistance to bacterial diseases of plants. In : Zambolim, L.; Vale, F.X.R. (ed.) Resistência de plantas a doenças CONGRESSO BRASILEIRO DE FITOPATOLOGIA (30: 1997: Poços de Caldas, MG). Brasília: Sociedade Brasileira de Fitopatologia, p.51-56, 1997.
- TAI, T.H.; DAHLBECK, D.; CLARK, E.T.; GAJIWALA, P.; PASION, R.; WHALEN, M.C.; STALL, R.E.; STASKAWICZ, B.J. Expression of resistance the Bs2 pepper gene confers resistance to bacterial spot disease in tomato. Proceedings of the National Academy of Sciences USA, v.96, n.24, p.14153-14158, 1999.
- VAUTERIN, L.; HOSTE, B.; KERSTERS, K.; SWINGS, J. Reclassification of Xanthomonas International Journal of Systematic Bacteriology, v.45, p.472-489, 1995.
- WANG, J.F.; JONES, J.B.; SCOTT, J.W.; STALL, R.E. A new race of the tomato group of strains of Xanthomonas campestris pv. vesicatoria Phytopathology, v.80, n.10, p.1070, 1990. (Resumo)
- WANG, J.F.; JONES, J.B.; SCOTT, J.W.; STALL, R.E. Several genes in Lycopersicon esculentum control hypersensitivity to Xanthomonas campestris pv. vesicatoria Phytopathology, v.84, n.7, p.702-706, 1994.
- WHALEN, M.; STALL, R.E.; STASKAWICZ, B.J. Characterization of a gene from a tomato pathogen determining hypersensitive resistance in non-host species and genetic analysis of this resistance in bean. Proceedings of the National Academy of Sciences USA, v.85, p.6743-6747, 1988.
- WHALEN, M.C.; WANG, J.F. CARLAND, F.M.; HEISKELL, M.E.; DAHLBECK, D.; MINSAVAGE, G.V.; JONES, J.B.; SCOTT, J.W.; STALL, R.E.; STASKAWICZ, B.J. Avirulence gene avrRxv from Xanthomonas campestris pv. vesicatoria specifies resistance on tomato line Hawaii 7998. Molecular Plant-Microbe Interactions, v.6, n.5, p.616-627, 1993.
- WILSON, K. Preparation of genomic bacteria. In: AUSUBEL, F.M.; BRENT, R.; KINGSTON, R.E., MOORE, D.D.; SEIDMAN, J.G.; SMITH, J.A.; STRUHL, K. Short protocols in molecular biology 4 ed. New York: John Wiley & Sons. 1999. p.2-122-14.
- YU, Z.H.; WANG, F.J.; STALL, R.E.; VALLEJOS, C.E. Genomic localization of tomato genes that control a hypersensitive reaction to Xanthomonas campestris pv. vesicatoria (Doidge) Dye. Genetics, v.141, p.675-682, 1995.
Datas de Publicação
-
Publicação nesta coleção
26 Maio 2004 -
Data do Fascículo
Mar 2004
Histórico
-
Recebido
23 Dez 2003 -
Aceito
08 Out 2003