Abstracts
The importance of the study of acetic bacteria, on species of the Gluconobacter genus is based on its industrial application, as these possess the capacity of bioconversion of sorbitol to sorbose, enabling the process of vitamin C production. The study involved samples collected in industries of soft drinks, flowers, fruits and honey, followed by purification, phenotypic identification, molecular identification with the use of primer defined from Nucleotide Sequence Database consultation. Strains preserved were identified as members of the Acetobacteraceae family, Gluconobacter genus. 110 strains had been isolated of substrate: Pyrostegia venusta (ker-gawler), honey, Vitis vinifera (grape), Pyrus communis (pear), Malus sp. (apple) and in two samples of soft drinks. Of this total 57 strains had been recovered in manitol medium (manitol, yeast extract, peptone), 12 in YMG medium (glucose, manitol, yeast extract, ethanol, acetic acid), 41 in enrichment medium (De Ley and Swings) and later in the GYC medium (glucose, yeast extract and calcium carbonate). 68 strains were identified as Gram negative bacilli rods. Of these, 31 were characterized biochemically as belonging to the Acetobacteriaceae family as they were catalase positive, oxidase negative and producers of acid from glucose. The characterization of these strains was complemented with the biochemistry tests: gelatin liquefaction, nitrate reduction, indole and H2S production, oxidation of ethanol to acetic acid and molecular tests for genus identification. Only eight strains were characterized as pertaining to the Gluconobacter genus. The strains are maintained in collection cultures at the Microbiology Laboratory of the Biology Department at the São Paulo State University (UNESP) in Assis, stored in malt extract at -196 ºC.
Gluconobacter; Isolation; Identification; Molecular Markers; PCR
A importância do estudo de bactérias acéticas, em especial as do gênero Gluconobacter, está baseada em suas aplicações industriais, pois estas possuem a capacidade de bioconversão de sorbitol a sorbose, viabilizando o processo de produção de vitamina C. O estudo envolveu coletas de amostras em indústrias de refrigerante, flores, frutos e mel, seguidas de purificação, identificação fenotípica e identificação molecular, com a utilização de iniciador definido a partir de consulta ao Nucleotide Sequence Database. Preservaram-se as linhagens identificadas como membros da família Acetobacteriaceae, gênero Gluconobacter. Foi isolado um total de 110 linhagens dos substratos: Pyrostegia venusta (Cipó de São João), mel, Vitis vinifera (uva), Pyrus communis (pêra), Malus sp. (maçã) e de duas amostras de refrigerantes envasados em embalagens de PET de 2 L. Deste total, 57 linhagens foram recuperadas em meio MYP (manitol, extrato de levedura, peptona), 12 em meio YGM (glicose, manitol, extrato de levedura, etanol, ácido acético), 41 em meio de enriquecimento e, posteriormente, em meio GYC (glicose, extrato de levedura e carbonato de cálcio). Obtiveram-se 68 linhagens identificadas como bastonetes Gram negativos. Destas, 31 foram caracterizadas bioquimicamente como pertencentes à família Acetobacteriaceae por serem catalase positivas, oxidase negativas e produtoras de ácido a partir de glicose. A caracterização dessas linhagens foi complementada com os testes bioquímicos: liquefação da gelatina, redução de nitrato, formação de indol e H2S e oxidação de etanol a ácido acético. Métodos moleculares foram aplicados para identificação do gênero Gluconobacter. Finalmente, oito linhagens foram caracterizadas como pertencentes ao gênero Gluconobacter. As linhagens encontram-se depositadas em coleção de cultura do laboratório de Microbiologia do Departamento de Biologia da UNESP, campus de Assis, estocadas em extrato de malte 20 a -196 ºC.
Gluconobacter; isolamento; identificação; marcadores moleculares; PCR
ORIGINAL
Estudo de bactérias do gênero Gluconobacter: isolamento, purificação, identificação fenotípica e molecular
Study of bacteria Gluconobacter sp.: isolation, purification, phenotypic and molecular identification
André Luiz Dorini de OliveiraI,II,III,1 1 A quem a correspondência deve ser enviada ; Vitório Santos JuniorI,II,III; Rhavena Graziela LiottiI; Estevão ZilioliI; Wilma Aparecida SpinosaI; João Tadeu Ribeiro-Paes1
1 Departamento de Ciências Biológicas, Universidade Estadual Paulista - UNESP, Assis, SP, Brasil
2Departamento de Saúde, ADR - Biomavale, Rua Dr. Geraldo Nogueira Leite, 1735, CEP 19800-000, Assis, SP, Brasil, e-mail: olivaldo3@yahoo.com.br
3 Instituto de Saúde, Universidade Paulista - UNIP, Assis, SP, Brasil
RESUMO
A importância do estudo de bactérias acéticas, em especial as do gênero Gluconobacter, está baseada em suas aplicações industriais, pois estas possuem a capacidade de bioconversão de sorbitol a sorbose, viabilizando o processo de produção de vitamina C. O estudo envolveu coletas de amostras em indústrias de refrigerante, flores, frutos e mel, seguidas de purificação, identificação fenotípica e identificação molecular, com a utilização de iniciador definido a partir de consulta ao Nucleotide Sequence Database. Preservaram-se as linhagens identificadas como membros da família Acetobacteriaceae, gênero Gluconobacter. Foi isolado um total de 110 linhagens dos substratos: Pyrostegia venusta (Cipó de São João), mel, Vitis vinifera (uva), Pyrus communis (pêra), Malus sp. (maçã) e de duas amostras de refrigerantes envasados em embalagens de PET de 2 L. Deste total, 57 linhagens foram recuperadas em meio MYP (manitol, extrato de levedura, peptona), 12 em meio YGM (glicose, manitol, extrato de levedura, etanol, ácido acético), 41 em meio de enriquecimento e, posteriormente, em meio GYC (glicose, extrato de levedura e carbonato de cálcio). Obtiveram-se 68 linhagens identificadas como bastonetes Gram negativos. Destas, 31 foram caracterizadas bioquimicamente como pertencentes à família Acetobacteriaceae por serem catalase positivas, oxidase negativas e produtoras de ácido a partir de glicose. A caracterização dessas linhagens foi complementada com os testes bioquímicos: liquefação da gelatina, redução de nitrato, formação de indol e H2S e oxidação de etanol a ácido acético. Métodos moleculares foram aplicados para identificação do gênero Gluconobacter. Finalmente, oito linhagens foram caracterizadas como pertencentes ao gênero Gluconobacter. As linhagens encontram-se depositadas em coleção de cultura do laboratório de Microbiologia do Departamento de Biologia da UNESP, campus de Assis, estocadas em extrato de malte 20 a -196 ºC.
Palavras-chave: Gluconobacter; isolamento; identificação; marcadores moleculares; PCR.
ABSTRACT
The importance of the study of acetic bacteria, on species of the Gluconobacter genus is based on its industrial application, as these possess the capacity of bioconversion of sorbitol to sorbose, enabling the process of vitamin C production. The study involved samples collected in industries of soft drinks, flowers, fruits and honey, followed by purification, phenotypic identification, molecular identification with the use of primer defined from Nucleotide Sequence Database consultation. Strains preserved were identified as members of the Acetobacteraceae family, Gluconobacter genus. 110 strains had been isolated of substrate: Pyrostegia venusta (ker-gawler), honey, Vitis vinifera (grape), Pyrus communis (pear), Malus sp. (apple) and in two samples of soft drinks. Of this total 57 strains had been recovered in manitol medium (manitol, yeast extract, peptone), 12 in YMG medium (glucose, manitol, yeast extract, ethanol, acetic acid), 41 in enrichment medium (De Ley and Swings) and later in the GYC medium (glucose, yeast extract and calcium carbonate). 68 strains were identified as Gram negative bacilli rods. Of these, 31 were characterized biochemically as belonging to the Acetobacteriaceae family as they were catalase positive, oxidase negative and producers of acid from glucose. The characterization of these strains was complemented with the biochemistry tests: gelatin liquefaction, nitrate reduction, indole and H2S production, oxidation of ethanol to acetic acid and molecular tests for genus identification. Only eight strains were characterized as pertaining to the Gluconobacter genus. The strains are maintained in collection cultures at the Microbiology Laboratory of the Biology Department at the São Paulo State University (UNESP) in Assis, stored in malt extract at -196 ºC.
Keywords: Gluconobacter; Isolation; Identification; Molecular Markers; PCR.
1 Introdução
O gênero Gluconobacter pertence à família Acetobacteriaceae. É constituído por bactérias que possuem forma de bastonetes, Gram-negativos e não formam endósporos. São aeróbicas obrigatórias, tendo metabolismo estritamente respiratório com oxigênio como o aceptor final de elétrons. As colônias são opacas, a temperatura ótima de crescimento está entre 25 e 30 ºC, e o pH ótimo é de 5,5. Quanto às reações bioquímicas, são: catalase positivas, negativas para oxidase, redução de nitrato, liquefação da gelatina, produção de indol e formação de H2S. São quimiorganotróficas e oxidam etanol a ácido acético. Não oxidam acetato ou lactato a CO2 e H2O. Linhagens de Gluconobacter têm sido isoladas de flores, terra de jardim, levedura de panificação, mel de abelha, frutas, sidra, cerveja, vinho, vinagre de vinho, cerveja Bantu da África do Sul, seiva de palma e bebidas suaves. Causam doença rosa em abacaxis e apodrecem maçãs e pêras (De LEY; GOSSELÉ; SWINGS, 1984).
Na lista aprovada dos nomes das bactérias, uma única espécie, Gluconobacter oxidans, foi listada no gênero Gluconobacter (SKERMAN et al., 1980 apud YAMADA; YUKPHAN, 2008; GOSSELÉ et al. , 1983). Uma segunda espécie, Gluconobacter cerinus, foi proposta pelo baixo conteúdo do grupo C + G no DNA em contraste com o alto conteúdo do grupo C + G no DNA de Gluconobacter oxidans. A terceira e quarta espécies: Gluconobacter frateurii (MASON; CLAUS, 1989) e Gluconobcter asaii (MASON; CLAUS, 1989) foram descritas. Entretanto, G. asaii foi descrita como sendo a mesma espécie de G. cerinus por Katsura et al. (2002), Tanaka et al. (1999) e Yamada et al. (1999).
Além destas, mais duas espécies também foram descritas, Gluconobacter albidus (YUKPHAN et al. , 2005) e Gluconobacter thailandicus (TANASUPAWAT et al., 2006). O número de espécies descritas nesse gênero ficou em cinco no total, número muito baixo quando comparado ao gênero Acetobacter.
As bactérias acéticas constituem um grupo com importantes aplicações industriais, particularmente as do gênero Gluconobacter, que podem ser usadas para conversão oxidativa de sorbitol a sorbose, um intermediário importante na produção (química) do ácido L-ascórbico (vitamina C). A capacidade de suboxidação destes microrganismos vem sendo explorada na síntese de vitamina C, o que torna a sua produção industrial economicamente viável (SPINOSA, 2002).
Devido à alta demanda e ao crescimento do mercado consumidor, inúmeras empresas têm investido em melhorias na tecnologia de produção da vitamina C. A fermentação do sorbitol tem sido amplamente utilizada nas indústrias e estudos vêm sendo realizados para a otimização da produção. A empresa escocesa Hoffmann La Roche, sediada em Dalry, por exemplo, investiu entre os anos de 1998 e 2001 um capital de $200 milhões para melhorar a tecnologia de produção da vitamina C (CHEMIKAL, 2004).
Apesar do grande investimento mundial realizado pelo setor industrial, os resultados são pouco divulgados. No Brasil, os estudos nesta linha são escassos e direcionados à aplicação de bactérias acéticas nas indústrias produtoras de vinagre. Santos (2000)realizou um trabalho de isolamento, purificação, identificação rápida e armazenamento de bactérias acéticas isoladas em fermentadores de vinagre, entre as quais algumas cepas foram identificadas como pertencentes ao gênero Gluconobacter.
As bactérias acéticas são frequentemente citadas como agentes deteriorantes nas indústrias de bebidas. O tipo de deterioração que elas produzem inclui, especialmente, sabores desagradáveis, aumento na viscosidade e formação de gás. Com o aumento da produção de bebidas em embalagens plásticas, as bactérias acéticas não fermentativas tornaram-se importantes objetos de estudo. Várias são as razões que contribuem para esse fato, tais como a resistência de Gluconobacter a sanitizantes comumente empregados na indústria engarrafadora de bebidas, a sua habilidade de crescer na presença de ácido sórbico, ácido benzoico e nos altos níveis de oxigênio livre que caracterizam as bebidas em recipientes plásticos (CIRIGLIANO, 1982).
O presente trabalho teve como objetivo instituir um banco de microrganismos que apresentassem as características: bastonetes, Gram negativos, oxidase negativos, catalase positivos, capazes de acidificar glicose, que não liquefizessem gelatina, não formassem indol a partir do triptofano e que pudessem ser identificados como pertencentes à família Acetobacteriaceae, gênero Gluconobacter complementados com análise de caracterização molecular pela metodologia de RAPD-PCR.
2 Material e métodos
2.1 Microrganismos padrão
Para comparação com as linhagens isoladas, foram utilizadas culturas padrão de Gluconobacter e Acetobacter. Foram utilizadas linhagens de Gluconobacteroxydans (CCT0554) da Fundação Tropical André Tosello em Campinas-SP e outra de Gluconobacteroxydans (ATCC 621H) proveniente da coleção de culturas do Instituto de Pesquisas Tecnológicas - IPT; linhagens de Acetobacter aceti (CCT0190) obtidas também da coleção de culturas da Fundação Tropical de Pesquisas e Tecnologia André Tosello, sediada em Campinas-SP. e outra linhagem de Acetobacter sp. (086/06), produtora de vinagre, de coleção de culturas particular (SPINOSA, 2002) e depositada na coleção de culturas do laboratório de Microbiologia do Departamento de Biologia da UNESP, campus de Assis.
2.2 Meios de cultura
Os meios de cultura utilizados neste estudo foram: Meio 1, meio de enriquecimento proposto por De Ley, Gosselé, Swings, 1984; Meio 2, Yeast Glucose Manitol (YGM) citado por Ohmori et al., 1980; Meio 3, Manitol Yeast Peptone (MYP); Meio 4, Glucose Yeast CaCO3 (GYC), estes dois últimos citados por De Ley; Gosselé; Swings, 1984; Meio 5, Dextrose Sorbitol Manitol (DSM) proposto por CIRIGLIANO, 1982; Meio 6, meio de diferenciação de Carr(CARR, 1968). Os meios de cultura sólidos utilizados na amostragem foram preparados segundo a técnica da camada dupla, com 0,5% de ágar na camada inferior e 1% de ágar na camada superior (SOKOLLEK; HERTEL; HAMMES, 1998).
2.3 Amostragem
As amostras foram coletadas de indústrias produtoras de refrigerante, de flores, de frutos e de mel de abelha. No próprio local de coleta, foram inoculadas em meios líquidos (1 a 3), previamente preparados, em duplicatas, para enriquecimento. Em seguida, as amostras foram para incubação em incubadora rotatória a 120 rpm por 96 horas a 30 ºC. Após o crescimento no meio líquido (meio 2 e 3), 0,1 mL da cultura foi distribuído em placas de Petri com alça de Drigalski para o mesmo meio sólido, e foram incubadas em estufa bacteriológica a 30 ºC por 96 horas. Para o meio 1, dentre os tubos que mostraram acidificação, amostras foram estriadas em placas de Petri com meio GYC (meio 4). Colônias que dissolveram o CaCO3 foram purificadas e caracterizadas De Vero, Giudici (2008).
2.4 Isolamento e purificação
Fez-se a triagem dos microrganismos que se desenvolveram em cada tipo de meio, observando-se as características morfológicas das colônias crescidas. Executou-se a técnica de esgotamento e incubação à temperatura de 30 ºC por 96 horas como mínimo. Para a perfeita purificação, este procedimento foi feito por, no mínimo, quatro vezes. A pureza da cultura foi verificada em microscópio ótico, após a coloração de Gram.
2.5 Triagem e identificação fenotípica das linhagens
A triagem foi realizada com base no teste de coloração de Gram (RIBEIRO, SOARES, 1993). Posteriormente, as cepas que apresentaram forma de bastonetes Gram-negativos e produziram ácido a partir de glicose foram submetidas a uma rápida identificação fenotípica, para diferenciação da família Acetobacteriaceae. Os testes utilizados para esta identificação foram: oxidase, catalase, produção de indol, redução de nitrato, liquefação de gelatina, oxidação de etanol a ácido acético e formação de H2S (De LEY; GOSSELÉ; SWINGS, 1984).
As linhagens identificadas como membros da Família Acetobacteriaceae foram submetidas à identificação do gênero, conforme proposto por CARR, 1968 em meio com verde bromocresol (meio 6) e Cirigliano, 1982em meio DSM com púrpura de bromocresol (meio 5). Em ambos os métodos, as culturas foram estriadas nos respectivos meios e o gênero foi identificado pela capacidade de produção de ácido acético evidenciada pela mudança da cor do meio de cultura por meio dos corantes de pH utilizados na formulação dos meios de cultura.
2.6 Caracterização Molecular
A extração do DNA genômico das linhagens selecionadas foi realizada por meio do uso de tampão de lise contendo CTAB (2% p/v de brometo de acetiltrimetilamônio - CTAB - e 1.4 M NaCl; 0,2% v/v de β-mercapto etanol; 20 mM EDTA; 100 mM Tris HCl pH 8), e 3 etapas utilizando: fenol, fenol:clorofórmio (1:1) e clorofórmio: álcool isoamílico (24:1). Em cada etapa, o material foi centrifugado a 12000 g, por 12 minutos e recolhido o sobrenadante. Após a última centrifugação o sobrenadante foi incubado com 7,5 µL de RNAse (350 µg/mL), durante 1 hora, a 37 ºC. Em seguida, foi adicionado isopropanol à amostra (na proporção de 2:3 v/v) para precipitação do DNA. O DNA, após ser mantido overnight, foi isolado por centrifugação a 12000 g, durante 10 minutos, a 4 ºC, tratado com 500 µL de etanol 70%, contendo 10 mM de acetato de amônio e centrifugado a 12000 g, durante 10 minutos, a 4 ºC. O DNA foi seco ao ar e dissolvido em tampão TE (10 mM Tris e 1 mM EDTA, pH 8.0), variando-se o volume do tampão conforme o rendimento da extração (SPADA, SILVA, ABDEL-HAMID, 2002).
A análise da pureza do DNA e sua quantificação foram realizadas por meio de leitura da absorvância em espectrofotômetro (Ultrospec III - Pharmacia, UK), nos comprimentos de onda 260 nm e 280 nm.
As amostras das linhagens selecionadas foram amplificadas por PCR. Cada uma das reações foi realizada num volume final de 20 µL, contendo 0,5 unidade de Taq DNA polimerase (GIBCO - BRL - MD, USA), 1 X tampão de PCR , 7 pmol do iniciador (Carr, 1968) (5'-ATGTACCACTGTACCACG-3' - GenBank, acesso no. AB003953) e 0,2 mM de cada dNTP. O conteúdo foi submetido à denaturação inicial em Termociclador (PTC 200 Peltier Thermal Cycler, MJ Research - USA) a 95 ºC, durante 5 minutos. Foram realizados dois ciclos nas seguintes condições: 95 ºC por 30 segundos para a denaturação; 30 ºC, 2 minutos para o anelamento e 72 ºC por 1 minuto para o alongamento, seguidos de 31 ciclos, durante os quais, a temperatura de anelamento foi alterada para 40 ºC e o tempo de alongamento aumentado para 5 minutos no ciclo final (SPADA et al., 2002). Os produtos de amplificação foram visualizados em eletroforese em gel de poliacrilamida 8%, corado com solução de nitrato de prata saturada, conforme descrito por Sanguinetti, Dias-Neto e Simpson (1994).
2.7 Análise dos dados
As bandas polimórficas foram analisadas visualmente e ordenadas em uma matriz binária de dados, caracterizada pela presença de banda (1) e ausência (0) dela. A matriz de dados binários (retangular) foi analisada pelo programa computacional TREECON for Windows versão 1.3b (VAN DE PEER; WACHTER; TREECON, 1994). A similaridade e a diversidade genética das linhagens foram calculadas por meio do coeficiente de similaridade Simple-Matching (SM). As matrizes de similaridade obtidas a partir do coeficiente SM foram usadas para agrupar os dados, usando o método de agrupamento de pares não ponderados com médias aritméticas (UPGMA)(SNEATH; SOKAL, 1973), incluídas no programa computacional TREECON for Windows versão 1.3b.
A análise da variabilidade genética foi realizada mediante o cálculo do coeficiente de variação, dado pela fórmula: CV = 100. s/SX, onde s é o desvio padrão e SX é a média de similaridade obtida entre as espécies. A partir da matriz de similaridade de SimpleMatching foram obtidas as similaridades médias (SMM) interespecificamente. Com base na matriz de similaridade SMM foram calculadas as variâncias e os coeficientes de variação (SPADA et al., 2002).
3 Resultados e discussão
O isolamento e a identificação de bactérias do gênero Gluconobacter, que apresentam características apropriadas para as condições de trabalhos biotecnológicos, são fundamentais para o melhoramento e desenvolvimento produtivo no setor industrial.
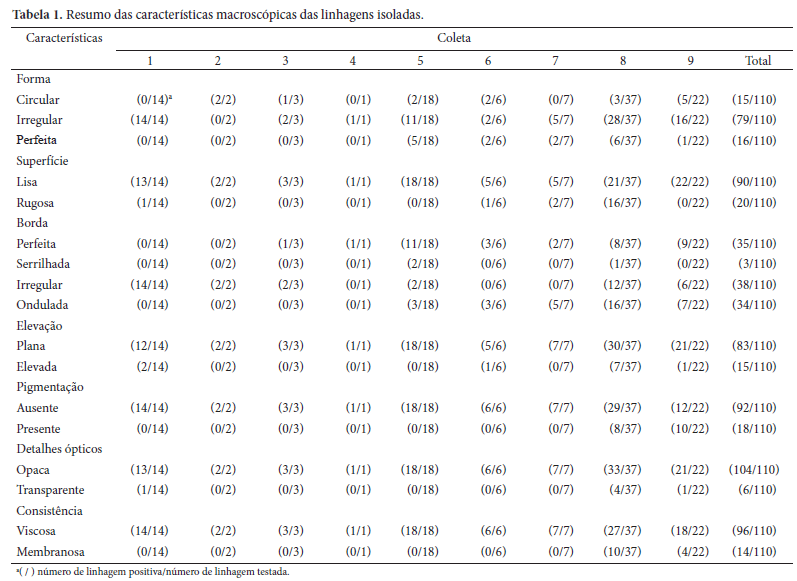
No presente estudo, utilizaram-se as metodologias de isolamento, identificação bioquímica e molecular para caracterizar microrganismos coletados em diversos nichos, característicos do gênero. A Tabela 1 apresenta o resumo das características macroscópicas das 110 linhagens isoladas no decorrer deste levantamento. Estas linhagens foram isoladas dos substratos: Cipó de São João (32), mel (8), uva (9), pêra (6), maça (7); e de duas amostras de refrigerantes (48), nichos estes estabelecidos previamente por De Ley, Gosselé e Swings (1984). Deste total, 57 linhagens foram recuperadas em meio MYP (manitol, extrato de levedura, peptona), 12 em meio YGM (glicose, manitol, extrato de levedura, etanol, ácido acético), 41 em meio de enriquecimento, posteriormente, para o meio GYC (glicose, extrato de levedura e carbonato de cálcio). Dentre as 110 linhagens recuperadas, 68 foram identificadas como bastonetes Gram-negativos. Destas, 32 foram caracterizadas bioquimicamente como catalase positivas, oxidase negativas e produtoras de ácido a partir de glicose.
A caracterização dessas linhagens foi complementada com testes bioquímicos de liquefação da gelatina, redução de nitrato, formação de indol e H2S, bem como o de oxidação de etanol a ácido acético, selecionando para estudo, nessa etapa, 31 linhagens (Tabela 2). As linhagens apresentaram variações bioquímicas quando comparadas com os dados encontrados na literatura e com as linhagens tipo.
O teste de diferenciação de gênero (Acetobacter versusGluconobacter) foi aplicado conforme proposto por Carr (1968)e por Cirigliano (1982). A leitura desses resultados não possibilitou a diferenciação ao nível de gênero, por mais criteriosa que fosse sua aplicação, ambos se mostrando inconsistentes para essa finalidade.
A caracterização das amostras selecionadas foi efetuada mediante PCR. Inicialmente, avaliou-se a variabilidade genética das 31 linhagens isoladas e caracterizadas bioquimicamente. Deste total foram selecionadas 15 linhagens para caracterização molecular, com base nos dados preliminares obtidos no gel (dados não mostrados). Para uma maior confiabilidade na leitura dos dados, foram escolhidos padrões eletroforéticos reprodutíveis e invariáveis a partir de DNAs de uma mesma linhagem obtidos em diferentes extrações, mantendo o mesmo iniciador, com intensidade de fragmentos de média a forte e sem contaminação dos produtos de amplificação. Podem-se observar na Figura 1 os perfis eletroforéticos de amplificação das linhagens em estudo.
O coeficiente de similaridade SM foi utilizado para análise dos dados, por ser o coeficiente geralmente mais usado para comparação de similaridade entre espécies relacionadas (DUNN; EVERITT, 1982). Skroch, Tivang e Nienhuis (1992), afirmaram que o coeficiente de similaridade SM é o mais apropriado para estimar relações de similaridade entre seres vivos, de acordo com a hipótese de haver uma alta probabilidade de que, entre indivíduos intimamente relacionados, tanto a presença como a ausência em comum de um produto amplificado (banda no gel), entre pares de genótipos, resultem das mesmas mudanças genéticas.
A análise do fenograma obtido pelo coeficiente de similaridade SM (Figura 2), mostra que as linhagens incluídas neste estudo formaram 2 grupos de maior similaridade. O primeiro grupo incluiu 10 linhagens, sendo 8 isoladas e 2 Gluconobacter (tipo). O segundo conjunto foi composto por oito linhagens, a saber, 6 isoladas, 1 Acetobacter (tipo) e 1 linhagem produtora de vinagre de coleção de cultura de Spinosa (2002). Esses dois grupos se relacionaram com nível de similaridade de, aproximadamente, 0,50 (50%). A linhagem identificada com o número 194 se relacionou ao conjunto formado pelos dois grupos anteriores com nível de similaridade menor que 0,50 (50%), formando um ramo isolado. No fenograma, pode-se constatar que os dois grupos maiores são formados por linhagens isoladas que apresentaram níveis de maior similaridade genética com as linhagens tipo, tanto as do gênero Gluconobacter como as do gênero Acetobacter.
O maior valor de similaridade foi observado entre as linhagens CCT0554 e 173, que apresentaram nível de relacionamento de 0,95 ou 95%. O menor valor, 0,45 (45%) foi aquele observado entre a linhagem 194 e o grande grupo formado pelas demais representantes das linhagens. Essa linhagem apresentou um comportamento de outgroup em relação ao grande grupo formado pelas demais estudadas (Figura 2). Esses resultados indicam que a linhagem 194 possa ter uma origem genética distinta.
Quanto à variabilidade detectada entre as 10 linhagens pertencentes ao primeiro grupo, pode-se verificar que a maior similaridade entre um par de linhagens foi de 0,95 ou 95% e a menor foi de 0,65 ou 65%. De um modo geral, a similaridade variou de 0,94 a 0,65, tendo sido formados pequenos grupos, com similaridades variáveis.
A partir do iniciador selecionado, foram detectados 45 fragmentos, variando o número de fragmentos obtidos por linhagem. Entretanto, apenas 21 fragmentos polimórficos foram utilizados para a construção da matriz binária de presença e ausência, necessária para a obtenção dos coeficientes de similaridade. Os fragmentos restantes, em número de 24, não foram considerados devido à falta de reprodutibilidade ou por não permitirem análises precisas da presença das bandas. Pode-se sugerir que a técnica molecular empregada correspondeu ao objetivo específico desejado.
De um modo geral, os resultados obtidos indicaram que a PCR permitiu caracterizar 8 linhagens selecionadas, a saber, 173, 144, 209, 186, 146, 135, 171 e 117. Estas linhagens apresentaram níveis de similaridade genética mais intimamente relacionados aos indivíduos tipo, pertencentes à família Acetobacteriaceae, gênero Gluconobacter (Figura 2). De acordo com os dados obtidos, pelo menos nestas linhagens, pode-se observar que o coeficiente de variação foi igual a 0,3653 ou 36,53% (Tabela 3).
Esses índices sugerem a existência de uma variabilidade genética considerável neste conjunto de linhagens selecionadas. O emprego de análises moleculares mais específicas, tais como: Análise de Microssatélite, Desenvolvimento de marcadores espécie-específicos, Análise de DNA ribossômico por PCR-DGGE (Polymerase chain reaction-denaturing gradient gel electrophoresis) citados por De Vero e Guidici (2007), especialmente com a linhagem 173, podem ser úteis para programas de melhoramento e desenvolvimento biotecnológicos produtivos no setor industrial.
4 Conclusões
O presente trabalho mostrou ser possível isolar e preservar linhagens pertencentes à família Acetobacteriaceae gênero Gluconobacter utilizando os meios de cultura: YGM, MYP e meio de enriquecimento DE VERO, GIUDICI (2008) e, posteriormente, meio GYC. O meio MYP apresenta melhor performance para isolamento. O meio YGM é específico, possibilitou o menor número de linhagens. Uma das linhagens triada em todos os testes e selecionada para a identificação molecular, isolada no meio YGM, apresentou maior grau de similaridade (95%) com o microrganismo tipo (CCT0554). As técnicas propostas por Carr (1968)e Cirigliano(1982)mostraram pouca reprodutibilidade, por mais criterioso que seja a condução dos testes. Provas bioquímicas mostraram-se insuficientes para caracterização dos microrganismos estudados, apresentaram variações em relação aos dados encontrados na literatura e entre linhagens consideradas tipo. A análise molecular empregada mostrou-se eficiente para a caracterização de linhagens isoladas, agrupando-as com as linhagens tipo. É possível preservar linhagens de Gluconobacter a -196 ºC com crioprotetor extrato de malte 20%, conforme descrito por Spinosa (2002).
Referências bibliográficas
CARR, J. G. Methods for identification acetic acid bacteria. In: GIBBS, B. M.; SHAPTON, D. A. (Eds.). Identification methods for microbiologists. London: Academic Press, 1968. (v. 2)
CHEMICAL MARKET REPORTER. Ascorbic acid. Surrey, 1998. Disponível em: <http://www.findarticles.com/cf_dls/m0FVP/1998_Oct_12/53089150/p1/article.jhtml>. Acesso em: 10 jan. 2004.
CIRIGLIANO, M. C. A selective medium for the isolation and differentiation of Gluconobacter and Acetobacter. Journal of Food Science, v. 47, n.3, p. 1038-1039, 1982.
DE LEY, J.; GOSSELÉ, F.; SWINGS, J. Genus I Acetobacter. In: KRIEG, N. R.; HOLT, J. G. Bergey's manual of systematic bacteriology. Maryland: Williams & Wilkens, 1984. p. 268-274. (v. 1)
DE VERO, L.; GIUDICI, P. Genus-specific profile of acetic acid bactéria by 16S rDNA PCR-DGGE. International Journal of Food Microbiology,v. 125, n.1, p. 96-101, 2008.
DUNN, G.; EVERITT, B. S. An introduction to mathematical taxonomy. Cambridge: Cambridge University Press, 1982. 152 p.
KATSURA, K. et al. Gluconobacter asaii (Mason and Claus, 1989) is a Junior subjective synonym of Gluconobacter cerinus (Yamada and Akita 1984). International Journal of Systematic and Evolutionary Microbiology, v. 52, p. 1635-1640, 2002.
MASON, L. M.; CLAUS, G. W. Phenotypic characteristics correlated with deoxyribonucleic acid sequence similarities for threee species of Gluconobacter: G. oxidans, G. frateurii e G. asaii. International Journal of Systematic Bacteriology, v. 39, n.3, p. 174-184, 1989.
OHMORI, S. et al. Isolation and Identification of acetic acid bacteria submerged acetic fermentation at high temperature. Agricultural and Biological Chemistry, v. 44, n. 12, p. 2901-2906, 1980.
RIBEIRO, M. C.; SOARES, M. M. S. R. Microbiologia prática: roteiro e manual. Bactérias e fungos. Rio de Janeiro: Atheneu, 1993. 112 p.
SANGUINETTI, C. J.; DIAS NETO, E.; SIMPSON, A. J. G. Rapid silver staining and recovery of PCR products separated on polyacrylamide gels. Biotechniques, v. 17, n.5, p. 915-919, 1994.
SANTOS, V. J. Estudo sobre bactérias acéticas: isolamento, purificação, identificação e armazenamento. Assis: Universidade Estadual Paulista, 2000. (Iniciação científica)
SKROCH, P.; TIVANG, J.; NIENHUIS, J. Analysis of genetic relationships using RAPD marker data. In: SYMPOSIUM ON APPLICATION OF RAPD TECHNOLOGY TO PLANT BREEDING, 1992, Minneapolis. Proceedings... Madison: Crop Science Society of America, 1992. p. 26-30.
SNEATH, P. H. A.; SOKAL, R. R. Principles of numerical taxonomy. San Francisco: W. H. Freeman, 1973. p. 573.
SOKOLLEK, S. J.; HERTEL, C.; HAMMES, W. P. Cultivation and preservation of vinegar bacteria, Journal of Biotechnology, v. 60, n.3, p. 195-206, 1998.
SPADA, R. G. M. et al. Genetic markers between Biomphalaria glabrata snails susceptible and resistant to Schistosoma mansoni infection. Memóras do Instituto Oswaldo Cruz, v. 97, suppl. 1, p. 53-58, 2002.
SPINOSA, W. A. Isolamento, seleção, identificação e parâmetros cinéticos de bactérias acéticas provenientes de indústrias de vinagre. Campinas, 2002. 93 p. Tese (Doutorado em Ciências de Alimento) - Universidade Estadual de Campinas - UNICAMP.
SWINGS, J. The genera Acetobacter and Gluconobacter. In: BALOWS, A. et al. (ed.). The Prokaryotes. New York: Springer-Verlag, 1992. p. 2268-2286.
TANAKA, M. et al. Reclassification of the strains with low G+C contents of DNA belonging to the genus Gluconobacter Asai 1935 (Acetobacteriaceae). Bioscience, Biotechnology and Biochemistry, v. 63, n.6, p. 989-992, 1999.
TANASUPAWAT, S. et al. Gluconobacter thailandicus sp. nov. an acetic acid bacterium in the a-Proteobacteria. Journal of General and Applied Microbiology, v. 50, n.3, p. 159-167, 2006.
Van de PEER Y.; WACHTER, R. TREECON for Windows: a software package for the construction and drawing of evolutionary trees for the Microsoft Windows environment. Computer Applications in the Biosciences, v. 10, n5, p. 569-570, 1994.
YAMADA, Y. et al. Identification of acid acetic bactéria isolated from indonesian sources, especially of isolates classified in the genus Gluconobacter. Journal of General and Apllied Microbiology, v. 45, n.1, p. 23-28, 1999.
YAMADA, Y.; YUKPHAN, P. Genera and species in acetic acid bacteria. International Journal of Food Microbiology, v. 125, n.1, p. 96-101, 2008.
YUKPHAN, P. et al. Neoasia chiangmaiensis gen. sp. nov. a novel osmotolerant acetic acid bacterium in the a-Proteobacteria. Journal of General and Applied Microbiology, v. 51, n.5, p. 301-311, 2005.
Recebido para publicação em 12/2/2008
Aceito para publicação em 16/5/2009 (003227)
- CARR, J. G. Methods for identification acetic acid bacteria. In: GIBBS, B. M.; SHAPTON, D. A. (Eds.). Identification methods for microbiologists London: Academic Press, 1968. (v. 2)
- CHEMICAL MARKET REPORTER. Ascorbic acid Surrey, 1998. Disponível em: <http://www.findarticles.com/cf_dls/m0FVP/1998_Oct_12/53089150/p1/article.jhtml>. Acesso em: 10 jan. 2004.
- CIRIGLIANO, M. C. A selective medium for the isolation and differentiation of Gluconobacter and Acetobacter. Journal of Food Science, v. 47, n.3, p. 1038-1039, 1982.
- DE LEY, J.; GOSSELÉ, F.; SWINGS, J. Genus I Acetobacter. In: KRIEG, N. R.; HOLT, J. G. Bergey's manual of systematic bacteriology Maryland: Williams & Wilkens, 1984. p. 268-274. (v. 1)
- DE VERO, L.; GIUDICI, P. Genus-specific profile of acetic acid bactéria by 16S rDNA PCR-DGGE. International Journal of Food Microbiology,v. 125, n.1, p. 96-101, 2008.
- DUNN, G.; EVERITT, B. S. An introduction to mathematical taxonomy Cambridge: Cambridge University Press, 1982. 152 p.
- KATSURA, K. et al. Gluconobacter asaii (Mason and Claus, 1989) is a Junior subjective synonym of Gluconobacter cerinus (Yamada and Akita 1984). International Journal of Systematic and Evolutionary Microbiology, v. 52, p. 1635-1640, 2002.
- MASON, L. M.; CLAUS, G. W. Phenotypic characteristics correlated with deoxyribonucleic acid sequence similarities for threee species of Gluconobacter: G. oxidans, G. frateurii e G. asaii. International Journal of Systematic Bacteriology, v. 39, n.3, p. 174-184, 1989.
- OHMORI, S. et al. Isolation and Identification of acetic acid bacteria submerged acetic fermentation at high temperature. Agricultural and Biological Chemistry, v. 44, n. 12, p. 2901-2906, 1980.
- RIBEIRO, M. C.; SOARES, M. M. S. R. Microbiologia prática: roteiro e manual. Bactérias e fungos. Rio de Janeiro: Atheneu, 1993. 112 p.
- SANGUINETTI, C. J.; DIAS NETO, E.; SIMPSON, A. J. G. Rapid silver staining and recovery of PCR products separated on polyacrylamide gels. Biotechniques, v. 17, n.5, p. 915-919, 1994.
- SANTOS, V. J. Estudo sobre bactérias acéticas: isolamento, purificação, identificação e armazenamento. Assis: Universidade Estadual Paulista, 2000. (Iniciação científica)
- SKROCH, P.; TIVANG, J.; NIENHUIS, J. Analysis of genetic relationships using RAPD marker data. In: SYMPOSIUM ON APPLICATION OF RAPD TECHNOLOGY TO PLANT BREEDING, 1992, Minneapolis. Proceedings.. Madison: Crop Science Society of America, 1992. p. 26-30.
- SNEATH, P. H. A.; SOKAL, R. R. Principles of numerical taxonomy San Francisco: W. H. Freeman, 1973. p. 573.
- SOKOLLEK, S. J.; HERTEL, C.; HAMMES, W. P. Cultivation and preservation of vinegar bacteria, Journal of Biotechnology, v. 60, n.3, p. 195-206, 1998.
- SPINOSA, W. A. Isolamento, seleção, identificação e parâmetros cinéticos de bactérias acéticas provenientes de indústrias de vinagre Campinas, 2002. 93 p. Tese (Doutorado em Ciências de Alimento) - Universidade Estadual de Campinas - UNICAMP.
- SWINGS, J. The genera Acetobacter and Gluconobacter In: BALOWS, A. et al. (ed.). The Prokaryotes New York: Springer-Verlag, 1992. p. 2268-2286.
- TANAKA, M. et al. Reclassification of the strains with low G+C contents of DNA belonging to the genus Gluconobacter Asai 1935 (Acetobacteriaceae) Bioscience, Biotechnology and Biochemistry, v. 63, n.6, p. 989-992, 1999.
- TANASUPAWAT, S. et al. Gluconobacter thailandicus sp. nov. an acetic acid bacterium in the a-Proteobacteria. Journal of General and Applied Microbiology, v. 50, n.3, p. 159-167, 2006.
- Van de PEER Y.; WACHTER, R. TREECON for Windows: a software package for the construction and drawing of evolutionary trees for the Microsoft Windows environment. Computer Applications in the Biosciences, v. 10, n5, p. 569-570, 1994.
- YAMADA, Y. et al. Identification of acid acetic bactéria isolated from indonesian sources, especially of isolates classified in the genus Gluconobacter Journal of General and Apllied Microbiology, v. 45, n.1, p. 23-28, 1999.
- YAMADA, Y.; YUKPHAN, P. Genera and species in acetic acid bacteria. International Journal of Food Microbiology, v. 125, n.1, p. 96-101, 2008.
- YUKPHAN, P. et al. Neoasia chiangmaiensis gen. sp. nov. a novel osmotolerant acetic acid bacterium in the a-Proteobacteria. Journal of General and Applied Microbiology, v. 51, n.5, p. 301-311, 2005.
Publication Dates
-
Publication in this collection
26 Apr 2010 -
Date of issue
Mar 2010
History
-
Received
12 Feb 2008 -
Accepted
16 May 2009