RESUMO
Objetivo
Desenvolver um algoritmo computacional aplicado a imagens de ressonância magnética, para segmentação automática de tumores cerebrais.
Métodos
Foram utilizadas 130 imagens de ressonância magnética nas sequências T1c, T2 e FSPRG T1c e nos planos axial, sagital e coronal de pacientes acometidos com câncer cerebral. Os algoritmos empregaram técnicas de correção de contraste, normalização de histograma e binarização, para desconectar estruturas adjacentes do cérebro e realçar a região de interesse. A segmentação automática foi realizada por meio da detecção por coordenadas e por média aritmética da área. Operadores morfológicos foram utilizados para eliminar elementos indesejáveis e reconstruir a forma e a textura do tumor. Os resultados foram comparados com as segmentações manuais de dois médicos radiologistas, para determinar a eficácia dos algoritmos implementados.
Resultados
Os acertos foram de 89,23% na correspondência entre a segmentação obtida e o padrão-ouro.
Conclusão
É possível localizar e delimitar a região tumoral de forma automática, sem necessidade de interação com o usuário baseado em dois métodos inovadores de detecção dos extremos do cérebro e de exclusão dos tecidos não tumorais em imagens de ressonância magnética.
Diagnóstico por imagem; Neoplasias encefálicas; Processamento de imagem assistida por computador; Imagem por ressonância magnética; Simulação por computador
ABSTRACT
Objective
To develop a computational algorithm applied to magnetic resonance imaging for automatic segmentation of brain tumors.
Methods
A total of 130 magnetic resonance images were used in the T1c, T2 and FSPRG T1C sequences and in the axial, sagittal and coronal planes of patients with brain cancer. The algorithms employed contrast correction, histogram normalization and binarization techniques to disconnect adjacent structures from the brain and enhance the region of interest. Automatic segmentation was performed through detection by coordinates and arithmetic mean of the area. Morphological operators were used to eliminate undesirable elements and reconstruct the shape and texture of the tumor. The results were compared with manual segmentations by two radiologists to determine the efficacy of the algorithms implemented.
Results
The correlated correspondence between the segmentation obtained and the gold standard was 89.23%.
Conclusion
It is possible to locate and define the tumor region automatically with no the need for user interaction, based on two innovative methods to detect brain extreme sites and exclude non-tumor tissues on magnetic resonance images.
Diagnostic imaging; Brain neoplasms; Image processing, computer-assisted; Magnetic resonance imaging; Computer simulation
INTRODUÇÃO
Uma das técnicas mais avançadas e difundidas para visualização de tumores cerebrais é a imagem de ressonância magnética (RM),( 11. Jui SL, Zhang S, Xiong W, Yu F, Fu M, Wang D, et al. Brain MRI Tumor Segmentation with 3D Intracranial Structure Deformation Features. IEEE Intelligent Systems. 2016;31(2):66-76. )devido à sua capacidade de distinção entre diversos tipos de tecidos, e por ser um método não invasivo e de alta resolução anatômica, o que torna a obtenção do diagnóstico menos traumática para o paciente. Assim, a RM tem sido ferramenta importante para a detecção, o monitoramento e o diagnóstico precoces do tumor.( 22. Virupakshappa, Amarapur B. Cognition-based MRI brain tumor segmentation technique using modified level set method. Cognition, Technol Work. 2019; 21(3):357-69.
3. Szabó Z, Kapás Z, Lefkovits L, Gyõrfi Á, Szilágyi SM, Szilágyi L. Automatic segmentation of low-grade brain tumor using a random forest classifier and Gabor features.In:14th International Conference on Natural Computation, Fuzzy Systems and Knowledge Discovery (ICNC-FSKD) [Internet]. Huangshan (CHN): ICNC-FSKD 2018 [cited 2019 Feb 6]. Available from: http://real.mtak.hu/86054/1/2023.pdf
http://real.mtak.hu/86054/1/2023.pdf...
- 44. Zhou M, Scott J, Chaudhury B, Hall L, Goldgof D, Yeom KW, et al. Radiomics in Brain Tumor: image assessment, quantitative feature descriptors, and machine-learning approaches. AJNR Am J Neuroradiol. 2018;39(2):208-16. Review. )
Embora as RM apresentem alto grau de detalhamento, o diagnóstico feito somente com a inteligência humana é cansativo e demanda tempo, podendo ser suscetível às variações interpessoais, à perda de informação e à fadiga visual. Além disso, durante a aquisição, a imagem pode apresentar baixa qualidade, contraste ruim e presença de artefatos, que são problemas difíceis de lidar por meio do olho humano.( 55. Goetz M, Weber C, Bloecher J, Stieltjes B, Meinzer HP, Maier-Hein KH. Extremely randomized trees based brain tumor segmentation [Internet]. Proceeding of MICCAI 2014 Brain Tumor Segmentation Challenge. 2014;6-11 [cited 2019 Feb 19]. Available from: http://people.csail.mit.edu/menze/papers/proceedings_miccai_brats_2014.pdf
http://people.csail.mit.edu/menze/papers...
)Ademais, o processo de segmentar as bordas da massa tumoral é normalmente realizado visualmente com a segmentação manual do tumor, que pode detectar apenas mudanças grandes,( 66. Schmidt M. Automatic brain tumor segmentation [Tese]. Edmonton: University of Alberta; 2005. 161f. )obter grande variação na interpretação por diferentes radiologistas e demandar tempo para realização de segmentação detalhada.( 77. Clarke LP, Velthuizen RP, Clark M, Gaviria J, Hall L, Goldgof D, et al. MRI measurement of brain tumor response: comparison of visual metric and automatic segmentation. Magn Reson Imaging. 1998;16(3):271-9. , 88. Işın A, Direkoğlu C, Şah M. Review of MRI-based brain tumor image segmentation using deep learning methods. Procedia Computer Science. 2016;102:317-24. )
Diante disso, têm sido desenvolvidos sistemas computacionais de processamento de imagens e inteligência artificial, denominados diagnóstico assistido por computador (CAD - computer-aided diagnosis ), para detectar e/ou avaliar anormalidades em imagens médicas, e auxiliar os médicos na precisão do diagnóstico e em neurocirurgias.( 99. Jorritsma W, Cnossen F, van Ooijen PM. Improving the radiologist–CAD interaction: designing for appropriate trust. Clin Radiol. 2015;70(2):115-22. Review. )A análise e o processamento da imagem são usados para desenvolver a competência analítica dos médicos( 1010. Zhang J, Cui X, Li J, Wang R. Imbalanced classification of mental workload using a cost-sensitive majority weighted minority oversampling strategy. Cognition, Technol Work. 2017;19(4):633-53. )e reduzir o tempo necessário para a avaliação exata, o planejamento da terapia e o rastreamento da evolução do tumor.( 33. Szabó Z, Kapás Z, Lefkovits L, Gyõrfi Á, Szilágyi SM, Szilágyi L. Automatic segmentation of low-grade brain tumor using a random forest classifier and Gabor features.In:14th International Conference on Natural Computation, Fuzzy Systems and Knowledge Discovery (ICNC-FSKD) [Internet]. Huangshan (CHN): ICNC-FSKD 2018 [cited 2019 Feb 6]. Available from: http://real.mtak.hu/86054/1/2023.pdf
http://real.mtak.hu/86054/1/2023.pdf...
, 1111. Doupi P, Svaar H, Bjørn B, Deilkås E, Nylén U, Rutberg H. Use of the global trigger tool in patient safety improvement efforts: nordic experiences. Cognition, Technol Work. 2015;17(1):45-54. )Essas técnicas fornecem uma segunda opinião aos radiologistas, na compreensão de imagens médicas, melhorando a precisão do diagnóstico.( 1212. Mohan G, Subashini MM. MRI based medical image analysis: survey on brain tumor grade classification. Biomedical Signal Processing Control. 2018;39:139-61. )
Embora os sistemas CAD tenham alcançado relativo progresso em seu desempenho, ainda há vários problemas que precisam ser solucionados para melhorar sua sensibilidade. Além disso, a análise automática de imagens médicas apresenta problemas relacionados à imagem, que precisam ser superados, como ruídos que podem alterar a intensidade do pixel e a não uniformidade de intensidade da imagem.( 1313. Clarke LP, Velthuizen RP, Camacho MA, Heine JJ, Vaidyanathan M, Hall LO, et al. MRI segmentation: methods and applications. Magn Reson Imaging. 1995;13(3):343-68. Review. )Assim, a investigação completa de algoritmos alternativos para todas as etapas de um sistema CAD torna-se relevante, no que concerne desde a etapa de pré-processamento até a fase final de classificação, utilizando métodos que possam minimizar o processamento de RM.
Neste trabalho, foi desenvolvido um novo algoritmo de segmentação automática de tumores cerebrais em RM nas sequências T1c, T2 e FSPGR T1c, empregando-se métodos matemáticos, operações lógicas e morfológicas. Ao final da análise, o algoritmo foi capaz de segmentar a região tumoral em concordância com a segmentação manual dos radiologistas (o padrão-ouro).
OBJETIVO
Desenvolver e validar um conjunto de ferramentas computacionais para segmentação automática de tumores cerebrais em imagens de ressonância magnética.
MÉTODOS
A metodologia foi estruturada com um sistema composto por três etapas: pré-processamento, segmentação e pós-processamento. Esse sistema funciona com três tipos diferentes de RM: T1c, T2 e FSPRG T1c, nos cortes axial, coronal e sagital. Assim, todas as etapas do sistema foram projetadas para funcionar com essas três modalidades de RM e para qualquer um dos cortes. O sistema e as etapas são descritos na tabela 1 .
A pesquisa foi iniciada após a aprovação do Comitê de Ética em Pesquisa da Universidade Federal do Vale do São Francisco (UNIVASF), CAAE: 90399118.5.0000.5196 e parecer 2.954.791.
Local de realização da pesquisa
A pesquisa foi desenvolvida na cidade de Juazeiro (BA), no Laboratório de Pesquisa em Engenharia Elétrica, no campus da UNIVASF.
Base de dados
Para o presente estudo, foram analisados 14 casos reais de tumores cerebrais de RM, cuja presença de tumores foi confirmada por meio da segmentação manual feita por médicos especialistas, o padrão-ouro, demarcada nas imagens e descritas no prontuário de cada paciente anonimamente.
Foi construída uma base de dados com 116 imagens cedidas por uma clínica médica de radiologia da cidade de Petrolina por anonimidade (base privada). A máquina utilizada para realização dos exames foi a Intera de 1,5 T da Philips Medical Systems, os slices (fatias) possuíam 3,5mm de espessura, e o espaçamento entre cada corte era de 3,85mm. Além disso, utilizaram-se 14 imagens obtidas da base de dados da REpository of Molecular BRAin Neoplasia DaTa (base pública) fornecida gratuitamente pelo The Cancer Imaging Archive (TCIA). Assim, totalizam-se 130 imagens (fatias) de 14 pacientes de ambos os sexos e idade variada, sendo 19 T1c, 48 T2 e 63 FSPGR T1c.
As imagens encontravam-se no formato de arquivos Digital Imaging and Communications in Medicine (DICOM®), apresentando diferentes resoluções para cada tipo de sequência, a saber: 256×256 (T2), 512×512 (FSPGR T1c) e 704×704 (T1c). Os algoritmos foram estudados e implementados no sistema MATrix LABoratory ®(MATLAB®), que consiste em uma plataforma que utiliza linguagem de programação de alto nível e ambiente de desenvolvimento de algoritmos, análise e visualização de dados e computação numérica.
Critérios de inclusão e exclusão
Foram selecionadas imagens pré-diagnosticadas com tumores cerebrais em hipersinal nas ponderações T1c, T2 e FSPGR T1c e em diferentes cortes (axial, sagital e coronal) em pacientes de ambos os sexos e de idade variada. A mesma quantidade de imagens com ausência de tumor foi incluída. Imagens sem diagnósticos prévios e imagens com alta densidade de ruídos (extremamente borradas) que impediam qualquer visualização das estruturas do cérebro foram excluídas dos casos.
Pré-processamento
O pré-processamento de imagens consiste na aplicação de técnicas para redução de artefatos e realce de imagens, que visam melhorar sua qualidade e destacar uma região de interesse (ROI), fornecendo sua visualização mais detalhada para melhorar a precisão de segmentação.( 1414. Gonzalez RC, Woods RE. Processamento de imagens digitais, tradução do original digital image processing. São Paulo: Edgard Blücher; 2000. p. 509. , 1515. Isa IS, Sulaiman SN, Mustapha M, Karim NK. Automatic contrast enhancement of brain MR images using Average Intensity Replacement based on Adaptive Histogram Equalization (AIR-AHE). Biocybernetics Biomedical Engineering. 2017;37(1):24-34. )Esta etapa baseou-se no aprimoramento do contraste da imagem com o objetivo de evidenciar a região do tumor. Todas as imagens apresentam valores de intensidade de pixel em níveis de cinza e estas intensidades dos pixels foram normalizadas entre zero e 1.
Equalização de histograma
Diferente de outras técnicas de imagem, na RM, a intensidade do pixel não tem valor fixado em relação à imagem do tecido, ou seja, o mesmo tecido pode ter intensidades diferentes, o que torna mais difícil a adoção de características de intensidade como fontes de informação na segmentação das imagens.( 1616. Alegro MC. Segmentação computadorizada de tumores do encéfalo em imagens de ressonância magnética [Dissertação]. São Paulo: Universidade de São Paulo; 2009. )Essa falta de padronização de intensidade é reduzida por meio da aplicação de um procedimento de normalização. A equalização do histograma é aplicada para deixar áreas desejadas mais claras do que o resto da imagem e, assim, facilitar sua extração.( 22. Virupakshappa, Amarapur B. Cognition-based MRI brain tumor segmentation technique using modified level set method. Cognition, Technol Work. 2019; 21(3):357-69. , 1717. Isselmou AE, Zhang S, Xu G. A novel approach for brain tumor detection using MRI Images. J Biomedical Sci Eng. 2016;9(10):44-52.
18. Ulku EE, Camurcu AY. Computer aided brain tumor detection with histogram equalization and morphological image processing techniques. In: International Conference on Electronics, Computer and Computation (ICECCO) [Internet]. Ancara:(TR); 2013 [cited 2019 Feb 6]. Available from: https://ieeexplore.ieee.org/document/6718225?tp=&arnumber=6718225
https://ieeexplore.ieee.org/document/671...
- 1919. Tharani SS, Sumathi B. A Novel Algorithm for Enhancing an Image of Brain Tumor. Int J Scientific Res Comput Sci Eng Inf Technol. 2018;3(3):186-90. )Utiliza-se a função histeq
( 2020. Thompson CM, Shure L. Image processing toolbox [for use with Matlab] (No. BOOK). The Math Works. 1995. )para obter tal resultado.
Ajuste de intensidade
Para realizar o ajuste de intensidade, utilizaram-se as funções imhmax e imhmin ,( 2020. Thompson CM, Shure L. Image processing toolbox [for use with Matlab] (No. BOOK). The Math Works. 1995. )que suprimem todos os máximos com valor de cinza inferior a um limiar h ou todos os mínimos maiores que h.( 2121. Kawaguchi K, Kageyama R, Sano M. Topological defects control collective dynamics in neural progenitor cell cultures. Nature. 2017;545 (7654):327-31. , 2222. Malhotra H, Naaz S. Analysis of MRI images using data mining for detection of brain tumor. Int J Advanced Res Comput Sci. 2018;9(2):15-8. )Esses limiares foram prefixados de acordo com a ocorrência de níveis de cinza das imagens (histograma), sendo definidos empiricamente, a partir da observação, dois intervalos ótimos para o conjunto das imagens estudadas. Cada imagem de RM apresenta quantidade variável com intensidades mais próximas de zero ou de 1, sendo algumas predominantemente escuras e outras mais claras. Assim, de acordo a área de fragmentos do histograma das imagens, foi possível determinar tais limiares para o ajuste de intensidade.
Binarização
Para separar o fundo da imagem e as ROI, aplicou-se a binarização, com auxílio da função imregionalmax ,( 2020. Thompson CM, Shure L. Image processing toolbox [for use with Matlab] (No. BOOK). The Math Works. 1995. )que identifica os pixels de maior intensidade e retorna uma imagem binária cujos pixels máximos adquirem valor 1, enquanto que, aos demais, são atribuídos valor zero.( 1515. Isa IS, Sulaiman SN, Mustapha M, Karim NK. Automatic contrast enhancement of brain MR images using Average Intensity Replacement based on Adaptive Histogram Equalization (AIR-AHE). Biocybernetics Biomedical Engineering. 2017;37(1):24-34. )Após esse processo, a imagem compõe-se de um número menor de subregiões, desconectadas em grande parte, implicando em menor quantidade de dados e menor tempo para o processamento nas próximas etapas.
Segmentação
A etapa de segmentação do cérebro é responsável por separar o encéfalo das áreas adjacentes, como as meninges, os ossos do crânio e os tecidos saudáveis.( 1616. Alegro MC. Segmentação computadorizada de tumores do encéfalo em imagens de ressonância magnética [Dissertação]. São Paulo: Universidade de São Paulo; 2009. )Uma das inovações deste trabalho é o algoritmo para realizar a segmentação automática de tumores cerebrais, que consiste em dois métodos: o de detecção por coordenada e o de detecção por área.
Método de detecção por coordenada
O primeiro diferencial deste trabalho é o método de detecção por coordenada (MDC), que elimina, de forma totalmente automática, as regiões indesejáveis externas ao cérebro, como o osso do crânio, as meninges e a gordura subcutânea, utilizando apenas conceitos matemáticos básicos e operações lógicas. O algoritmo marca automaticamente quatro pontos ao redor do cérebro denominados de extremos superior, inferior, esquerdo e direito e elimina todas as regiões que se localizam antes do extremo esquerdo-superior e após o extremo direito-inferior. Para isso, considerou-se que a cabeça do paciente ocupa a região central da imagem, visto que as RM se apresentam dessa maneira, e observou-se que o encéfalo é delimitado por pixels com intensidade superior a zero.
Considerando-se uma imagem com dimensões X e Y, as coordenadas do extremo esquerdo-direito são determinadas fixando-se a ordenada na metade da imagem (Y/2). Para os extremos superior e inferior, é fixada a abscissa (X/2). O algoritmo varre a linha que corresponde a Y/2 da esquerda para a direita e a coluna correspondente a X/2 de cima para baixo, até encontrar o primeiro pixel maior que zero, criando dois vetores com todos os pixels diferentes de zero. O primeiro elemento de cada vetor corresponde ao pixel do extremo esquerdo-superior do encéfalo, e o último elemento equivale aos limites opostos, isto é, extremo direito-inferior.
No entanto, na maioria das imagens, o osso do crânio possui pixels de intensidades superiores a zero. Assim, os extremos são marcados nessa região. Como o interesse é delimitar o cérebro, e este encontra-se a uma pequena distância do crânio, que é praticamente invariável do ponto de vista anatômico, cada extremo é deslocado de um valor correspondente a tal distância, para serem fixados nos limites do cérebro.
Além disso, o algoritmo também leva em consideração cada tipo de corte, visto que o cérebro apresenta formas diferentes para cada situação. Nos cortes coronal e sagital, as imagens apresentam também a região do pescoço, a qual contém pixels maiores que zero. Para evitar erros na marcação do extremo inferior, por exemplo, estimou-se a distância entre a parte mais inferior do pescoço e do cérebro, deslocando-se a ordenada do extremo inferior para cima, em uma proporção maior.
Após delimitado o cérebro por seus pontos extremos, o algoritmo procura subregiões ( labels ) que possuam coordenadas menores que o extremo esquerdo- superior, e maiores que o extremo direito-inferior. Os labels são delimitados por um retângulo utilizando o parâmetro bounding box da função regionprops. ( 2020. Thompson CM, Shure L. Image processing toolbox [for use with Matlab] (No. BOOK). The Math Works. 1995. )O retângulo intercepta cada label com pelo menos dois de seus vértices. Assim, se pelo menos um vértice da bounding box que contém o label estiver externo aos limites dos extremos do cérebro, isto indica que a região é externa ao cérebro e, portanto, atribui-se valor zero a todos os pixels de tal label . Com isso, eliminam-se grande parte do crânio, gordura e meninges. A figura 1 apresenta, de forma ilustrativa, o procedimento realizado para eliminação das regiões externas ao cérebro, utilizando o MDC.
Segmentação por meio do método de detecção por coordenada. À esquerda, é feita a demarcação dos pontos extremos do cérebro. No segundo quadro, é ilustrada a localização das subregiões externas e internas do cérebro. Por último, eliminam-se todas as subregiões externas ao cérebro
Método de detecção por área
Após a aplicação da detecção por coordenadas, o algoritmo mantém subregiões na área interna ao cérebro, que não necessariamente correspondem ao tumor que se deseja segmentar. No entanto, a área do tumor cerebral resultante da detecção por coordenadas é, na maioria das vezes, maior que a média aritmética das áreas de todos os elementos espúrios que venham a permanecer. Assim, o segundo diferencial deste algoritmo é a utilização de uma propriedade bidimensional, ou seja, a área, como forma de eliminar tecidos não tumorais dentro da região do cérebro.
Antes de executar o método de detecção por área (MDA), aplicou-se a função imerode,( 2020. Thompson CM, Shure L. Image processing toolbox [for use with Matlab] (No. BOOK). The Math Works. 1995. )que realiza a operação morfológica de erosão com a finalidade de desconectar os elementos espúrios e reduzir suas áreas com a formação de novas subregiões com áreas menores. O MDA calcula a área de cada label resultante e sua média aritmética, atribuindo valor zero aos pixels dos elementos que possuam área menor que a média aritmética das áreas de todos os labels . Desta forma, elimina-se grande parte do tecido cerebral ou em sua totalidade, restando o tumor.
A figura 2 esquematiza o MDA, em que o maior quadrado em azul representa o tumor, os demais quadrados azuis representam tecidos não tumorais, e o quadrado verde representa o label que corresponde à média aritmética das áreas. Utilizando operações lógicas (comparações), o algoritmo verifica as regiões que possuem área menor que a área média, e as elimina, atribuindo valor lógico zero aos seus pixels . Desta forma, idealmente, elimina-se o número máximo de elementos indesejáveis, mantendo-se apenas o tumor.
Método da eliminação de subregiões por meio do método de detecção por área. Na primeira coluna, é feita a detecção das subregiões internas ao cérebro. Na segunda, calcula-se a média aritmética das áreas das subregiões detectadas. Na terceira, realizam-se a comparação entre o tamanho de cada subregião e a média aritmética. Na última, eliminam-se todas as subregiões com área inferior ao da média
Pós-processamento
Utilizaram-se operadores morfológicos de dilatação ( imdilate )( 2020. Thompson CM, Shure L. Image processing toolbox [for use with Matlab] (No. BOOK). The Math Works. 1995. )para recapturar os tecidos que foram perdidos no processo de erosão. A fim de reter a maior região do tumor, a máscara binária é obtida preenchendo buracos na imagem dilatada ( imfill ).( 2020. Thompson CM, Shure L. Image processing toolbox [for use with Matlab] (No. BOOK). The Math Works. 1995. )Finalmente, a máscara binária é aplicada à imagem original, por meio de convolução espacial, para se obterem o tamanho próximo ao real dos elementos segmentados e a posterior extração dos atributos de textura.( 1515. Isa IS, Sulaiman SN, Mustapha M, Karim NK. Automatic contrast enhancement of brain MR images using Average Intensity Replacement based on Adaptive Histogram Equalization (AIR-AHE). Biocybernetics Biomedical Engineering. 2017;37(1):24-34. , 2323. Alegro Mde C, Amaro Junior E, Lopes Rde D. Computerized brain tumor segmentation in magnetic resonance imaging. einstein (São Paulo). 2012; 10(2):158-63. )
Avaliação dos resultados
Os resultados quantitativos da segmentação automática foram calculados utilizando comparação entre o padrão-ouro e os resultados do método proposto de segmentação. As métricas comumente utilizadas na literatura( 2424. Deng W, Luo L, Lin X, Fang T, Liu D, Dan G, et al. Head and neck cancer tumor segmentation using support vector machine in dynamic contrast-enhanced MRI. Contrast Media Mol Imaging. 2017;2017:8612519.
25. Vaibhavi P, Rupal K. Brain Tumor Segmentation Using K-means-FCM Hybrid Technique. In: Perez GM, Tiwari S, Trivedi MC, Mishra KK. Ambient Communications and Computer Systems. New York: Springer; 2018. p 341-52. - 2626. Ma Z, Wu X, Song Q, Luo Y, Wang Y, Zhou J. Automated nasopharyngeal carcinoma segmentation in magnetic resonance images by combination of convolutional neural networks and graph cut. Experimental Therapeutic Medicine. 2016;16(3):2511-21. )para análise de desempenho são a percentagem de acerto (PA), a qual é calculada em termos do número de verdadeiro-positivos (VP) e a quantidade determinada pelo padrão-ouro; e a taxa de correspondência (TC), em função do número de falso-positivos (FP). Nos trabalhos citados, a PA e a TC são definidas, respectivamente, como:
Os valores da PA mais altos indicam a existência de um número maior de pixels verdadeiro-positivos na área segmentada. O valor de TC avalia quão distante está a segmentação do padrão-ouro. O valor ideal de TC corresponde a 1, o que indica combinação perfeita entre o padrão-ouro e a segmentação. Os valores de TC próximos a 1 indicam que a segmentação possui uma percentagem bem maior de verdadeiro-positivos com relação aos falso-positivos na segmentação.( 1616. Alegro MC. Segmentação computadorizada de tumores do encéfalo em imagens de ressonância magnética [Dissertação]. São Paulo: Universidade de São Paulo; 2009. )
RESULTADOS
A etapa de pré-processamento foi implementada seguindo a descrição apresentada. Ao final dela, foram obtidas as imagens tratadas com as técnicas de pré-processamento. A figura 3 mostra a saída do processo final após serem aplicados a equalização de histograma, o ajuste de intensidade para correção de contraste e a técnica de binarização. As imagens à direita são imagens binarizadas (só apresentam pixels com intensidade zero ou 1). São apresentados os três tipos de sequência de RM e os três tipos de cortes de fatias do encéfalo de diferentes pacientes.
A primeira, terceira e sexta colunas referem-se à imagem antes do processamento, enquanto que as demais colunas apresentam as imagens após o pré-processamento. A primeira linha mostra os resultados de imagens do tipo T1 com contraste, a segunda linha mostra os respectivos resultados para a sequência T2 e a terceira linha para a sequência T1, com contraste na sequência gradiente eco com uso de spoiller.
A etapa de segmentação teve início com a delimitação do cérebro por pontos extremos e extração das regiões externas a eles, seguida da aplicação de uma função morfológica de dilatação, e finalizando com a aplicação do algoritmo MDA. Devido à técnica de erosão, que acarreta perda de pixels de determinadas regiões, e ao processo de binarização, que converte a imagem em escala de cinza em apenas dois níveis de intensidade, foram utilizadas operações morfológicas de dilatação, preenchimento de buracos vazios e reconstrução, para que as regiões segmentadas fossem reconstruídas e retomadas à textura original. O procedimento morfológico foi usado nessas imagens binárias para refinar a margem e o conteúdo das imagens do tumor.
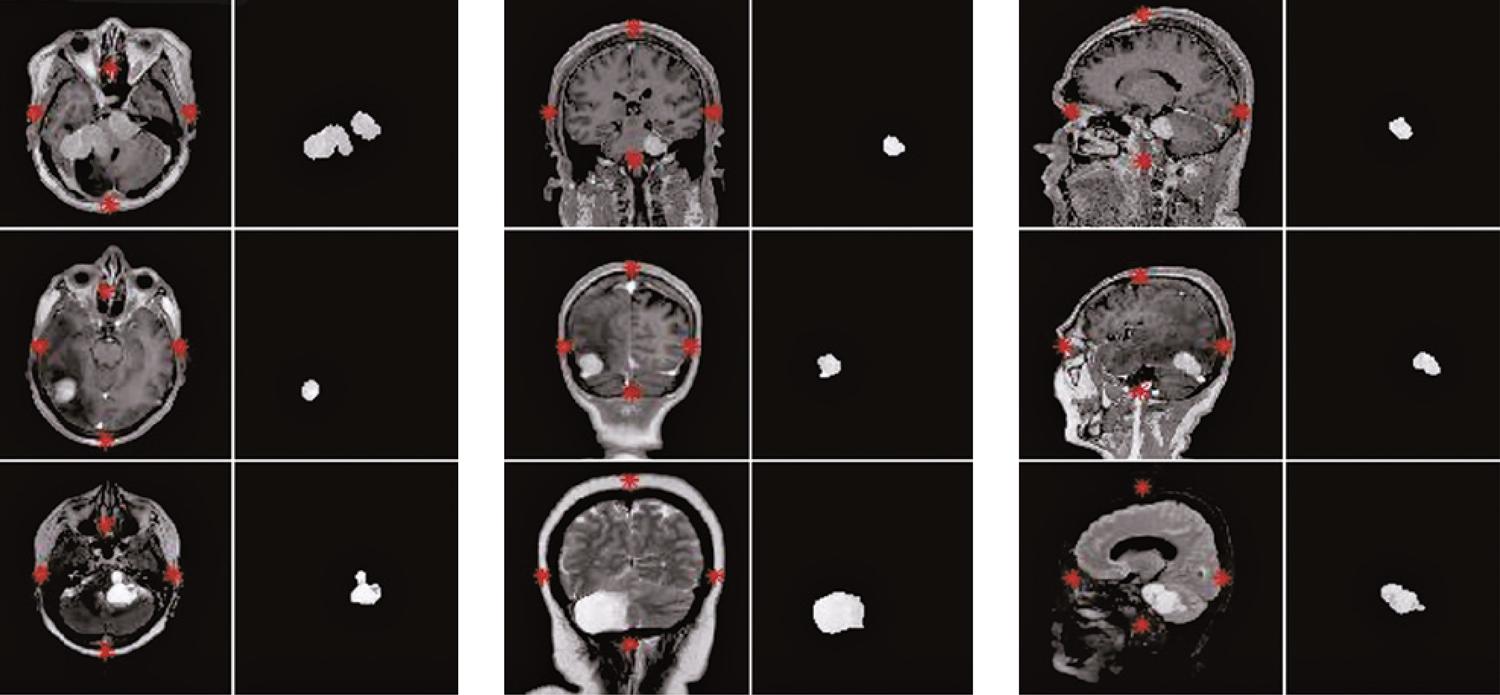
A figura 4 apresenta alguns resultados finais de total sucesso da segmentação implementada, que abrangeu exames de RM de pacientes distintos, nas ponderações T1c, T2 e FSPGR T1c nos planos axial, coronal e sagital. A segmentação de imagens na ponderação T1 com contraste para três pacientes diferentes pode ser observada na primeira linha. A segunda linha mostra os respectivos resultados das segmentações para a ponderação T2. Na terceira linha, apresentam-se os resultados de segmentação da ponderação T1 com contraste na sequência gradiente eco com uso de spoiller.
Resultado final da segmentação automática em imagens de ressonância magnética na ponderação T1, T2 e T1 com contraste na sequência gradiente eco com uso de spoiller
Análise de desempenho comparativo da segmentação automática
As RM utilizadas no teste foram segmentadas manualmente por radiologistas. Tais resultados foram considerados padrão-ouro e utilizados para avaliar o sistema de modo eficiente. Na tabela 2 , encontram-se os percentuais de VP, FP, FN e de PA em relação ao padrão-ouro, como também os respectivos valores de TC. Em trabalhos similares, autores obtiveram PA de 86% e TC de 0,79, o que foi considerado resultado satisfatório.( 2424. Deng W, Luo L, Lin X, Fang T, Liu D, Dan G, et al. Head and neck cancer tumor segmentation using support vector machine in dynamic contrast-enhanced MRI. Contrast Media Mol Imaging. 2017;2017:8612519.
25. Vaibhavi P, Rupal K. Brain Tumor Segmentation Using K-means-FCM Hybrid Technique. In: Perez GM, Tiwari S, Trivedi MC, Mishra KK. Ambient Communications and Computer Systems. New York: Springer; 2018. p 341-52. - 2626. Ma Z, Wu X, Song Q, Luo Y, Wang Y, Zhou J. Automated nasopharyngeal carcinoma segmentation in magnetic resonance images by combination of convolutional neural networks and graph cut. Experimental Therapeutic Medicine. 2016;16(3):2511-21. )Neste estudo, alcançou-se PA de 89,23% e TC de 0,70, ou seja, um resultado quantitativamente semelhante ao apresentado pelos autores, o que indica boa acurácia na segmentação proposta. Os melhores resultados de PA foram obtidos para as sequências T2 e FSPGR-T1c.
Foram obtidos resultados também satisfatórios para a TC, embora tenha sido gerada quantidade considerável de FP. Entretanto, a ocorrência desses FP não superou a de VP, como ocorreu no estudo de Alegro et al.( 2323. Alegro Mde C, Amaro Junior E, Lopes Rde D. Computerized brain tumor segmentation in magnetic resonance imaging. einstein (São Paulo). 2012; 10(2):158-63. )Uma abordagem inovadora e robusta de segmentação de imagens para a extração de tumores cerebrais em IRM foi desenvolvida aplicando-se a função Kernel à máquina de vetores de suporte (K-SVM), obtendo-se PA e TC equivalentes a 87,8% e 0,85, respectivamente.( 2727. Singh R, Agarwal P, Bhattacharya M. MR brain tumor detection employing Laplacian Eigen maps and kernel support vector machine. In: 2016 IEEE International Conference On Bioinformatics And Biomedicine (BIBM). IEEE. 2016;827-30. )Para melhor comparar o desempenho do algoritmo de segmentação proposto com os encontrados na literatura, organizaram-se os resultados quantitativos de estudos semelhantes na tabela 3 .
DISCUSSÃO
As técnicas de pré-processamento conseguiram eliminar grande parte do cérebro e algumas regiões do encéfalo, no entanto, ainda restaram elementos espúrios, como o osso da cabeça, os ventrículos laterais e o corpo caloso, que, em algumas situações, apresentam pixels com intensidades próximas ou superiores às do tecido tumoral, sendo difíceis de serem eliminados. Ainda, foi testada utilizando o filtro mediano, que é uma ferramenta muito utilizada para filtragem do ruído em RM cerebrais.( 1717. Isselmou AE, Zhang S, Xu G. A novel approach for brain tumor detection using MRI Images. J Biomedical Sci Eng. 2016;9(10):44-52. , 3030. Anithadevi D, Perumal K. Brain tumour extraction based on segmentation. IJRITCC. 2014;2(9):2682-9. )No entanto, não foram obtidos resultados superiores ao método desenvolvido, tendo como desvantagem o aumento do tempo de processamento do algoritmo. De fato, a operação de convolução com máscaras exige grande esforço computacional.( 3131. Marques Filho O, Vieira Neto H. Processamento digital de imagens. Rio de Janeiro: Brasport; 1999. )
A ponderação T1 apresenta imagens com a tonalidade do tecido tumoral muito próxima à do cérebro. Este é um problema que dificulta bastante a etapa de pré-processamento, visto que os critérios e limiares utilizados para intensificar a região tumoral acabam por ser também aplicados às demais regiões do cérebro que não são de interesse. Isto pode gerar tecidos saudáveis conectados ao tumor, mesmo após o uso de operações morfológicas, além de aumentar o número de falso-positivos após a segmentação. Os resultados do pré-processamento foram melhores nas ponderações T2 e FSPRG T1c, em consequência dessas imagens apresentarem maior contraste entre a região hiper-intensa e o restante do cérebro.
Ao analisar as figuras 3 e 4, percebe-se que o pré-processamento e a segmentação baseados nas técnicas descritas anteriormente obtiveram bons resultados com a implementação de um algoritmo simples, que utiliza funções e operações lógicas básicas. A tabela 4 apresenta a metodologia realizada por alguns autores para aprimorar a qualidade de imagens de tumores cerebrais por RM e extrair o crânio, os quais obtiveram resultados qualitativamente comparáveis aos deste estudo.
O algoritmo MDC é capaz de marcar os extremos do cérebro em qualquer tipo de corte (axial, coronal ou sagital) do encéfalo. Isso torna o sistema mais preciso, visto que a forma geométrica do cérebro varia nos três tipos de corte. Ainda, ele funciona também quando ocorre assimetria na localização da cabeça dentro da imagem, seja horizontal ou verticalmente, o que é de comum ocorrência.( 3030. Anithadevi D, Perumal K. Brain tumour extraction based on segmentation. IJRITCC. 2014;2(9):2682-9. , 4141. Arunkumar N, Mohammed MA, Mostafa SA, Ibrahim DA, Rodrigues JJ, Albuquerque VH. Fully automatic model-based segmentation and classification approach for MRI brain tumor using artificial neural networks. Concurrency Computat Pract Exper. 2018;32:e4962. )Ademais, abrange diversas possibilidades para extração das regiões externas ao cérebro, aplicando-se a imagens com hipersinal em distintas ponderações, visto que independe de características qualitativas, como entropia e textura. Ao contrário, ele é baseado na localização em que o encéfalo se encontra dentro da imagem, em sua anatomia e intensidade dos pixels circundantes, semelhante ao que foi proposto no método S3.( 3333. Roy S, Maji P. A simple skull stripping algorithm for brain MRI. In: Eighth International Conference On Advances In Pattern Recognition (ICAPR) [Internet]. Kolkata: (IN); 2015 [cited 2019 Aug 20]. Available from: https://ieeexplore.ieee.org/abstract/document/7050671/
https://ieeexplore.ieee.org/abstract/doc...
)
O MDC apresentou resultados satisfatórios quanto à sua funcionalidade e à sua abrangência, podendo ser comparado, qualitativamente, com os algoritmos apresentados na tabela 4 , que têm bom desempenho na extração do crânio. Nos casos em que a área externa ao cérebro não é completamente eliminada, a operação morfológica de dilatação é capaz de subdividir tais “sobras” em partes menores, que podem ser eliminadas com a aplicação do MDA. Assim, o MDA executa a tarefa de complemento à segmentação por detecção de coordenadas. Ele está diretamente ligado à quantidade de FP existentes na pós-segmentação, visto que quanto mais tecidos não tumorais forem eliminados, menor a quantidade de FP e melhor o desempenho da segmentação.
No entanto, o MDA é falho em situações que existem regiões pós-processadas com área maior que o tumor, pois há o risco de a área do tumor ser inferior à área média das subregiões existentes e causar sua eliminação, reduzindo a quantidade de VP; ou produzir imagens com tumor e não tumor, aumentando o número de FP. Isso pode acontecer em fatias em que os tecidos normais possuem intensidades de pixel semelhantes às intensidades dos pixels do tumor, e nas quais a diferenciação dessas regiões é dificultada com as técnicas de pré-processamento adotadas. Além dessas limitações, o MDC e o MDA foram desenvolvidos para segmentar apenas RM com tumor em hipersinal.
A sequência T1c apresentou valores de PA e TC mais baixos comparados às demais ponderações, por apresentaram imagens em que a região cerebral é composta por pixels com intensidades muito semelhantes ao do tumor. Com isso, foi gerado um número elevado de FP conectados, que, em alguns casos, possuíam área muito superior à do tumor; desta forma, o tumor foi eliminado, e a imagem segmentada não apresentou tecido tumoral, gerando os FN.
Os casos falhos utilizando a ponderação T2 devem-se à presença de ventrículos em algumas fatias de corte coronal e dos olhos e nariz, em fatia axial, que apresenta pixels com intensidade muito alta, quase próxima a 1, o que acarretou elevado número de FP, além do tumor não ter sido detectado em três fatias FN. Este último problema também acarretou falhas em fatias no plano axial da ponderação FSPGR T1c, explicando seu elevado número de FP.
Embora tenha sido gerada uma quantidade considerável de FP, foram obtidos resultados satisfatórios para PA e TC, alcançando-se média de 89,23% de acertos entre as áreas segmentadas automaticamente e o padrão-ouro, o que indica boa acurácia na segmentação proposta.
CONCLUSÃO
Os resultados obtidos mostraram que o sistema proposto foi capaz de localizar e delimitar a região tumoral sem qualquer interação com o usuário, utilizando método inovador de segmentação automática, simples e fácil implementação.
AGRADECIMENTOS
O presente trabalho foi realizado com apoio da Fundação de Amparo à Ciência e Tecnologia do Estado de Pernambuco (FACEPE) - Brasil - Código de Financiamento IBPG-0924-16.
REFERENCES
-
1Jui SL, Zhang S, Xiong W, Yu F, Fu M, Wang D, et al. Brain MRI Tumor Segmentation with 3D Intracranial Structure Deformation Features. IEEE Intelligent Systems. 2016;31(2):66-76.
-
2Virupakshappa, Amarapur B. Cognition-based MRI brain tumor segmentation technique using modified level set method. Cognition, Technol Work. 2019; 21(3):357-69.
-
3Szabó Z, Kapás Z, Lefkovits L, Gyõrfi Á, Szilágyi SM, Szilágyi L. Automatic segmentation of low-grade brain tumor using a random forest classifier and Gabor features.In:14th International Conference on Natural Computation, Fuzzy Systems and Knowledge Discovery (ICNC-FSKD) [Internet]. Huangshan (CHN): ICNC-FSKD 2018 [cited 2019 Feb 6]. Available from: http://real.mtak.hu/86054/1/2023.pdf
» http://real.mtak.hu/86054/1/2023.pdf -
4Zhou M, Scott J, Chaudhury B, Hall L, Goldgof D, Yeom KW, et al. Radiomics in Brain Tumor: image assessment, quantitative feature descriptors, and machine-learning approaches. AJNR Am J Neuroradiol. 2018;39(2):208-16. Review.
-
5Goetz M, Weber C, Bloecher J, Stieltjes B, Meinzer HP, Maier-Hein KH. Extremely randomized trees based brain tumor segmentation [Internet]. Proceeding of MICCAI 2014 Brain Tumor Segmentation Challenge. 2014;6-11 [cited 2019 Feb 19]. Available from: http://people.csail.mit.edu/menze/papers/proceedings_miccai_brats_2014.pdf
» http://people.csail.mit.edu/menze/papers/proceedings_miccai_brats_2014.pdf -
6Schmidt M. Automatic brain tumor segmentation [Tese]. Edmonton: University of Alberta; 2005. 161f.
-
7Clarke LP, Velthuizen RP, Clark M, Gaviria J, Hall L, Goldgof D, et al. MRI measurement of brain tumor response: comparison of visual metric and automatic segmentation. Magn Reson Imaging. 1998;16(3):271-9.
-
8Işın A, Direkoğlu C, Şah M. Review of MRI-based brain tumor image segmentation using deep learning methods. Procedia Computer Science. 2016;102:317-24.
-
9Jorritsma W, Cnossen F, van Ooijen PM. Improving the radiologist–CAD interaction: designing for appropriate trust. Clin Radiol. 2015;70(2):115-22. Review.
-
10Zhang J, Cui X, Li J, Wang R. Imbalanced classification of mental workload using a cost-sensitive majority weighted minority oversampling strategy. Cognition, Technol Work. 2017;19(4):633-53.
-
11Doupi P, Svaar H, Bjørn B, Deilkås E, Nylén U, Rutberg H. Use of the global trigger tool in patient safety improvement efforts: nordic experiences. Cognition, Technol Work. 2015;17(1):45-54.
-
12Mohan G, Subashini MM. MRI based medical image analysis: survey on brain tumor grade classification. Biomedical Signal Processing Control. 2018;39:139-61.
-
13Clarke LP, Velthuizen RP, Camacho MA, Heine JJ, Vaidyanathan M, Hall LO, et al. MRI segmentation: methods and applications. Magn Reson Imaging. 1995;13(3):343-68. Review.
-
14Gonzalez RC, Woods RE. Processamento de imagens digitais, tradução do original digital image processing. São Paulo: Edgard Blücher; 2000. p. 509.
-
15Isa IS, Sulaiman SN, Mustapha M, Karim NK. Automatic contrast enhancement of brain MR images using Average Intensity Replacement based on Adaptive Histogram Equalization (AIR-AHE). Biocybernetics Biomedical Engineering. 2017;37(1):24-34.
-
16Alegro MC. Segmentação computadorizada de tumores do encéfalo em imagens de ressonância magnética [Dissertação]. São Paulo: Universidade de São Paulo; 2009.
-
17Isselmou AE, Zhang S, Xu G. A novel approach for brain tumor detection using MRI Images. J Biomedical Sci Eng. 2016;9(10):44-52.
-
18Ulku EE, Camurcu AY. Computer aided brain tumor detection with histogram equalization and morphological image processing techniques. In: International Conference on Electronics, Computer and Computation (ICECCO) [Internet]. Ancara:(TR); 2013 [cited 2019 Feb 6]. Available from: https://ieeexplore.ieee.org/document/6718225?tp=&arnumber=6718225
» https://ieeexplore.ieee.org/document/6718225?tp=&arnumber=6718225 -
19Tharani SS, Sumathi B. A Novel Algorithm for Enhancing an Image of Brain Tumor. Int J Scientific Res Comput Sci Eng Inf Technol. 2018;3(3):186-90.
-
20Thompson CM, Shure L. Image processing toolbox [for use with Matlab] (No. BOOK). The Math Works. 1995.
-
21Kawaguchi K, Kageyama R, Sano M. Topological defects control collective dynamics in neural progenitor cell cultures. Nature. 2017;545 (7654):327-31.
-
22Malhotra H, Naaz S. Analysis of MRI images using data mining for detection of brain tumor. Int J Advanced Res Comput Sci. 2018;9(2):15-8.
-
23Alegro Mde C, Amaro Junior E, Lopes Rde D. Computerized brain tumor segmentation in magnetic resonance imaging. einstein (São Paulo). 2012; 10(2):158-63.
-
24Deng W, Luo L, Lin X, Fang T, Liu D, Dan G, et al. Head and neck cancer tumor segmentation using support vector machine in dynamic contrast-enhanced MRI. Contrast Media Mol Imaging. 2017;2017:8612519.
-
25Vaibhavi P, Rupal K. Brain Tumor Segmentation Using K-means-FCM Hybrid Technique. In: Perez GM, Tiwari S, Trivedi MC, Mishra KK. Ambient Communications and Computer Systems. New York: Springer; 2018. p 341-52.
-
26Ma Z, Wu X, Song Q, Luo Y, Wang Y, Zhou J. Automated nasopharyngeal carcinoma segmentation in magnetic resonance images by combination of convolutional neural networks and graph cut. Experimental Therapeutic Medicine. 2016;16(3):2511-21.
-
27Singh R, Agarwal P, Bhattacharya M. MR brain tumor detection employing Laplacian Eigen maps and kernel support vector machine. In: 2016 IEEE International Conference On Bioinformatics And Biomedicine (BIBM). IEEE. 2016;827-30.
-
28Hsieh TM, Liu YM, Liao CC, Xiao F, Chiang IJ, Wong JM. Automatic segmentation of meningioma from non-contrasted brain MRI integrating fuzzy clustering and region growing. BMC Medical Informatics Decision Making. 2011;11:54.
-
29Gao F, Lin T. Application of Computer-Aided Diagnosis Technology in Brain Tumour Detection. NeuroQuantology. 2018;16(5):725-33.
-
30Anithadevi D, Perumal K. Brain tumour extraction based on segmentation. IJRITCC. 2014;2(9):2682-9.
-
31Marques Filho O, Vieira Neto H. Processamento digital de imagens. Rio de Janeiro: Brasport; 1999.
-
32Sujan M, Alam N, Noman SA, Islam MJ. A Segmentation based Automated System for Brain Tumor Detection. IJCA. 2016;153(10):41-9.
-
33Roy S, Maji P. A simple skull stripping algorithm for brain MRI. In: Eighth International Conference On Advances In Pattern Recognition (ICAPR) [Internet]. Kolkata: (IN); 2015 [cited 2019 Aug 20]. Available from: https://ieeexplore.ieee.org/abstract/document/7050671/
» https://ieeexplore.ieee.org/abstract/document/7050671/ -
34khandelwal P, Kaur G. Comparative study of different image enhancement technique. IJECT. 2016;7(2):116-21.
-
35Kaur R, Chawla M, Khiva NK, Ansari MD. Comparative Analysis of Contrast Enhancement Techniques for Medical Images. Pertanika J Sci Technol. 2018;26(3):965-78.
-
36Shattuck DW, Sandor-Leahy SR, Schaper KA, Rottenberg DA, Leahy RM. Magnetic resonance image tissue classification using a partial volume model. Neuroimage. 2001;13(5):856-76.
-
37Smith SM. Fast robust automated brain extraction. Hum Brain Mapp. 2002;17(3):143-55. Review.
-
38Roura E, Oliver A, Cabezas M, Vilanova JC, Rovira A, Ramió-Torrentà L, et al. MARGA: multispectral adaptive region growing algorithm for brain extraction on axial MRI. Comput Methods Programs Biomed. 2014;113(2):655-73.
-
39Somasundaram K, Mercina JH, Magesh Kalaiselvi ST. Brain Portion Extraction Scheme using Region Growing and Morphological Operation from MRI of Human Head Scans. IJCSE. 2018;6(4):298-302.
-
40Kalavathi P, Prasath VB. Methods on Skull Stripping of MRI Head Scan Images–a Review. J Digit Imaging. 2016;29(3):365-79. Review.
-
41Arunkumar N, Mohammed MA, Mostafa SA, Ibrahim DA, Rodrigues JJ, Albuquerque VH. Fully automatic model-based segmentation and classification approach for MRI brain tumor using artificial neural networks. Concurrency Computat Pract Exper. 2018;32:e4962.
Datas de Publicação
-
Publicação nesta coleção
09 Mar 2020 -
Data do Fascículo
2020
Histórico
-
Recebido
16 Jan 2019 -
Aceito
2 Set 2019